A epidermólise bolhosa é caracterizada por fragilidade cutânea e formação de bolhas na pele. Historicamente, o diagnóstico é realizado por mapeamento de imunofluorescência ou microscopia eletrônica de transmissão. A análise genética, especialmente por sequenciamento de nova geração, é ferramenta útil para o diagnóstico da doença. No Brasil, o acesso aos métodos de diagnóstico é limitado e, consequentemente, muitos pacientes não recebem um diagnóstico preciso. O diagnóstico possibilita a indicação do prognóstico e o aconselhamento genético do paciente.
ObjetivosAvaliar a relação custo‐efetividade de um painel de genes em comparação ao mapeamento de imunofluorescência e à microscopia eletrônica de transmissão, analisando benefícios, limitações e aspectos econômicos envolvidos.
MétodosO painel incluiu os 11 principais genes associados à epidermólise bolhosa. Foi realizada comparação entre as técnicas, com avaliação do custo, vantagens e limitações, por meio de pesquisa de preços e revisão da literatura.
ResultadosOs métodos de mapeamento de imunofluorescência e microscopia eletrônica de transmissão exigem biópsia de pele, dependem da expertise do analista e estão sujeitos a resultados inconclusivos. O painel de genes se mostra efetivo para o diagnóstico da epidermólise bolhosa, com alta eficiência e acurácia, é economicamente viável e dispensa a necessidade de biópsia. O painel possibilita o prognóstico, diagnóstico genético pré‐natal e aconselhamento genético.
Limitações do estudoNão foram identificados laboratórios que realizem microscopia eletrônica de transmissão para epidermólise bolhosa no Brasil.
Conclusão: Nossos resultados apoiam o painel de genes como primeira escolha para o diagnóstico da epidermólise bolhosa.
A epidermólise bolhosa (EB) compreende um grupo heterogêneo de doenças dermatológicas genéticas, caracterizada pela fragilidade cutânea, com a formação de bolhas, erosões e cicatrizes em resposta ao mínimo trauma mecânico.1 A EB é classificada em quatro tipos principais, de acordo com a camada da pele na qual ocorre a formação de bolhas: EB simples (EBS; intraepidérmica), EB juncional (EBJ; dentro da lâmina lúcida da membrana basal), EB distrófica (EBD; abaixo da membrana basal) e EB kindler (padrão misto de clivagem da pele).1,2 A classificação nos subtipos de EB leva em consideração as características clínicas, a distribuição de bolhas (localizada ou generalizada) e a gravidade dos sinais cutâneos e extracutâneos.2 Variantes patogênicas em pelo menos 16 genes que codificam proteínas com funções estruturais são responsáveis pela heterogeneidade genética e alélica da EB.2,3
A incidência e prevalência da EB foram estimadas nos Estados Unidos (EUA) em 19,57 e 11,07 por um milhão de indivíduos, respectivamente.4 No Brasil, não há estudos epidemiológicos sobre a doença. A DEBRA‐Brasil, em um esforço para cadastramento dos pacientes brasileiros com EB, apresenta registro de mais de 900 casos no país (dados não publicados). Estima‐se que o número de pacientes brasileiros seja ainda mais elevado, levando‐se em conta o alto índice de subdiagnóstico (especialmente em casos de menor gravidade), os erros de diagnóstico e a morte prematura em casos graves, além da dificuldade de acesso aos principais centros de saúde.
O diagnóstico da EB em centros especializados é feito principalmente por exame clínico seguido da técnica de imunofluorescência direta no tecido (conhecida como mapeamento por imunofluorescência ou imunomapeamento) e/ou microscopia eletrônica de transmissão, realizados a partir de amostras de biópsia de pele.5 Quando acessível, o teste genético também é uma importante ferramenta para o diagnóstico da EB. A classificação do tipo de EB contribui para a avaliação prognóstica do paciente e é importante para o aconselhamento genético familiar.
O mapeamento por imunofluorescência (MIF) se baseia na utilização de anticorpos específicos que possibilitam a identificação da proteína não funcional e a camada da pele em que ocorre a clivagem nos tipos mais graves de EB.5–7 Entretanto, a informação é dependente da qualidade e do número de anticorpos utilizados, e resultados inconclusivos podem ocorrer por diferentes fatores. Em primeiro lugar, é possível a formação de clivagens na amostra de pele durante o armazenamento e processamento, o que leva a resultados imprecisos.3,7 Além disso, a camada de clivagem pode não estar presente e, consequentemente, a classificação do tipo de EB não ser possível nesses casos.7 Por fim, a interpretação dos resultados depende do conhecimento e expertise do investigador, o que exige equipe especializada para o correto diagnóstico.3,5
A microscopia eletrônica de transmissão (MET) também torna possível o diagnóstico da EB pela determinação da camada da pele em que ocorre a clivagem. Como diferencial, a técnica ainda possibilita a visualização e avaliação semiquantitativa de estruturas que estão alteradas em subtipos específicos de EB (filamentos de queratina, desmossomos, hemidesmossomos, filamentos e fibrilas de ancoragem).5 Entretanto, trata‐se de um método de alto custo e de difícil processamento e análise, o que exige, portanto, laboratórios altamente qualificados com apropriada expertise.5 Além disso, os resultados da MET podem não ser informativos, por ausência de clivagem na pele, alterações não específicas ou modificações muito sutis nas estruturas de adesão. Artefatos também podem ocorrer em decorrência do processamento e da técnica de biópsia.3 Em função da escassez de laboratórios aptos a realizar a técnica, a MET é raramente a primeira escolha para o diagnóstico de EB.
Até pouco tempo, o diagnóstico genético de pacientes com EB só era possível por meio de sequenciamento de Sanger de genes candidatos, definidos após a realização de MIF e/ou MET, uma estratégia laboriosa e de alto custo.1 Recentemente, a tecnologia de sequenciamento de nova geração (NGS, do inglês, next‐generation sequencing) emergiu como uma ferramenta útil para identificação de variantes genéticas, e estudos em EB têm demonstrado sua alta sensibilidade diagnóstica.8–12
No Brasil, o acesso aos métodos de diagnóstico da EB está disponível apenas para uma pequena parcela dos pacientes. A primeira razão para isso se dá pelas dificuldades técnicas envolvidas, que exigem especialistas para a análise. Embora essa dificuldade seja comum a todos que realizam as técnicas, é ainda mais marcante em um país como o Brasil, que carece de analistas experientes. A segunda razão está relacionada com a insuficiência de centros de saúde especializados no Brasil e com a indisponibilidade dos exames pelo sistema público de saúde. Consequentemente, a maioria dos pacientes brasileiros com a doença não tem um diagnóstico preciso e conclusivo do tipo de EB associado, seja por dificuldades técnicas, que levam a resultados inconclusivos, seja por falta de acesso a esses métodos no país.
Tendo em vista a dificuldade de diagnóstico da doença no Brasil, propomos um painel de genes específico para EB por meio da tecnologia de NGS. Em estudo anterior, apresentamos as variantes novas e recorrentes identificadas em pacientes brasileiros por meio do painel de genes.13 O objetivo do presente estudo é avaliar a relação custo‐efetividade do painel de genes em comparação com os exames historicamente empregados, discutindo as principais vantagens e limitações de cada método, bem como os aspectos econômicos envolvidos.
MétodosAspectos éticosO presente estudo foi aprovado pelo Comitê de Ética em Pesquisa da Universidade Federal do Rio Grande do Sul (projeto 31608) e pela Plataforma Brasil (CAAE 2.481.885).
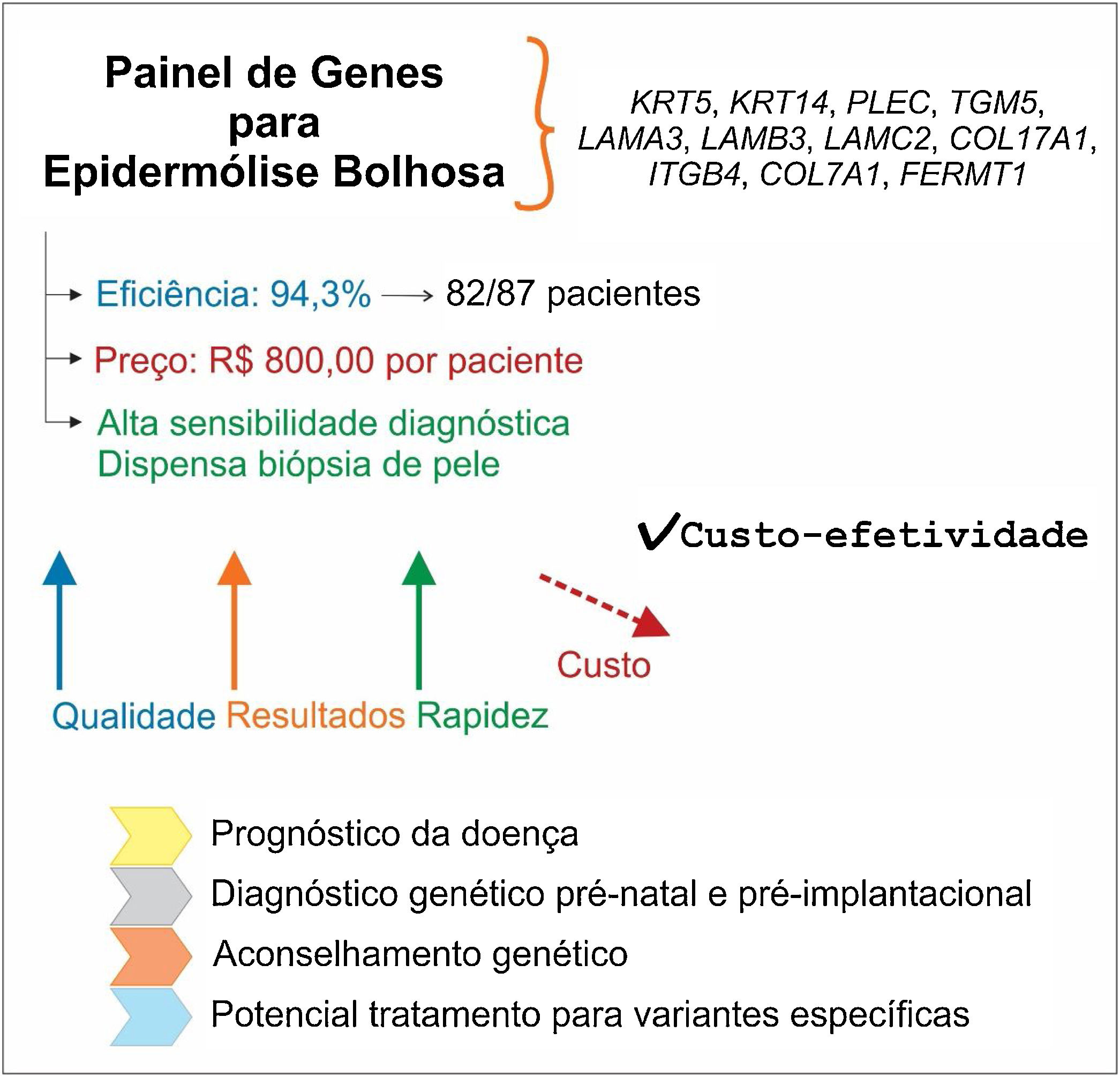
Painel da genes de EBO painel de genes de NGS foi desenhado com a finalidade de ser específico para EB, resolver a extensa maioria dos casos da doença e apresentar um preço viável para a realidade brasileira. Assim, os 11 principais genes associados aos quatro tipos de EB foram incluídos no painel (KRT5, KRT14, PLEC, TGM5, LAMA3, LAMB3, LAMC2, COL17A1, ITGB4, COL7A1 e FERMT1). O painel de 62,72kb e 574 amplicons foi desenhado a partir da ferramenta AmpliSeq Designer (Thermo Fisher Scientific, EUA). A tecnologia ion torrent (Thermo Fisher Scientific, EUA) foi empregada para o sequenciamento. Informações sobre os parâmetros das corridas, análise de dados, investigação, classificação e detalhamento das variantes identificadas foram descritas em estudo prévio.13
Avaliação dos métodos de diagnósticoA fim de se obter uma estimativa do valor a ser investido para a execução das técnicas de MIF, MET e sequenciamento por painel de genes, foi realizado levantamento de preços em hospitais/laboratórios brasileiros que executam cada um desses métodos. Para a técnica de MET foi realizado levantamento em laboratórios dos EUA, uma vez que o Brasil não tem laboratórios que realizem o método para diagnóstico de EB. Como o custo do sequenciamento pelo painel de genes depende do número de amostras a serem analisadas, para fins de cálculo, a estimativa de preço levou em consideração um número de 20 amostras. As características de cada método, bem como suas vantagens e desvantagens inerentes, foram revisadas em literatura especializada.
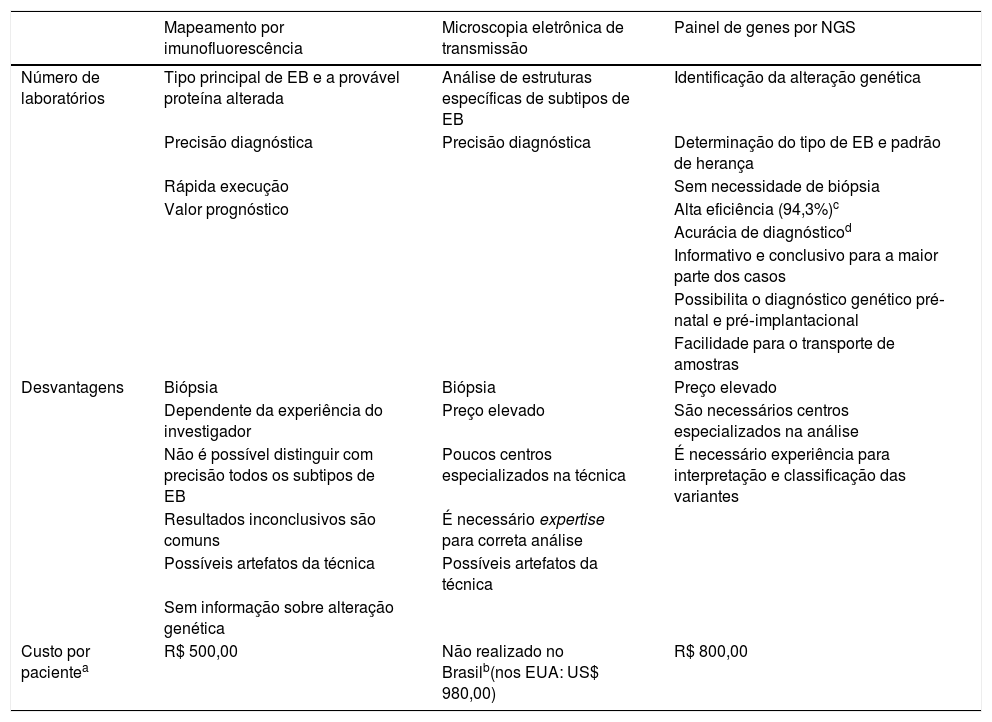
ResultadosOs pontos positivos e negativos das técnicas utilizadas para o diagnóstico da EB estão compilados na tabela 1. Como demonstrado em estudo prévio,13 a eficiência do painel de genes desenvolvido foi de 94,3% (em 82 dos 87 casos‐índice estudados, as variantes patogênicas foram identificadas). Ainda que a MIF e a MET tenham relativa precisão de diagnóstico, apenas o painel de genes torna possível uma análise completa e definitiva, com a identificação da alteração genética causal. Além disso, o painel de genes apresenta como significativo benefício a dispensabilidade de realização de biópsia (particularmente nociva a esses pacientes com uma pele sensível), imprescindível nos outros dois métodos.
Comparação entre as técnicas utilizadas para o diagnóstico de epidermólise bolhosa
| Mapeamento por imunofluorescência | Microscopia eletrônica de transmissão | Painel de genes por NGS | |
|---|---|---|---|
| Número de laboratórios | Tipo principal de EB e a provável proteína alterada | Análise de estruturas específicas de subtipos de EB | Identificação da alteração genética |
| Precisão diagnóstica | Precisão diagnóstica | Determinação do tipo de EB e padrão de herança | |
| Rápida execução | Sem necessidade de biópsia | ||
| Valor prognóstico | Alta eficiência (94,3%)c | ||
| Acurácia de diagnósticod | |||
| Informativo e conclusivo para a maior parte dos casos | |||
| Possibilita o diagnóstico genético pré‐natal e pré‐implantacional | |||
| Facilidade para o transporte de amostras | |||
| Desvantagens | Biópsia | Biópsia | Preço elevado |
| Dependente da experiência do investigador | Preço elevado | São necessários centros especializados na análise | |
| Não é possível distinguir com precisão todos os subtipos de EB | Poucos centros especializados na técnica | É necessário experiência para interpretação e classificação das variantes | |
| Resultados inconclusivos são comuns | É necessário expertise para correta análise | ||
| Possíveis artefatos da técnica | Possíveis artefatos da técnica | ||
| Sem informação sobre alteração genética | |||
| Custo por pacientea | R$ 500,00 | Não realizado no Brasilb(nos EUA: US$ 980,00) | R$ 800,00 |
É importante notar a diferença entre “eficiência” e “acurácia”. Eficiência se refere à proporção de casos solucionados (isto é, casos com a mutação patogênica identificada) em relação ao total de pacientes analisados. O termo “acurácia”, por sua vez, se refere à precisão (exatidão, correção) dos resultados.
O valor a ser investido no diagnóstico da EB pelo painel de genes foi estimado em R$ 800,00 por amostra, levando‐se em consideração um total de 20 amostras analisadas. A média dos preços das análises de MIF praticados no mercado brasileiro é de R$ 500,00. Após extensa busca em diferentes centros de referência no Brasil, não foi encontrado laboratório que realize a técnica de MET para diagnóstico de EB no país. Para fins de estimativa, foi realizado levantamento do preço da técnica em laboratórios dos EUA, que indicou um preço médio de US$ 980,00 (R$ 1,00=US$ 0,25).
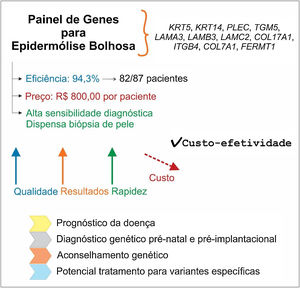
A figura 1 apresenta um resumo das principais características, vantagens e implicações da utilização do painel de genes para o diagnóstico da EB.
O painel de genes como primeira escolha para o diagnóstico da EB: principais características. O painel de genes apresenta alta eficiência e precisão, preço comparável às demais metodologias de diagnóstico de EB, além de dispensar a necessidade de biópsia de pele. O custo da tecnologia de NGS vem diminuindo, o que indica que o painel de genes deve tornar‐se mais acessível nos próximos anos. A identificação da alteração genética possibilita o prognóstico da doença, o diagnóstico genético pré‐natal e pré‐implantacional, tem implicações para o aconselhamento genético e é importante para o desenvolvimento de futuras terapias para EB.
No presente estudo, apresentamos um levantamento das vantagens e desvantagens dos métodos de diagnóstico da EB e uma avaliação acerca da potencialidade do painel de genes como primeira escolha para diagnóstico da doença no Brasil.
Painel de genes, MIF e MET: efetividade, benefícios imediatos e futuro potencialAs técnicas de MIF e MET são, historicamente, as mais empregadas para o diagnóstico da EB e possibilitam a determinação do nível de clivagem na pele e a consequente indicação do tipo de EB associada.5 Ainda que apresentem precisão diagnóstica para o tipo principal de EB, esses métodos não indicam o modo de herança genética e dependem de experiência para análise, estando sujeitos a frequentes resultados imprecisos ou inconclusivos (tabela 1).3,14 A MET, em especial, é uma técnica bastante laboriosa e que necessita de notável conhecimento técnico – é, por isso, muito pouco empregada em todo o mundo. A identificação da variante patogênica associada é o critério mais definitivo e preciso para o diagnóstico de EB. O sequenciamento dos genes candidatos pelo método de Sanger é, portanto, realizado em centros especializados, posteriormente ao MIF e/ou à MET.5,14 Ainda que informativa na maioria das vezes, a combinação dos métodos de MIF, MET e sequenciamento de Sanger falha em aproximadamente 15% dos casos, seja por incerteza no exame clínico ou por limitações intrínsecas das técnicas.8 Ademais, essa abordagem combinada pode apresentar alto custo e exigir grande demanda de trabalho, visto que diversos dos genes associados à EB contêm múltiplos éxons, como o COL7A1 (118 éxons) e o COL17A1 (56 éxons).8
Diferentemente do sequenciamento de Sanger, o painel de genes por NGS torna possível o sequenciamento simultâneo de um grande número de genes com conhecida associação à doença,1 dispensando a determinação prévia de genes candidatos. A análise genética pelo painel de genes é rápida, de alta precisão e conclusiva para o diagnóstico da EB, sem demandar a realização de exames prévios como MIF e MET (tabela 1).
Os métodos de MIF e MET requerem biópsia de pele, geralmente realizada a partir de bolha íntegra recente. Esse exame por si só já é altamente invasivo, mas em pacientes com EB, que apresentam uma pele tão frágil, torna‐se ainda mais agressivo. Ainda, não é incomum a necessidade de repetição de biópsia em alguns pacientes, em decorrência de resultados inconclusivos. Uma vez que as análises por NGS dispensam a necessidade de biópsia, podem ser realizadas com amostra de saliva e fornecem diagnóstico com rapidez e alta precisão, autores têm defendido que os métodos atuais para avaliação dos pacientes com EB devem ser repensados.15 A identificação das variantes causais associadas à EB possibilita a indicação do prognóstico da doença, o diagnóstico genético pré‐natal e pré‐implantacional e o aconselhamento genético preciso.3
Embora o sequenciamento por NGS seja uma ferramenta poderosa para o diagnóstico genético, algumas limitações devem ser consideradas. O principal desafio se refere às análises de bioinformática, essenciais para a identificação das variantes genéticas e, ainda mais importante, para indicar sua causalidade.8 Nesse sentido, a interpretação de variantes de significado incerto pode ser bastante difícil, causando dilemas para os clínicos e incertezas de como informar os pacientes e fornecer o aconselhamento genético apropriado. Além disso, grandes rearranjos e sequências repetitivas são complexos de se analisar por NGS e, por isso, frequentemente não são detectados de maneira correta. Portanto, é importante notar que, embora altamente efetiva, a tecnologia de NGS não é capaz de solucionar todos os casos de doenças genéticas.
O painel de genes desenvolvido por nosso grupo apresentou alta eficiência (94,3%) na amostra estudada, equiparada aos resultados obtidos por outros pesquisadores que utilizaram painéis com um número maior de genes.10–12 A escolha pelos 11 principais genes associados à EB se deu a fim de se estabelecer uma metodologia economicamente viável, ainda que conhecidamente existam outros genes também reportados na literatura.2,3 Embora o painel desenvolvido falhe em diagnosticar raros casos associados a outros genes (como DST, EXPH5, KLHL24, CD151, ITGA3 e ITGA6), a eficiência aqui obtida demonstra que mesmo um painel de genes menor pode ser de notável relevância para o diagnóstico da EB. Cabe destacar que a metodologia de NGS não torna possível a identificação adequada de grandes rearranjos genéticos, o que pressupõe que mesmo um painel incluindo todos os genes já associados à EB não garante 100% de eficiência.
Afora os benefícios imediatos, a análise genética pode ser uma ferramenta essencial para o desenvolvimento e a aplicação de terapias para EB. Embora atualmente não exista tratamento, recentes estudos pré‐clínicos têm sugerido potenciais terapias para EB, muitas delas dependentes da variante patogênica associada.16–20 O avanço no diagnóstico genético por meio da tecnologia de NGS, caminhando junto com o desenvolvimento de novas terapias moleculares, promete trazer benefícios significativos a esse grupo de pacientes.21 Ainda que não seja uma realidade atual, é possível que, em um futuro não muito distante, pacientes com variantes patogênicas específicas tenham possibilidade de tratamento.
Relação custo‐efetividade do painel de genesEmbora o custo estimado por amostra para o painel de genes (R$ 800,00) seja maior comparado ao do MIF (R$ 500,00), ele é consideravelmente menor em relação à MET (US$ 980,00; R$ 3.920,00). É importante notar, entretanto, que o custo da MET foi baseado nos preços praticados em laboratórios dos EUA, visto que, ao que sabemos, o método não é oferecido comercialmente no Brasil. Ainda que algumas universidades brasileiras disponham de um microscópio eletrônico, a realização do método para diagnóstico da doença se dá apenas em contexto acadêmico para casos de específico interesse científico. O sequenciamento de Sanger apresenta um preço médio de R$ 35,00 por fragmento gênico analisado (geralmente um éxon), por amostra. Assim, o valor final dessa metodologia depende do número de éxons dos genes analisados. Hipoteticamente, se um indivíduo com EB distrófica (associada ao gene COL7A1) fosse analisado por sequenciamento de Sanger, seria investido um valor de aproximadamente R$ 2.520,00 apenas com essa metodologia (levando‐se em consideração um número de 72 pares de primers que englobam os 118 éxons do gene, como descrito por Christiano et al.22).
Desse modo, ainda que o painel de genes não seja um método de baixo custo, o resultado obtido a partir dele é mais informativo e conclusivo do que o obtido por MIF e MET. Em comparação ao sequenciamento de Sanger, o valor a ser investido é menor, assim como o tempo de processamento, evidenciando o custo‐efetividade do painel de genes para o diagnóstico de EB. Além disso, o preço dessa metodologia vem diminuindo ao longo do tempo, o que deve tornar o método mais acessível nos próximos anos.10
Cabe ressaltar, porém, que o custo estimado nesse estudo para o diagnóstico por meio do painel de genes não incluiu o preço da compra do painel (aproximadamente R$ 20.000,00) e o investimento para validá‐lo. Nossa proposta aqui é o diagnóstico genético no contexto de saúde pública, visando, em longo prazo, incluir o maior número de pacientes com EB do Brasil. Assim, à medida que mais pacientes são analisados, o custo investido no painel, por paciente, vai diminuindo, até o momento que se torna irrisório. Além disso, não foi adicionado ao cálculo o custo a ser investido em um analista com expertise para análise dos resultados. Em nossa experiência, realizada em contexto acadêmico, essa questão não se aplicou; entretanto, deve‐se levá‐la em consideração antes de estabelecer o método no âmbito de assistência pública.
Panorama brasileiro: o exame clínico não resolve todos os casos de EBO diagnóstico da EB no Brasil é feito, na maioria dos casos, por avaliação dos sinais clínicos apresentados. São poucos os centros especializados que disponibilizam os métodos de diagnóstico de EB, o que resulta em dificuldade para diagnosticar e subclassificar os pacientes e, consequentemente, fornecer correto acompanhamento e aconselhamento genético.
Mesmo quando disponível, o MIF frequentemente proporciona resultados inconclusivos, em razão da falta de analistas experientes, especialmente em regiões brasileiras mais pobres e de difícil acesso. Levando‐se em consideração o dano causado pelas biópsias de pele e que a maior parte dos pacientes não tem as condições apropriadas (incluindo os curativos e os cuidados de saúde adequados), alguns médicos têm optado por não indicar MIF para subclassificar EB. Consequentemente, o diagnóstico preciso e a definição do tipo de EB dependem de um exame clínico completo. Entretanto, mesmo se realizado por dermatologista especialista em EB, o exame clínico não é suficiente para resolver todos os casos, tendo em vista a extrema variabilidade fenotípica observada. A tabela 2 apresenta um exemplo de caso clínico cujo resultado genético não coincidiu com a hipótese clínica prévia, surpreendendo os dermatologistas. Casos similares foram descritos em estudos anteriores e indicaram que o efeito das variantes genéticas pode resultar em fenótipos clínicos atípicos.23,24
O exame clínico não resolve todos os casos de EB
| Caso clínico |
|---|
| Paciente de 32 anos de idade, diagnosticada tardiamente com epidermólise bolhosa (aos 15 anos) em razão de leve quadro clínico que apresentava. Os sintomas da paciente são sutis em comparação aos casos mais graves de EB. A distribuição das bolhas é localizada na região pré‐tibial e apenas presença discreta de bolhas é encontrada nas mãos. Distrofia de unhas é observada somente nas unhas dos pés. |
| A paciente foi submetida a exame de biópsia, cujo resultado se mostrou inconclusivo. O exame clínico realizado por dermatologistas especialistas sugeriu como provável tipo de EB: EB simples localizada. |
| A análise genética pelo painel de genes foi contrastante com o que era esperado pela clínica: foram encontradas duas variantes patogênicas no gene COL7A1, revelando tratar‐se de um caso de EB distrófica recessiva (EBDR). |
| O fenótipo mais conhecido de EBDR é bastante grave e envolve alta indução e distribuição generalizada de bolhas e pseudosindactilia. Formas leves de EBDR são mais raras e de difícil determinação por exame clínico. Estudos têm demonstrado que a alteração genética tem grande influência na apresentação clínica. Ambas as variantes patogênicas identificadas na paciente atuam alterando o splicing de COL7A1. Outros casos muito leves de EBDR já foram associados a esse mesmo tipo de variante. |
| O caso aqui relatado exemplifica a importância da análise genética para determinação correta do tipo de EB. Uma vez que o padrão de herança varia entre os tipos de EB (EBS: dominante, EBJ: recessivo, EBD: dominante ou recessivo), o conhecimento da sua classificação é essencial para o correto aconselhamento genético. |
Em nossa experiência brasileira, constatamos que, para a maior parte dos casos, o preciso diagnóstico acerca do tipo de EB associado só foi possível após o resultado genético, que por sua vez tornou possível o correto aconselhamento genético. Além disso, algumas peculiaridades do país também são importantes. As taxas de endogamia e consanguinidade são maiores em algumas populações brasileiras, especialmente na região Nordeste, o que sugere uma alta prevalência de herança genética recessiva.25,26 De fato, em estudo anterior encontramos que 82,7% dos pacientes brasileiros com subtipos de EBD apresentavam herança recessiva.13 A determinação do padrão de herança é especialmente importante para o diagnóstico dos subtipos localizados de EBD, uma vez que as formas dominante e recessiva são clinicamente indistinguíveis, o que prejudica o correto aconselhamento na ausência de teste genético.
Considerando a dificuldade de se realizar um diagnóstico clínico preciso e, particularmente, a complexidade de classificar a EB, o resultado genético tem provado ser essencial para a correta determinação do tipo de EB associado.
Painel de genes como primeira opção para o diagnóstico da EBO levantamento realizado neste estudo demonstrou que o painel de genes é uma ferramenta efetiva para o diagnóstico conclusivo da EB, com alta eficiência, precisão diagnóstica e reduzido tempo de análise (cerca de 10 dias), economicamente viável e que dispensa a necessidade de exames invasivos (fig. 1). Alguns fatores, entretanto, são essenciais para a efetividade do método. O processamento dos resultados do painel de genes requer suporte de bioinformatas e o tempo de análise das variantes depende da experiência do investigador. Entretanto, em comparação com os métodos de MIF e MET, a interpretação dos resultados do painel de genes é mais simples, e não requer uma equipe com profunda expertise.3
Tendo em vista a maior eficiência diagnóstica, a robustez do resultado obtido, o preço comparável às técnicas hoje já realizadas e a possibilidade de aconselhamento genético, de prognóstico e de diagnóstico pré‐natal, o presente estudo apoia a utilização do painel de genes como primeira escolha para o diagnóstico da EB no Brasil. Levando‐se em consideração as dimensões continentais do território brasileiro e a dificuldade de se introduzir uma técnica minuciosa como o painel de genes, o estabelecimento de um centro de referência para o diagnóstico da EB no país traria muitos benefícios. A centralização das análises em um único lugar dispensa a necessidade de criação de múltiplas equipes especializadas e reduz o custo de processamento por amostra.
A dificuldade de diagnóstico dos pacientes com EB no Brasil pode ser atenuada com a abordagem metodológica aqui proposta. Para a efetivação da ideia, são necessários recursos financeiros e apoio governamental. É essencial que as autoridades atentem para esse grupo de pacientes que apresenta uma doença genética grave e que, ainda que rara, representa um conjunto de mais de 900 brasileiros.
ConclusãoO painel de genes de EB apresenta alta eficiência de diagnóstico e dispensa a necessidade de biópsia, contrastando com os métodos majoritariamente empregados. O custo por paciente da metodologia apresentada é comparável àquele das demais técnicas e tende a diminuir nos próximos anos. A detecção das variantes associadas à EB possibilita o prognóstico da doença, o diagnóstico genético pré‐natal e pré‐implantacional, e tem implicações para o aconselhamento genético. Por fim, embora ainda em estágios pré‐clínicos, potenciais terapias para EB têm mostrado resultados promissores para alguns tipos específicos de variantes genéticas. Os resultados aqui apresentados apoiam o painel de genes combinado com o exame clínico como primeira escolha para o diagnóstico da EB no Brasil.
Suporte financeiroEste estudo teve suporte financeiro do Instituto Nacional de Ciência e Tecnologia de Genética Médica Populacional (INaGeMP/CNPq/FAPERGS), n° 465549/2014‐4, e da Coordenação de Aperfeiçoamento de Pessoal de Nível Superior (CAPES).
Contribuição dos autoresLuiza Monteavaro Mariath: Elaboração e redação do manuscrito; revisão crítica da literatura, concepção e planejamento do estudo; análises genéticas e de bioinformática.
Ana Elisa Kiszewski: Aprovação da versão final do manuscrito; revisão crítica do manuscrito; concepção e planejamento do estudo; recrutamento de pacientes; avaliações dermatológicas dos pacientes.
Jeanine Aparecida Frantz: Aprovação da versão final do manuscrito; revisão crítica do manuscrito; recrutamento de pacientes; avaliações dermatológicas dos pacientes.
Marina Siebert: Aprovação da versão final do manuscrito; revisão crítica do manuscrito; análises genéticas; avaliação de custo‐efetividade do painel de genes.
Ursula Matte: Aprovação da versão final do manuscrito; revisão crítica do manuscrito; avaliação de custo‐efetividade do painel de genes.
Lavínia Schuler‐Faccini: Aprovação da versão final do manuscrito; revisão crítica do manuscrito; concepção e planejamento do estudo; revisão da literatura.
Conflito de interessesNenhum.
O nosso muito obrigada às colaboradoras Juliana Tosetto Santin e Maria Juliana Rodovalho Doriqui, por sua essencial contribuição para o estudo da epidermólise bolhosa no Brasil. Nosso agradecimento especial aos pacientes e familiares, pela participação no estudo. Em particular, agradecemos às associações de epidermólise bolhosa do Brasil e de suas diversas regiões: DEBRA‐Brasil, APPEB‐DF, ACPAPEB, APPAPEB, AAPEB‐SP e AMAPEB. A todos os médicos e profissionais que colaboram com nosso estudo, o nosso muito obrigada. Em especial, aos profissionais do estado do Maranhão: Eduardo Henrique Jorge Lago, Maria do Perpétuo Socorro Balby Pires e Cleidimar Souza Cutrim.
Como citar este artigo: Mariath LM, Kiszewski AE, Frantz JA, Siebert M, Matte U, Schuler‐Faccini L. Gene panel for the diagnosis of epidermolysis bullosa: proposal for a viable and efficient approach. An Bras Dermatol. 2021;96:155–62.
Trabalho realizado na Universidade Federal do Rio Grande do Sul, Porto Alegre, RS, Brasil.