Dermatite atópica (DA) é doença crônica, multifatorial, cujo fenótipo clínico resulta da interação de fatores genéticos e ambientais.1 A desregulação imunológica e a integridade da barreira cutânea determinam sua gravidade, predispondo a infecções e permeabilidade a antígenos.2 É motivo comum de atendimento dermatológico, principalmente na infância (< 12 anos), representando 25,8% das consultas dermatológicas.3
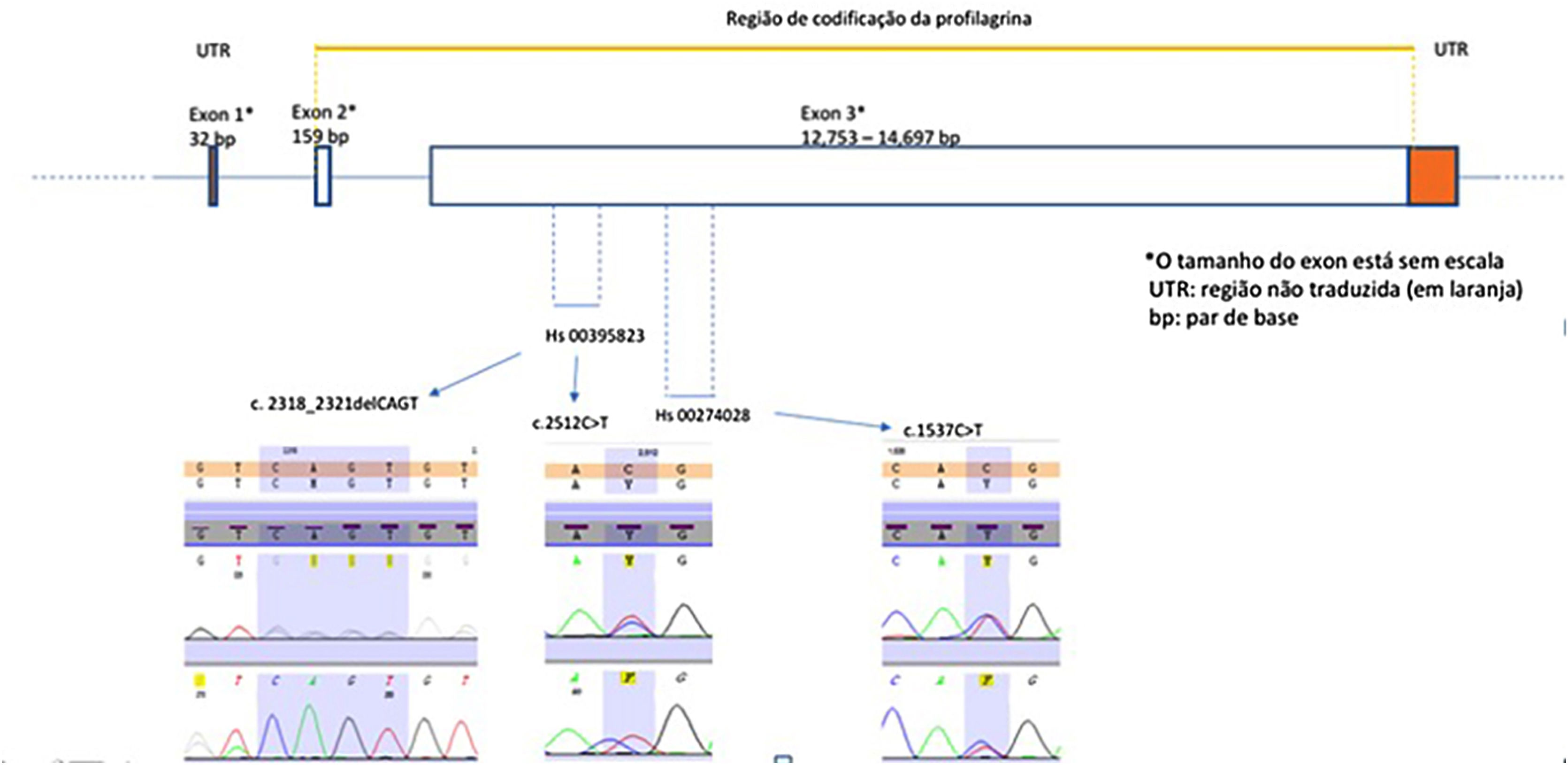
O gene que codifica a filagrina (FLG) é altamente polimórfico, na região do complexo de diferenciação epidérmica (1q21.3), codificando as proteínas mais importantes envolvidas na homeostase da barreira cutânea. FLG é o principal fator genético associado à DA, e seu éxon‐3 transcreve a maior parte da proteína profilagrina. Alterações da barreira cutânea estão presentes em pacientes com DA sem alterações do gene FLG; entretanto, a presença de variantes que levam à perda de função foram associadas a fenótipos clínicos como doença persistente de início precoce, asma e sensibilização alérgica.1,4 Intensa disparidade étnica na frequência de variantes que levam à perda de função do gene FLG associadas à DA tem sido observada.5
Mais de 60 variantes do gene FLG que levam à perda de função do gene FLG foram identificadas em associação com DA; as mais comuns entre os europeus são c.1537C>T:R501X e 2282del4:S761Cfs*36 e, entre os africanos subsaarianos, c.9947C>G:S3316*. Poucos estudos foram realizados em pacientes latino‐americanos com DA. Nosso objetivo foi avaliar a frequência de variantes do gene FLG (no éxon‐3) entre pacientes com DA para comparar populações brasileiras e internacionais e explorar suas características clínicas.
Foi realizado estudo transversal no ambulatório de dermatologia (FMABC; Santo André, São Paulo). Oitenta pacientes com DA (critérios de Hanifin e Rajka) de ambos os sexos foram incluídos e examinados por dermatologista experiente para avaliar a gravidade da doença (SCORAD, EASI) e coletar amostras de sangue venoso para análise laboratorial e da mucosa oral para análise genética. Os participantes/responsáveis assinaram termo de consentimento livre e esclarecido.
A coleta para análise genética foi realizada por swab da mucosa da região bucinadora dos pacientes e colocado num tubo de ensaio estéril (Oragene Collector OG‐500®, DNA Genotek Inc., Kanata, Ontario), que foi submetido ao sequenciamento pelo método de Sanger.
A extração de DNA foi realizada utilizando precipitação com etanol, e um reagente prepIT 2P fornecido pelo kit Oragene.
A reação em cadeia da polimerase (PCR) e a análise de seu sequenciamento foram realizadas com foco no éxon‐3 para identificar as variantes genéticas mais comuns – c.1537C>T:R501X (rs61816761) e c.2282del4:S761Cfs*36 (rs558269137) utilizando primers validados da Thermo Fischer Scientific® (Applied Biosystems, Foster City, CA), (Hs00274028 forward: 5’CTA ACA CTG GAT CCC TGG TTC CTA 3’ e reverse 5’ CTG AGA CAG CAG AGC CAC CAA GA 3’ e Hs00395823, forward: 5’ CAG ACC TAT CTA CCG ATT GCT CGT 3’ e reverse: 5’ AAA TCA GGC ACTCGT CAC ACA CAG AA 3’). Essa estratégia possibilitou investigar outras variantes em áreas vizinhas ao loci alvo, mas não cobriu toda a região de codificação do éxon‐3 (fig. 1). Os produtos de PCR foram purificados com esferas de DNA (Agencourt – AMPure XP‐Beckman Coulter, Brea, CA). As amostras purificadas juntamente com 10 μL desses primers foram utilizadas para a reação de sequenciamento. O ciclo de sequenciamento foi realizado com o kit Big Dye Terminator v3.1 (Thermo Fisher Scientific). Os produtos de sequenciamento foram submetidos à eletroforese capilar no ABI 3500 DNA Analyzer (Applied Biosystems, Foster City, CA). Os dados de sequenciamento foram avaliados com o software Seq A(14) – Applied Biosystems, Foster City, CA. O PROVEAN (Protein Variation Effect Analyzer) v1.1 foi usado para prever se uma variação de sequência de proteína causada por uma substituição missense afetaria a função da proteína (disponível em https://provean.jcvi.org/index.php).
A prevalência de cada variante identificada foi comparada ao banco de dados público brasileiro de variantes genômicas (ABraOm; hg38 – https://abraom.ib.usp.br/) com 1.171 amostras da população da mesma região e um banco de dados internacional (genomAD; v3.1.2 – https://gnomad.broadinstitute.org/) com 76.156 indivíduos não aparentados de diferentes etnias. A significância estatística foi definida como p ≤ 0,001.6
Dados demográficos principais, gravidade clínica, eosinofilia e níveis de IgE estão na tabela 1. A maioria dos pacientes apresentava DA moderada e grave (79%), níveis elevados de IgE (98%) e eosinofilia (68%).
Características principais clínicas, demográficas e laboratoriais da amostragem de DA (n=80)
| Variáveis | Valores |
|---|---|
| Sexo, n (%) | |
| Feminino | 40 (50) |
| Masculino | 40 (50) |
| Idade (anos), média (DP) | 16 (12) |
| Fototipo, n (%) | |
| I–II | 24 (30) |
| III–IV | 51 (64) |
| V–VI | 5 (6) |
| Etnia, n (%) | |
| Caucasiana | 54 (68) |
| Asiática | 1 (1) |
| Africana | 25 (31) |
| Gravidade da DA, n (%) | |
| Leve | 17 (21) |
| Moderada | 45 (56) |
| Grave | 18 (23) |
| SCORAD, média (DP) | 36 (17) |
| EASI, média (DP) | 13.5 (10.2) |
| Níveis elevados de IgEa, n (%) | 79 (98) |
| Eosinófilos>5%, n (%) | 46 (68) |
DA, dermatite atópica; IgE, imunoglobulina sérica E; DP, desvio padrão; SCORAD, Scoring Atopic Dermatitis; EASI, Eczema Area Severity Index.
Os resultados da análise genética da amostra de DA e a comparação com os dois controles populacionais estão na tabela 2. Vinte e seis variantes genéticas do éxon‐3 do gene FLG foram detectadas em 60 pacientes (75%; 95%IC: 65%–85%). Homozigose e heterozigose composta não foram identificadas.
Variantes do gene FLG (éxon 3) em 80 pacientes brasileiros com dermatite atópica e sua prevalência de acordo com os bancos de dados populacionais AbraOM (da mesma população brasileira) e com o gnomAD (de populações latinas, afro‐americanas, europeias e internacionais)
| Variante | Zigosidade | dbSNP | Consequência proteica | DA, n (%) | AbraOM (Brasil) | gnomAD | |||
|---|---|---|---|---|---|---|---|---|---|
| Latina/ miscigenada | Europeia | Afro‐americana | Internacional | ||||||
| c.1396A>G | HT | rs2011331 | T454A | 38 (47,50) | 33,09% | 39,36% | 16,02% | 47,84% | 30,25% |
| c.1468C>T | HT | rs11584340 | P478S | 39 (48,75) | 25,06% | 35,76% | 15,90% | 13,81% | 20,34% |
| c.1476T>C | HT | rs561848191 | Sinônimo | 2 (2,50) | <0,01% | 0,01% | <0,01% | 0,53% | 0,14% |
| c.1521G>A | HT | rs75530805 | Sinônimo | 1 (1,25) | NA | <0,01% | <0,01% | <0,01% | <0,01% |
| c.1521G>C | HT | rs75530805 | Sinônimo | 21 (26,25) | 1,49% | 0,60% | <0,01% | 5,1% | 1,47% |
| c.1530G>C | HT | rs13376095 | E498D | 3 (3,75) | 0,05% | 0,22% | <0,01% | 2,62% | 0,74% |
| c.1537C>T | HT | rs61816761 | R501Xa | 3 (3,75) | 0,08% | <0,01% | <0,01% | <0,01% | <0,01% |
| c.1591C>A | HT | rs12036682 | H519N | 1 (1,25) | 0,03% | 0,05% | 0,06% | <0,01% | 0,58% |
| c.1665T>G | HT | rs152285733 | Sinônimo | 8 (10,00) | NA | NA | NA | NA | NA |
| c.1712A>G | HT | rs546475787 | H559R | 6 (7,50) | <0,01% | <0,01% | <0,01% | <0,01% | <0,01% |
| c.1737A>C | HT | rs71625187 | Sinônimo | 25 (31,25) | NA | <0,01% | <0,01% | <0,01% | <0,01% |
| c.1777A>T | HT | rs145627745 | T581S | 31 (38,75) | 0,09% | 0,49% | 1,37% | 0,27% | 0,79% |
| c.1800T>C | HT | rs152285598 | Sinônimo | 14 (17,50) | NA | NA | NA | NA | NA |
| c.2167C>T | HT | rs115087788 | R711C | 1 (1,25) | <0,01% | 0,20% | <0,01% | 0,82% | 0,25% |
| c.2210C>T | HT | rs3120655 | T725I | 13 (16,25) | 8,07% | 3,53% | 0,17% | 34,83% | 9,98% |
| c.2217C>A | HT | rs7512779 | H727Q | 2 (2,50) | 1,96% | <0,01% | <0,01% | <0,01% | <0,01% |
| c.2261C>A | HT | rs3120654 | S742Y | 13 (16,25) | 8,03% | 3,47% | 0,13% | 34,77% | 10,06% |
| c.2271T>C | HT | rs150144110 | s745= | 1 (1,25) | 0,01% | <0,01% | <0,01% | 0,19% | <0,01% |
| c.2299G>A | HT | rs74129461 | E755K | 23 (28,75) | 23,40% | 34,96% | 15,90% | 7,73% | 18,57% |
| c.2282del4CAGT# | HT | rs558269137 | S761Cfs*36# | 3 (3,75) | 0,06% | <0,01% | 2,19% | <0,01% | 1,26% |
| c.2319A>C | HT | rs11204979 | Sinônimo | 2 (2,50) | 0,82% | 0,59% | <0,01% | 5,41% | 1,57% |
| c.2377G>C | HT | rs148739675 | D781H | 1 (1,25) | 0,21% | 0,07% | <0,01% | 0,66% | 0,19% |
| c.2396C>T | HT | rs77032592 | S787F | 1 (1,25) | 0,56% | 0,28% | <0,01% | 3,26% | 0,94% |
| c.2512C>T | HT | rs115746363 | R826Xa | 2 (2,50) | 0,13% | <0,01% | <0,01% | 0,72% | 0,21% |
| c.2544T>C | HM | rs3120653 | Sinônimo | 37(46,25) | 33,60% | <0,01% | <0,01% | 0,02% | <0,01% |
dbSNP (rs), referência SNP número nas bases de dados internacionais (posição cromossômica); HT, heterozigose; HM, homozigose; DA, dermatite atópica; NA, não achada.
Variantes do gene FLG com perda de função da filagrina foram observadas em oito pacientes com DA (10%; 95% IC 3%–17%). Duas variantes prevalentes no mundo (c.1537C>T:R501X e 2282delCAGT:S761Cfs*36) foram observadas em seis pacientes (7,5%). Outra variante patogênica (c.2512C>T:R826X) foi identificada em dois pacientes (2,5%). Essas três variantes patogênicas foram mais prevalentes na amostra de DA que nos controles brasileiros (p<0,001). A variante 2282delCAGT:S761Cfs*36 é comum em europeus, enquanto c.2512C>T:R826X é comum entre afro‐americanos.
Foram encontradas oito variantes sinônimas do gene FLG, quatro delas amplamente distribuídas no Brasil e no mundo. Entretanto, quatro delas (c.1665T>G:rs152285733, c.1737A>C: sinônimo, c.1521G>A: sinônimo e c.1800T>C: sinônimo) foram observadas como mais comuns na amostra de DA do que no mundo (p <0,001). A variante c.1476T>C: sinônimo, rara na população brasileira e internacional (p <0,001), é comum entre afro‐americanos. O sinônimo c.2544T>C, considerado comum entre pacientes com DA e controles regionais, é extremamente raro no mundo (p <0,001).
Catorze variantes missense foram detectadas; duas delas (c.1712A>G:H559R, c.1777A>T:T581S) foram mais comuns entre pacientes com DA que controles regionais e internacionais (p <0,001).
Nenhuma variante do gene FLG foi associada à gravidade clínica da DA, eosinofilia ou IgE sérica elevada (p> 0,1).
Esses resultados reforçam o alto polimorfismo do éxon‐3 do gene FLG e sua associação étnica, dificultando a generalização dos resultados genômicos em relação aos fenótipos de DA, especialmente em populações altamente miscigenadas.5,7,8
Segundo a literatura, variantes nulas eram esperadas em 14%–42% dos pacientes com DA, e dessas, 20 encontradas no éxon‐3. Além disso, variações foram relatadas do gene FLG entre pacientes com DA de diferentes etnias.8
A população brasileira é multirracial após 500 anos de miscigenação entre indivíduos da Europa Ocidental, África e Ameríndia.9 As variantes c.2512C>T:R826X e 2282delCAGT:S761Cfs*36 nos pacientes com DA corroboram essas ancestralidades, embora a variante c.2544T>C: sinônimo seja uma característica dessa região.
Considerando associação epidemiológica com o desenvolvimento da doença, variantes do gene FLG perse não explicam completamente a variação na gravidade da DA, eosinofilia ou níveis elevados de IgE, reforçando os aspectos multifatoriais da doença.1,8,10
Por fim, identificamos variantes nulas do gene FLG (no éxon‐3) (c.1537C>T:501X, c.2282del4:S761Cfs*36 e c.2512C>T:R826X) em 10% dos pacientes brasileiros com DA, mesmo sem associação com as principais características clínicas da DA.
Suporte financeiroFundo de Apoio à Dermatologia do Estado de São Paulo – Sebastião Sampaio (FUNADERSP).
Contribuição dos autoresCristina Marta Maria Laczynski: Concepção e desenho, aquisição, análise e interpretação dos dados.
Carlos D’Apparecida Santos Machado Filho: Revisão crítica do conteúdo intelectual.
Hélio Amante Miot: Revisão crítica do conteúdo intelectual.
Denise Maria Christofolini: Revisão crítica do conteúdo intelectual.
Itatiana Ferreira Rodart: Processamento do material genético, análise e interpretação dos dados e revisão do conteúdo intelectual.
Paulo Ricardo Coelho: Revisão crítica do conteúdo intelectual.
Todos os autores leram e aprovaram a versão final do manuscrito.
Conflito de interessesNenhum.
Aos pacientes que consentiram em participar deste estudo, ao Prof. Caio Parente Barbosa, do Departamento de Genética do Centro Universitário FMABC, a Juliana Zangirolami Raimundo, do Departamento de Bioestatística do Centro Universitário FMABC, e a Larissa K. Martinez Meirinho Magalhães, do Departamento de Dermatologia do Centro Universitário FMABC.
Como citar este artigo: Laczynski CMM, Machado Filho CDS, Miot HA, Christofolini DM, Rodart IF, Criado PF. Prevalence of filaggrin gene polymorphism (exon‐3) in patients with atopic dermatitis in a multiracial Brazilian population. An Bras Dermatol. 2023;98:240–3.
Trabalho realizado no Centro Universitário FMABC, Santo André, SP, Brasil.