A radiação UVB pode causar danos agudos, como queimaduras solares, ou fotoenvelhecimento e melanoma, que são grandes ameaças à saúde.
ObjetivoEste estudo objetivou investigar o mecanismo do fotoenvelhecimento cutâneo induzido pela radiação UVB em camundongos, utilizando a expressão diferencial de miRNAs.
MétodosFoi construído um modelo de fotoenvelhecimento por radiação UVB. As colorações pelos métodos da hematoxilina‐eosina e de Masson foram utilizadas para avaliar as modificações na epiderme e na derme de camundongos. O perfil de expressão de miRNA no modelo de pele de camundongo exposta à radiação UVB e de camundongos com pele normal foi avaliado utilizando‐se análise de sequência de miRNA. A análise de GO e Pathway foi empregada para prever os alvos de miRNA.
ResultadosTotal de 23 miRNAs foram avaliados para expressões significativamente diferentes em comparação com a pele normal. Entre eles, sete miRNAs foram up‐regulados e 16 foram down‐regulados no na pele com fotoenvelhecimento de camundongos expostos à radiação UVB. A expressão diferencial de miRNA está relacionada a uma variedade de vias de transdução de sinal, dentre as quais as vias de sinal mmu‐miR‐195a‐5p e mitogen‐activated protein kinase (MAPK) são cruciais. Houve uma expressão diferencial significativa de miRNA no tecido cutâneo de camundongos normais em comparação com a pele com fotoenvelhecimento cutâneo induzido pela radiação UVB.
Limitações do estudoEm virtude das restrições de tempo e energia, a verificação do nível de proteína específico, a exploração da via MAPK e o mecanismo molecular downstream do miR‐195a‐5p precisam ser estudados mais profundamente no futuro.
ConclusõesO fotoenvelhecimento cutâneo induzido por radiação UVB pode ser diagnosticado e tratado utilizando miRNA.
A radiação ultravioleta solar é um importante carcinógeno ambiental. Há três regiões, de acordo com os diferentes comprimentos de onda: a radiação UVC de onda curta tem de 200 a 280nm; a UVB de onda média tem de 280 a 320nm; e a UVA de onda longa tem de 320 a 400nm.1,2 A UVB tem dois lados. De acordo com diferentes doses de radiação, diferentes modelos de doenças apresentarão efeitos diferentes. Muitos pesquisadores utilizam a UVB para tratar doenças humanas, como o vitiligo.3 O modelo utilizado neste artigo é o modelo de fotoenvelhecimento da pele de camundongos. A radiação UVB pode causar danos agudos, como queimaduras solares, ou fotoenvelhecimento e melanoma, que são grandes ameaças à saúde. Em virtude de seu comprimento de onda curto e grande intensidade, a UVB (290‐320nm) causa danos significativos à epiderme. Os ceratinócitos são suas células‐alvo, o que pode induzir reações adversas na pele como edema, eritema, bolhas, pigmentação da pele e fotoenvelhecimento prematuro.4,5 Se essas lesões não forem reparadas a tempo, podem ocorrer graves defeitos estruturais das moléculas de DNA, destruindo a vitalidade das células e afetando sua função, e até mesmo levar ao envelhecimento da pele, inclusive ao câncer de pele.6
Os microRNAs (miRNAs) são considerados um grupo de pequenos RNAs endógenos não codificantes (cerca de 23 nucleotídeos), associados à supressão de genes pós‐transcricionais.7,8 Os miRNAs agem principalmente na regulação pós‐transcricional e são importantes na manutenção da homeostase celular normal.9 Vários estudos indicam que os miRNAs participam de vários processos biológicos, incluindo desenvolvimento, diferenciação, reprodução e apoptose. Recentemente, pesquisadores descobriram uma relação entre miRNAs e a ocorrência e progressão de algumas doenças de pele, tais como miRNAs e câncer de pele, pigmentação, doenças inflamatórias, doenças autoimunes, cicatrização de feridas e fotoenvelhecimento.10–13 Representando aproximadamente 1% a 5% do genoma humano, miRNAs têm cerca de 30% dos genes que codificam proteínas reguladoras,14 e sua importância para todo o organismo é evidente. Por outro lado, o mecanismo do fotoenvelhecimento da pele mediado pela exposição à UVB permanece desconhecido, e mais pesquisas podem determinar a relação mecânica entre miRNAs e radiação UVB, auxiliando na prevenção de tais lesões, além de fornecer biomarcadores diagnósticos e novos alvos de ácidos nucleicos para tratamento. A expressão diferencial de miRNAs no fotoenvelhecimento cutâneo foi pesquisada utilizando radiação UVB e tecido de pele normal, usando a tecnologia de detecção de sequência de miRNAs para explorar o mecanismo de fotoenvelhecimento cutâneo causado por UVB.
Material e métodoModelos animaisDoze camundongos C57BL/6 machos adultos foram selecionados para este estudo. Os camundongos foram obtidos do Centro de Animais Experimentais do Third Xiangya Hospital, Central South University, China. Os camundongos tinham cerca de 8 semanas de idade, pesavam 25 a 30g e foram mantidos em uma instalação com temperatura e umidade controladas, com comida e água suficientes. Todos os procedimentos de alimentação dos animais foram realizados de acordo com as Diretrizes do National Institutes of Health para o cuidado e uso de animais de laboratório. O Comitê de Bioética da Central South University autorizou todos os procedimentos para experimentos com animais.
Radiação ultravioletaOs camundongos foram divididos aleatoriamente em três grupos experimentais e um grupo controle: grupo A controle (com radiação de 0mJ/cm2), grupo B de radiação UVB (com radiação de 90mJ/cm2), grupo C de radiação UVB (com radiação de 180mJ/cm2) e grupo D de radiação UVB (com radiação de 360 mJ/cm2). Os camundongos foram primeiro sedados (injeção intraperitoneal de pentobarbital sódico) e, em seguida, seus dorsos foram depilados. O experimento foi realizado utilizando o espectro UVB (290‐320nm) do SS‐03B Ultraviolet Phototherapy Instrument. Os camundongos dos grupos A, B, C e D foram submetidos à radiação na dose de 0, 90, 180 e 360 mJ/m2, respectivamente. As modificações na pele do dorso dos camundongos foram continuamente monitoradas e fotografadas durante o experimento. No nono dia, os camundongos foram sacrificados e amostras da pele foram coletadas.
Processamento histológico da peleNo nono dia, o tecido cutâneo foi removido. Essas amostras foram fixadas em tampão fosfato 0,1M (PB, pH 7,4) com paraformaldeído a 4% por 24 horas. Os tecidos foram embebidos em parafina e cortes histológicos foram preparados (5μm). Estes foram corados com hematoxilina‐eosina e as lâminas analisadas e fotografadas ao microscópio.
Preparação da bibliotecaA biblioteca foi preparada utilizando o NEBNext® Multiplex Small RNA Library Prep Set for Illumina® e o nanodropND‐1000 Agilent 2100 Bioanalyzer.
A eletroforese em gel de agarose foi utilizada para verificar a integridade do RNA, que foi então quantificado no instrumento NanoDrop ND‐1000. Para remover as modificações de RNA que interferem na construção da pequena biblioteca de sequências de RNA, as amostras de RNA total foram submetidas à desacilação de 3’‐aminoacil (com carga) para 3’‐OH para ligação com o adaptador 3’, fosforilação de 5’‐OH (grupo hidroxilo) para 5’‐P para ligações do adaptador 5’ e desmetilação de m1A e m3C para transcrição reversa eficiente. Para a preparação da biblioteca de sequências de miRNA, o RNA total de cada amostra foi pré‐tratado. Os procedimentos de preparação da biblioteca incluíram: ligação do adaptador 3’, ligação do adaptador 5’, síntese de cDNA, amplificação da reação em cadeia da polimerase (PCR, do inglês polymerase chain reaction), seleção de tamanho de fragmentos amplificados por PCR de ∼134 a 160 pb (correspondendo a RNAs pequenos de aproximadamente 14 a 40 nt). O equipamento Agilent 2100 Bioanalyzer foi utilizado para quantificar as bibliotecas completas. Com base nos resultados da quantificação, as bibliotecas foram combinadas em proporções iguais e usadas para o sequenciamento.
SequenciamentoOs fragmentos de DNA das bibliotecas bem misturadas foram desnaturados com NaOH 0,1M para gerar moléculas de DNA de fita simples e carregados no cartucho de reagente a 1,8 pM. Os sequenciamentos foram realizados no sistema Illumina NextSeq 500 utilizando o kit NextSeq 500/550 V2 (#FC‐404‐2005, Illumina), de acordo com as recomendações da embalagem. O sequenciamento foi realizado executando‐se 50 ciclos.
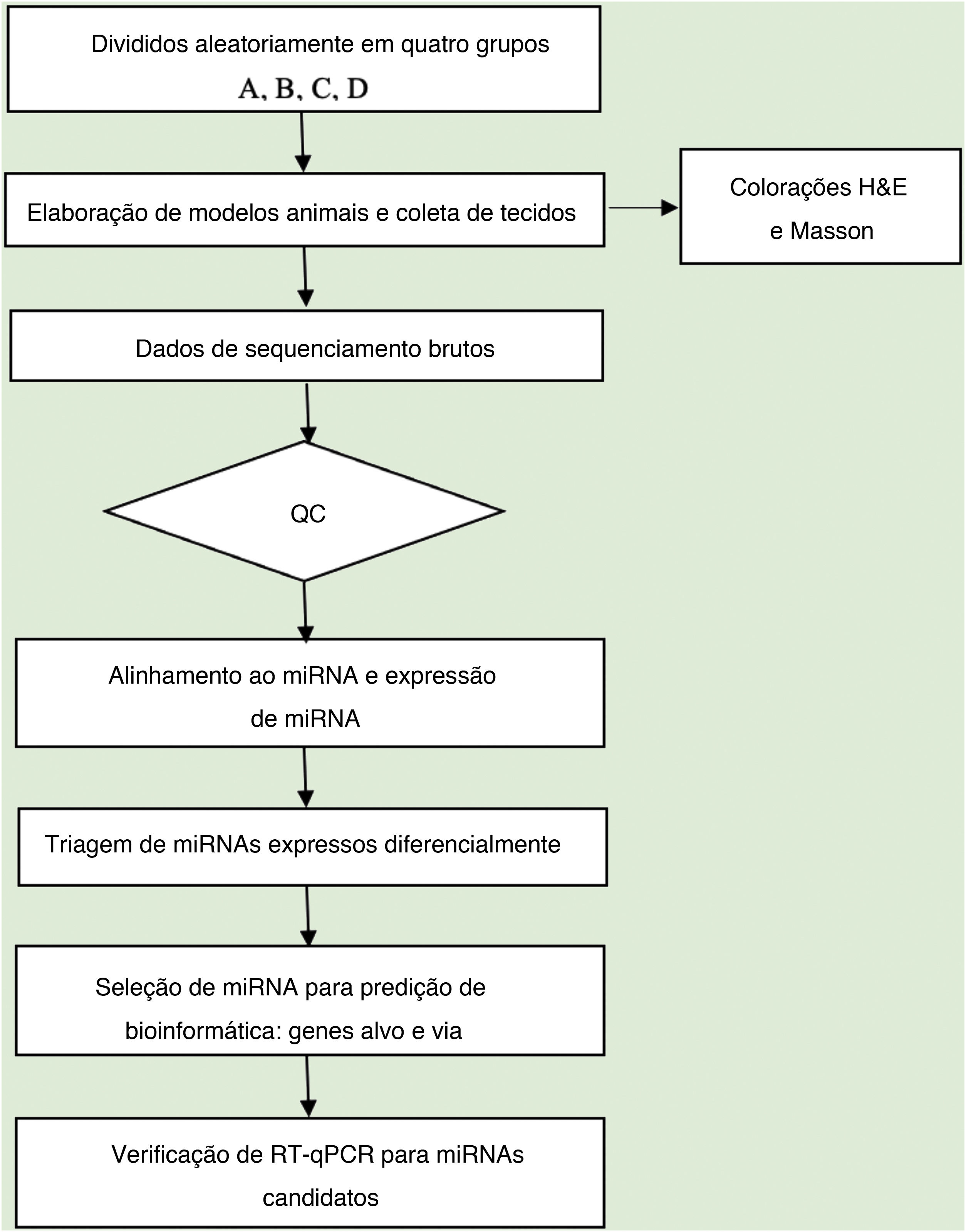
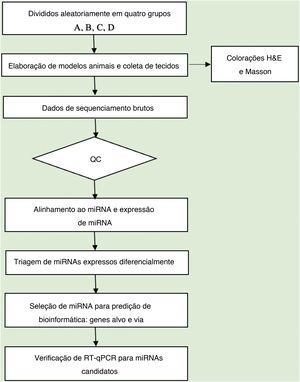
Análise de dadosA qualidade da sequência foi analisada via FastQC e as leituras aparadas foram alinhadas, permitindo apenas uma incompatibilidade. Depois disso, as leituras não mapeadas foram alinhadas, o que possibilitou apenas uma incompatibilidade com o software bowtie.15 Os segmentos lidos restantes foram alinhados, fazendo com que apenas um não correspondesse à sequência de referência do miRNA e ao miRDeep2.16 Com base no número de leituras mapeadas, o perfil de expressão de miRNA pode ser estimado. Os miRNAs expressos diferencialmente são selecionados com base no valor de contagem com o pacote R edgeR.17 O fluxo de trabalho de análise de dados do sequenciamento de miRNAs é mostrado na figura 1. As figuras foram produzidas em ambiente R ou Perl para cálculos estatísticos e gráficos da expressão de miRNAs.
Predição de gene alvo e análise de funçãoEstudos anteriores no TargetScan sugeriram que mirdbV5 prevê alvos de miRNA. Somente os genes que foram previstos por ambos os programas para serem miRNA foram aceitos. Análises de desenvolvimento da via de transdução de sinal com ontologia gênica (OG) e Kyoto Encyclopedia of Gene and Genomes (KEGG) foram realizadas para prever funções de miRNA específicos. O banco de dados OG foi classificado em três categorias: processo biológico, componente celular e função molecular. Para avaliar a rede de genes expressos diferencialmente, utilizou‐se o KEGG, um banco de dados de referência de análise de vias. O significado das palavras OG e termos de rota foram avaliados por meio do valor de p. Quanto menor o valor de p, mais importante é o termo (o valor de p recomendado foi ≤ 0,05).
RT‐qPCRElaboração dos primers: os primers relacionados foram elaborados e sintetizados pelo software na tabela 1. PCR quantitativo em tempo real: o sistema foi estabelecido para análise quantitativa em tempo real. Todas as amostras de cDNA foram configuradas com um sistema de reação de PCR em tempo real. Adiciona‐se a amostra a cada poço correspondente à placa de PCR. A placa de PCR é colocada no equipamento de PCR em tempo real para realizar a reação de PCR. Os genes alvo e os genes housekeeping de cada amostra são submetidos a uma reação de PCR em tempo real. O instrumento gera os achados de concentração dos genes alvo e genes housekeeping em cada amostra com base na curva padrão de diluição do gradiente de DNA. Os níveis relativos de miRNA são normalizados de acordo com o snRNA U6, e a média e o erro padrão são calculados.
Análise estatísticaOs dados foram apresentados como média±desvio padrão. A expressão de miRNA seguiu uma distribuição discreta. Assim, as diferenças significativas entre os grupos foram comparadas por meio de uma distribuição binomial negativa. Os miRNAs diferencialmente expressos foram então selecionados. O valor de p recomendado foi ≤ 0,05.
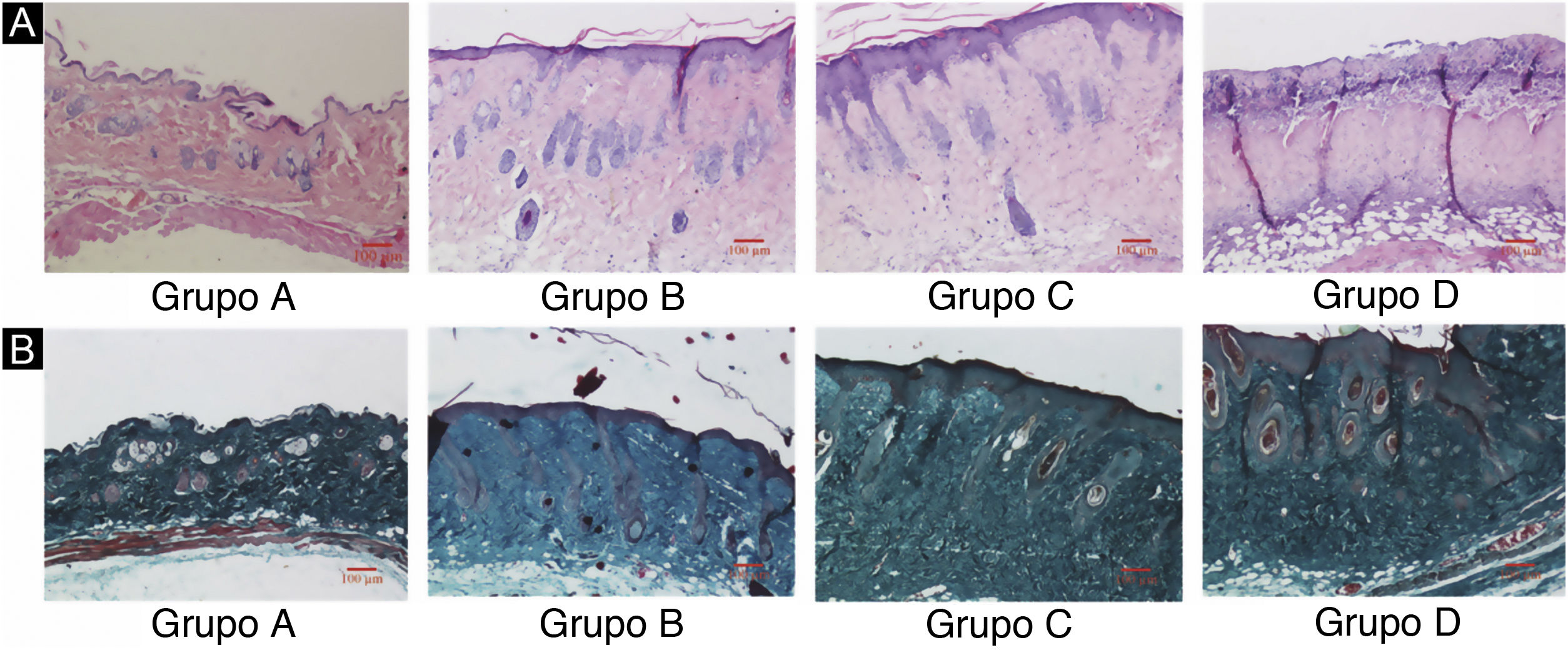
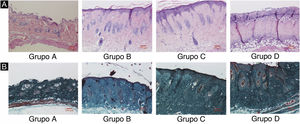
ResultadosDanos nos tecidos cutâneos induzidos por UVBAs colorações pelos métodos da hematoxilina‐eosina e de Masson são mostradas na figura 2. Em comparação com o grupo controle A, a epiderme dos grupos experimentais B/C/D aumentou em diferentes níveis. Além disso, a espessura aumentou com o aumento da dose de radiação. A derme do grupo controle A apresentou distribuição uniforme do tecido conjuntivo, enquanto a derme dos grupos experimentais B/C/D apresentou um arranjo desordenado das fibras. Comparado com o grupo controle A, o grupo experimental B apresentou leve espessamento epidérmico, membrana basal e derme papilar intactas e pequena quantidade de infiltração de células inflamatórias, indicando que a radiação ultravioleta exerceu efeito prejudicial na pele. Em comparação com o grupo experimental B, a epiderme do grupo experimental C era mais espessa, a derme papilar estava presente, e a degradação da membrana basal e a infiltração de células inflamatórias puderam ser observadas. Em comparação com o grupo controle A, a espessura da epiderme da pele no grupo experimental D foi significativamente diferente, até mesmo com algumas áreas de necrose e descolamento; o tecido conjuntivo da derme mostrou‐se desorganizado, com degradação da membrana basal e infiltração de células inflamatórias.
Alterações histopatológicas da pele do dorso de camundongos sob diferentes doses de radiação UVB. Consulte a figura A para coloração H&E e a figura B para coloração de Masson. A dose de radiação do Grupo A é de 0mJ/cm2; a dose de radiação do Grupo B é de 90mJ/cm2; a dose de radiação do Grupo C é de 180mJ/cm2; a dose de radiação do Grupo D é de 360mJ/cm2. As barras de escala correspondem a 100μm.
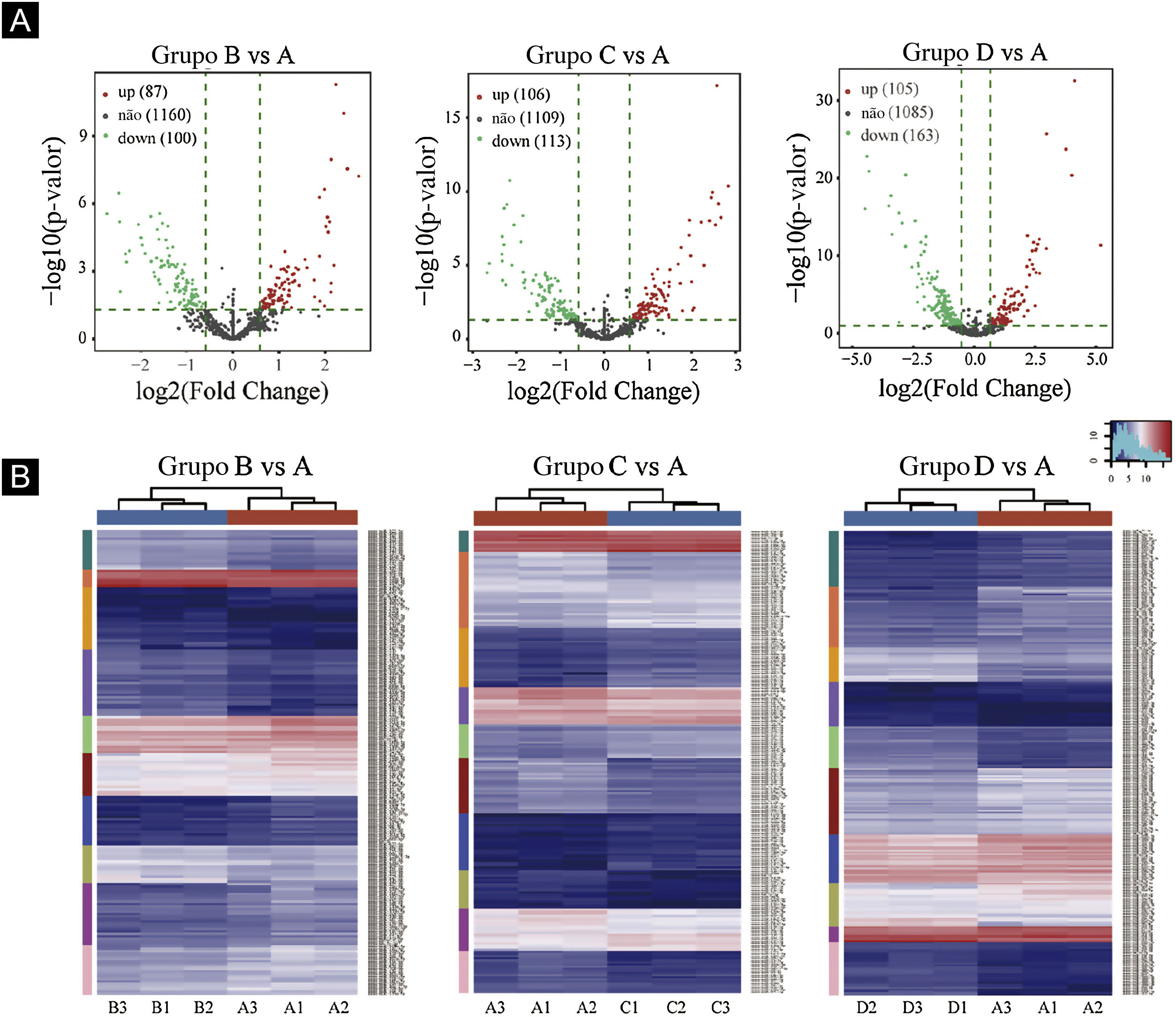
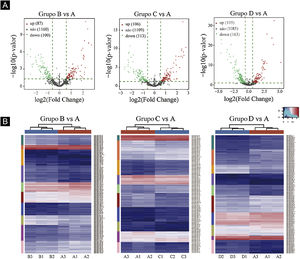
Gráficos de vulcão foram plotados com base em miRNA com valores de CPM (ver fig. 3). De acordo com esses gráficos de vulcão, pode‐se encontrar a expressão diferencial de miRNAs. Há 87 miRNAs up‐regulados e 100 miRNAs down‐regulados na figura 3A (B vs. A). Na figura 3B (C vs. A), há 106 miRNAs up‐regulados e 113 miRNAs down‐regulados. Na figura 3C (D vs. A), há 105 miRNAs up‐regulados e 163 miRNAs down‐regulados.
Triagem de miRNAs expressos diferencialmente. (A) Gráficos de vulcão. Os círculos vermelhos indicam expressão estatisticamente up‐regulada; os círculos verdes indicam expressão down‐regulada; e os círculos cinza indicam miRNAs expressos não diferencialmente. (B) Mapa de calor de agrupamento hierárquico para miRNAs. A escala de cores é mostrada abaixo: azul representa um nível de expressão abaixo da média, e vermelho representa um nível de expressão acima da média.
O agrupamento hierárquico usando miRNAs expressos diferencialmente é mostrado na figura 3. Cada linha representa um miRNA diferente e cada coluna representa uma amostra diferente. Os achados do agrupamento hierárquico revelaram que havia diferenças na expressão de miRNAs entre as amostras. Um total de sete miRNAs significativamente up‐regulados e 16 miRNAs significativamente down‐regulados foram selecionados nos grupos B vs. A, C vs. A e D vs. A (ver fig. 3D/E/F).
Primeiro, os dados originais foram padronizados. Em seguida, 23 miRNAs diferencialmente expressos foram expostos por meio de resultados de sequenciamento de miRNA. Comparados com o grupo Ctrl‐A, 16 miRNAs eram down‐regulados e sete miRNAs eram up‐regulados no grupo de camundongos submetidos à radiação (tabelas suplementares 1 e 2).
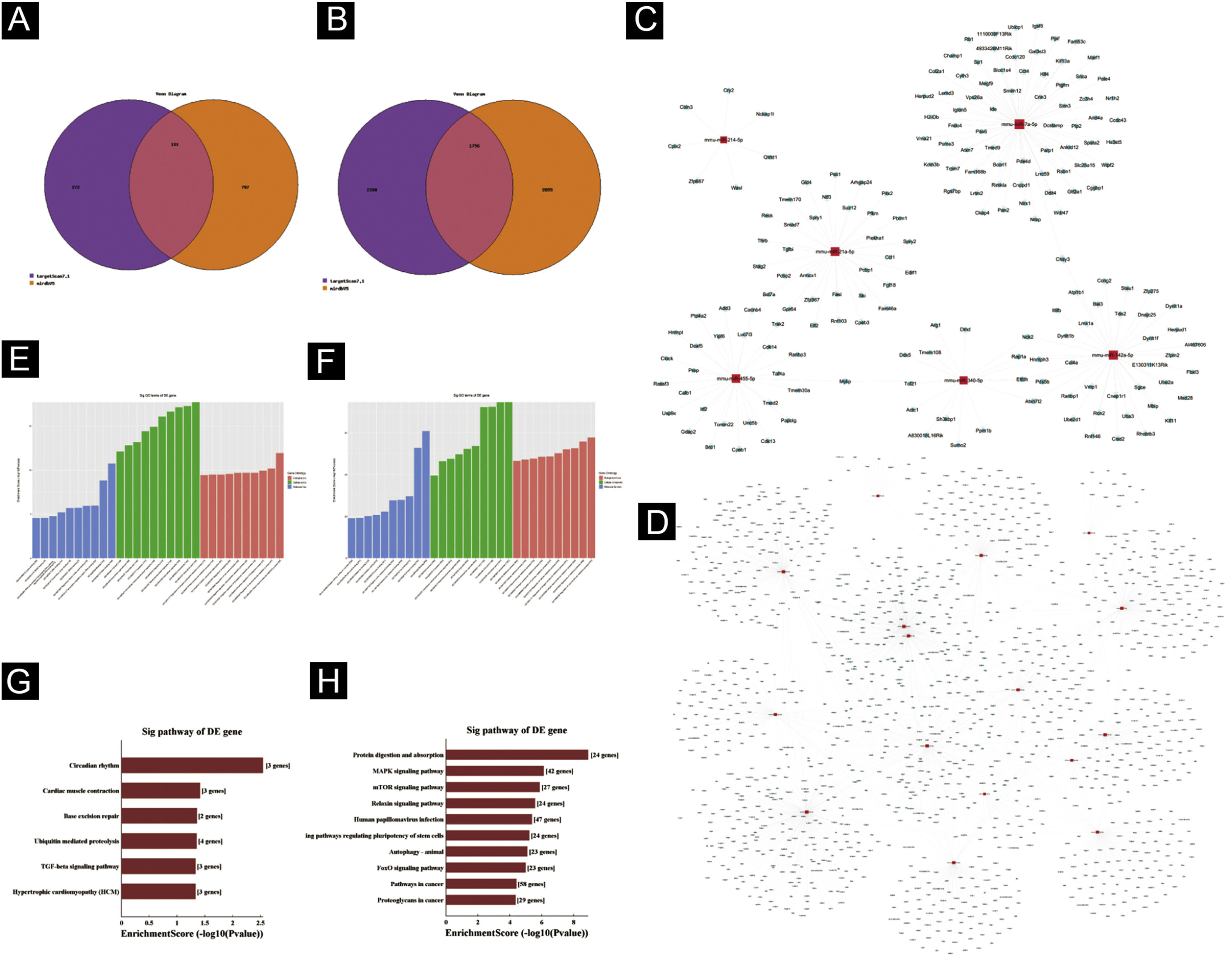
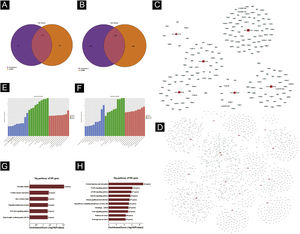
Predição de genes‐alvoTargetscan7.1 e mirdbV5 foram usados para prever os genes alvo dos miRNAs. Targetscan7.1: (de http://www.targetscan.org/mmu_71/). MirdbV5: (de http://mirdb.org/miRDB/.Targetscan7.1) identificou 463 genes alvo nos sete miRNAs up‐regulados; mirdbV5 previu 988 genes alvo, e a interseção de dois genes alvo foi de 191, como mostrado pelo Diagrama de Venn (fig. 4A). Nos 16 miRNAs down‐regulados, targetscan7.1 previu 4146 genes alvo, mirdbV5 previu 4711 genes alvo e a interseção de dois genes alvo foi de 1756, mostrados no Diagrama de Venn (fig. 4B). Utilizando os 191 genes alvo up‐regulados e 1.756 genes alvo down‐regulados que foram previstos, um diagrama de rede (fig. 4C‐D) foi desenvolvido para mostrar a relação entre miRNAs e genes alvo. Os círculos azuis de intersecção na rede representam mRNAs, enquanto os os retângulos vermelhos de intersecção representam miRNAs. É necessário prever o alvo do gene regulador do miRNA para entender melhor a função do miRNA. Por meio da análise de OG, detectamos os termos de OG para prever a função desses genes alvo. A figura 4E mostra todos os genes alvo de miRNA up‐regulados, bem como os 10 principais termos de OG com o maior desenvolvimento. Da mesma maneira, os genes alvo para miRNA com uma tendência a down‐regulação estão listados na figura 4F. De acordo com a análise de bioinformática, acredita‐se que a função do gene alvo previsto para up‐regulação do miRNA esteja concentrada principalmente na parte intracelular, processo metabólico de macromoléculas celulares e ligação a proteínas. A função do gene alvo na down‐regulação do miRNA é principalmente concentrada na ligação intracelular e no controle do processo metabólico da macromolécula. O gene mostrou ser um componente da via de sinalização KEGG. Por meio da análise de vias, foram selecionadas algumas vias de sinal intimamente relacionadas à lesão aguda da pele induzida pela radiação UV. De acordo com os resultados, os genes mais prevalentes dentre os genes de down‐regulação foram aqueles envolvidos no câncer, infecção pelo papilomavírus humano e no sistema de sinalização MAPK (ver fig. 4G‐H). A via de sinalização MAPK foi escolhida para um estudo mais aprofundado porque as duas primeiras vias apresentaram correlação mínima com a lesão aguda avaliada.
Predição da Bioinformática. (A‐B) Diagrama de Venn. (C) Análise de rede de miRNA up‐regulada. (D) Análise de rede de miRNA down‐regulada. Dados gerais de OG para componente celular, função molecular e processos biológicos dos genes alvo regulados pelos miRNAs up‐regulados (E) e miRNAs down‐regulados (F). Análise da via KEGG dos genes alvo regulados pelos miRNAs up‐regulados (G) e miRNAs down‐regulados (H).
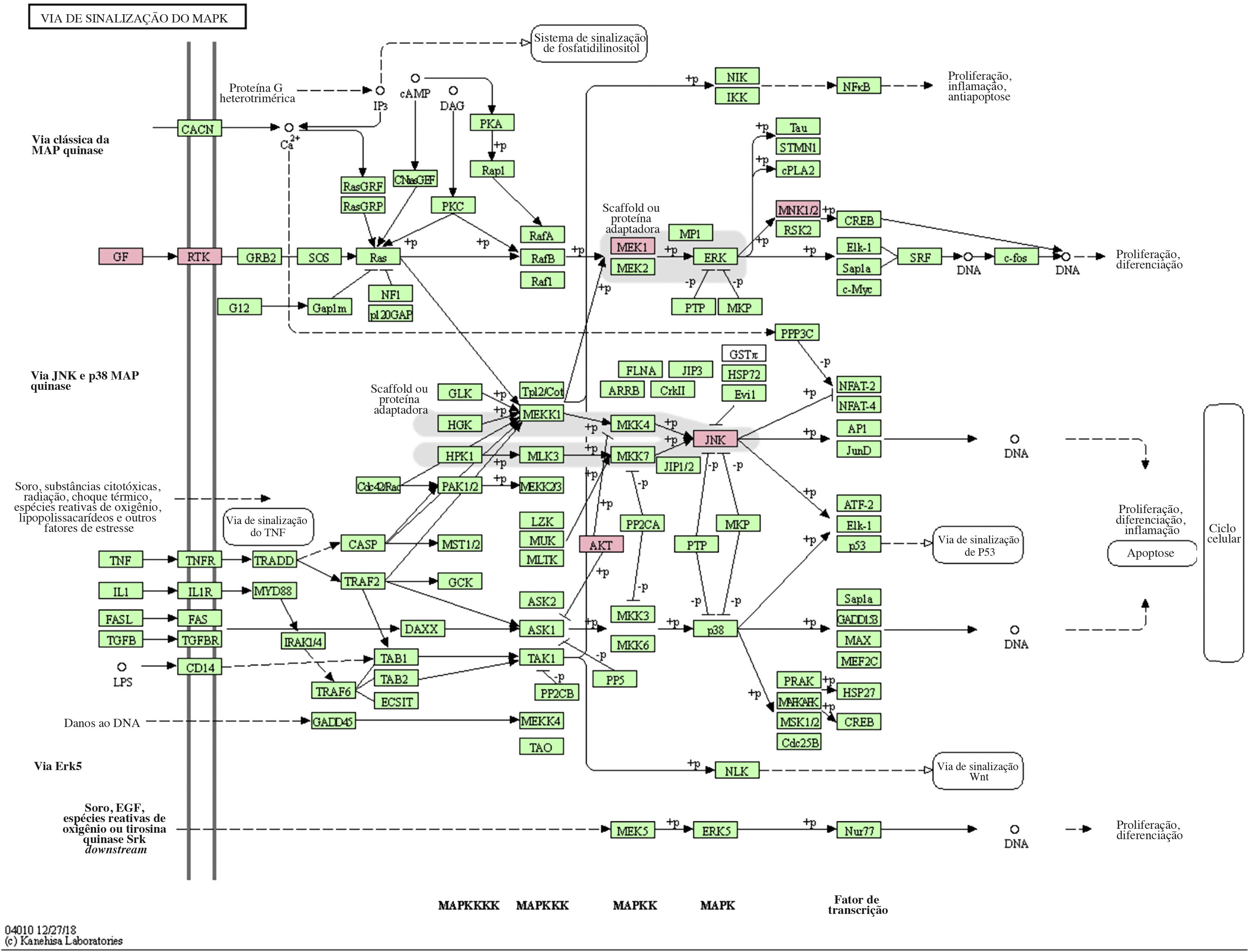
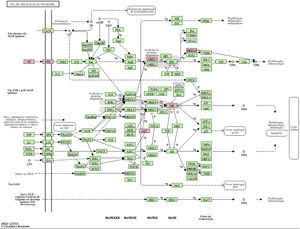
A via MAPK é uma via comum no corpo humano relacionada ao estresse e a outros processos fisiológicos complexos. Radiação, mudanças de pressão osmótica, fatores de crescimento e citocinas são todos exemplos de estímulos que podem ativar a via de transdução de sinal MAPK. Os cinco genes alvos importantes previstos pelo miR‐195a‐5p estavam todos localizados na via de sinalização MAPK, e eram AKT(AKT323797), RTK (INSR 16337), JNK (MAPK8 26419), MEK1(MAP2K1 26395), MNK1/2(MKNK1 17346), e GF (VEGFA 22339) (ver fig. 5). Foi sugerido que a radiação UV poderia induzir lesão cutânea aguda pela down‐regulação do miR‐195a‐5p e pela ativação da via de sinalização MAPK.
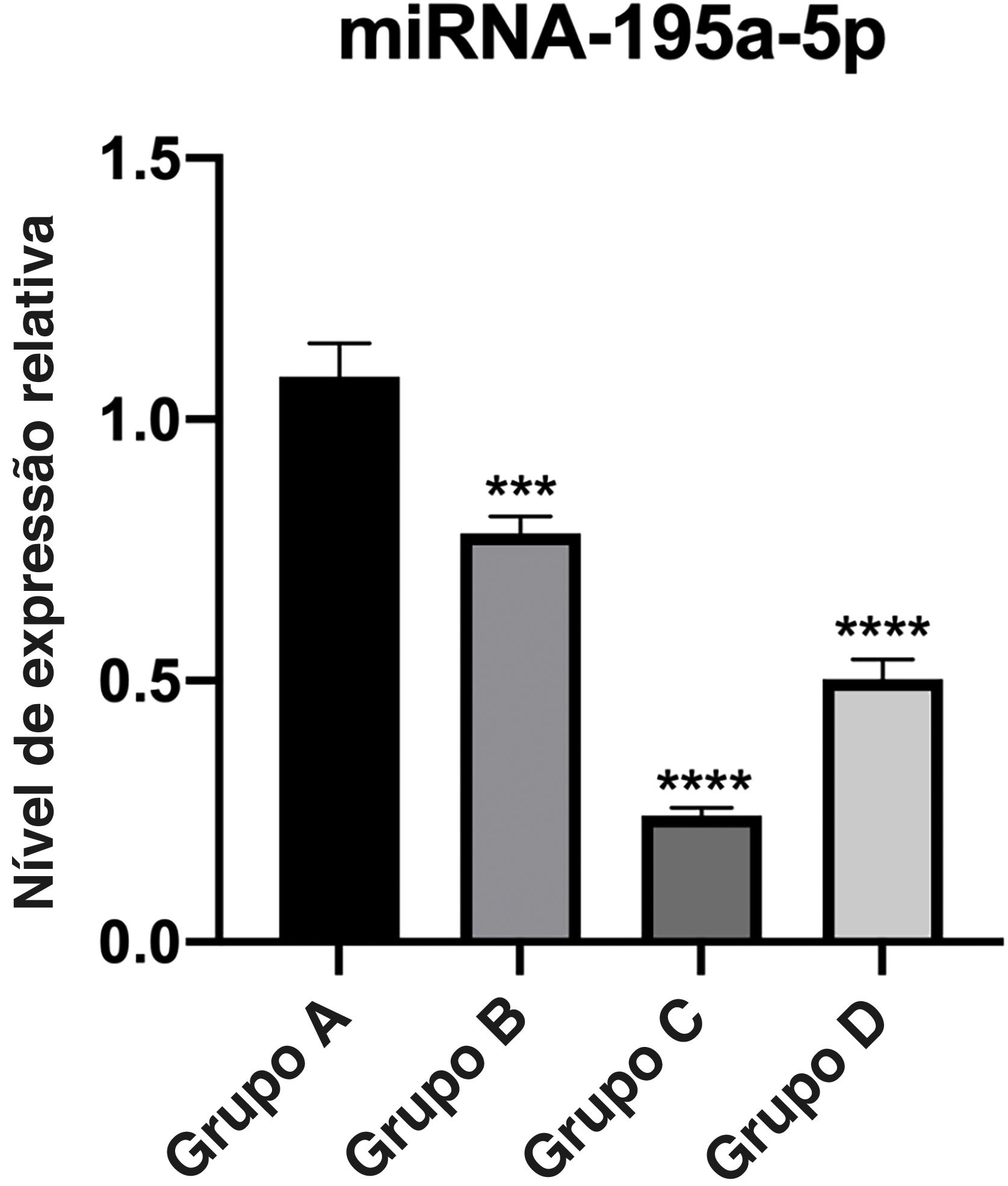
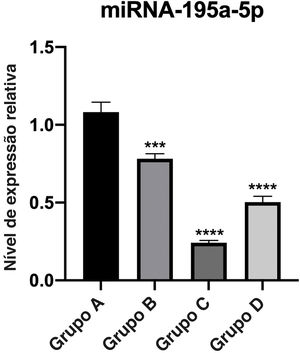
Verificação de RT‐qPCRA expressão relativa de miR‐195a‐5p em vários grupos experimentais foi analisada estatisticamente. Pode‐se observar na figura 6 que a expressão de miR‐195a‐5p nos três grupos experimentais mostrou uma tendência descendente após a radiação UV, o que estava de acordo com a predição de miR‐195a‐5p. Portanto, miR‐195a‐5p foi selecionado para um estudo mais aprofundado.
DiscussãoA radiação solar UV é um agente cancerígeno significativo no meio ambiente. O efeito clínico da radiação UV na pele normal pode ser agudo ou crônico. Os principais efeitos agudos causados pela UVB incluem eritema (queimadura solar), pigmentação (bronzeado), imunossupressão adquirida e aumento da imunidade inata.18,19 Mais de 80% do corpo humano sofre de fotoenvelhecimento da pele causado por radiação UV devido à perda da camada de ozônio causada pela poluição ambiental, exposição excessiva ao sol e outros fatores.20 Um dos focos de pesquisa é a prevenção e tratamento dos danos causados pela radiação UV. O grupo de estudo do presente trabalho tem uma base de pesquisa demonstrando que o fotoenvelhecimento induzido pela radiação UV está relacionado à via ERK/MAPK.21,22 Embora a UV seja um estimulador que pode ativar múltiplas cascatas de sinal, a ativação das mesmas e seu papel na via MAPK ainda não estão claros.
Foi relatado que muitos miRNAs estão intimamente associados a doenças relacionadas à pele, como o lúpus eritematoso sistêmico (LES), uma doença relacionada ao sistema imunológico.23 O miR‐377 pode ter como alvo e inibir diretamente o DNMT1, metilando assim o p53 e regulando o envelhecimento dos fibroblastos da pele humana.24 Outras pesquisas podem vir a identificar a relação entre o miRNA e o processo de fotoenvelhecimento da pele induzido por UVB; medidas preventivas eficientes, bem como biomarcadores para diagnóstico de doenças e novos alvos de ácidos nucleicos para terapia, seriam viáveis. De acordo com os resultados da sequência de miRNA, os miRNAs diferencialmente expressos foram avaliados. Havia sete miRNAs up‐regulados: mmu‐miR‐21a‐5p, mmu‐miR‐214‐5p, mmu‐miR‐126a‐3p, mmu‐miR‐142a‐5p, mmu‐miR‐7a‐5p, mmu‐miR‐455‐5p and mmu‐miR‐340‐5p. Dezesseis miRNAs que foram significativamente down‐regulados foram selecionados, incluindo mmu‐miR‐200a‐3p, mmu‐miR‐324‐5p, mmu‐miR‐133b‐3p, mmu‐miR‐133a‐3p, mmu‐miR‐101a‐3p, mmu‐miR‐101c, mmu‐miR‐195a‐5p, mmu‐let‐7b‐5p, mmu‐miR‐199b‐5p, mmu‐miR‐10a‐5p, mmu‐miR‐30b‐5p, mmu‐miR‐26a‐5p, mmu‐miR‐29a‐3p, mmu‐miR‐188b‐5p, mmu‐miR‐23a‐3p. Os resultados do sequenciamento precisam ser verificados por RT‐qPCR para confirmar a confiabilidade dos dados e resultados. Considerando a pesquisa anterior dos autores sobre muitos aspectos do mmu‐miR‐195a‐5p, o mmu‐miR‐195a‐5p foi selecionado para verificação. A expressão de mmu‐miR‐195a‐5p estava down‐regulada, o que foi consistente com os resultados do sequenciamento, sugerindo que os dados de sequenciamento do miRNA eram verdadeiros e confiáveis.
O miR‐195 é um dos principais membros da família micro‐15/16/195/424/497 que pode ser ativado em muitas doenças, como neoplasias, hemangioma e dor neuropática.25–27 O mecanismo de ação mediado pelo miR‐195 é extremamente complexo e envolve várias vias de transdução de sinal, como Rb‐E2F, PI3K/AKT, NF‐kB, MAPK/REK, Wnt/β‐catenina e assim por diante.28,29 Isso também confirma os achados do estudo dos autores sobre a via KEGG. A hipótese levantada é que o miR‐195a‐5p pode controlar a via MAPK. Os cinco genes alvos importantes previstos pelo miR‐195a‐5p estavam todos localizados na via de sinalização MAPK, e são Akt (akt323797), RTK (INSR 16337), JNK (MAPK8 26419), MEK1 (MAP2K1 26395), MNK1/2 (MKNK1 17346), GF (VEGFA 22339). Foi sugerido que a radiação ultravioleta poderia induzir o fotoenvelhecimento da pele através da down‐regulação do miR‐195a‐5p e ativando a via de sinalização MAPK.
Ainda há muitas limitações para este experimento. Um modelo celular de dano de ceratinócitos induzido por UV foi estabelecido, observou‐se uma série de alterações de expressão biológica relacionadas, verificando sua função e explorando ainda mais o papel específico do miR‐195a‐5p. A análise acima do mecanismo molecular downstream do miR‐195a‐5p é apenas especulativa, mas não foi confirmada. Este experimento apenas validou a predição da bioinformática a nível gênico. A transcrição e tradução de genes para expressão de proteínas é um processo complicado que deve ser validado ao nível da proteína utilizando técnicas como Western Blot ou imuno‐histoquímica. Em virtude das restrições de tempo e energia, a verificação ao nível da proteína específica, a exploração da via MAPK e o mecanismo molecular downstream do miR‐195a‐5p precisam ser estudados com mais profundidade no futuro.
ConclusãoO presente estudo estabeleceu uma relação entre miRNAs e radiação UVB. Esses achados podem ajudar a prevenir e retardar a ocorrência do envelhecimento da pele. Além disso, pode identificar biomarcadores para diagnóstico de doenças, bem como novos alvos para tratamento.
Suporte financeiroEste trabalho recebeu suporte através de uma bolsa do National Major Science and Technology Projects of China (2018ZX10303502) e Natural Science Foundation of China (n° 82002065).
Contribuição dos autoresYuan Fang: Aprovação da versão final do manuscrito; obtenção, análise e interpretação dos dados; participação efetiva na orientação da pesquisa; participação intelectual em conduta propedêutica e/ou terapêutica de casos estudados; revisão crítica do manuscrito; elaboração e redação do manuscrito; análise estatística; concepção e planejamento do estudo.
Lei Chen: Aprovação da versão final do manuscrito; revisão crítica do manuscrito.
Xin Wang: Aprovação da versão final do manuscrito; revisão crítica do manuscrito.
Xu Li: Aprovação da versão final do manuscrito; obtenção, análise e interpretação dos dados; participação efetiva na orientação da pesquisa; participação intelectual em conduta propedêutica e/ou terapêutica de casos estudados.
Wu Xiong: Aprovação da versão final do manuscrito; revisão crítica do manuscrito.
Xi Zhang: Aprovação da versão final do manuscrito; revisão crítica do manuscrito.
Yufang Zhang: Aprovação da versão final do manuscrito; participação efetiva na orientação da pesquisa; revisão crítica do manuscrito.
Lu Han: Aprovação da versão final do manuscrito; revisão crítica do manuscrito.
Ke Cao: Aprovação da versão final do manuscrito; revisão crítica do manuscrito.
Xiang Chen: Aprovação da versão final do manuscrito; revisão crítica do manuscrito.
Haibo Li: Aprovação da versão final do manuscrito; análise estatística; revisão crítica do manuscrito.
Jianda Zhou: Aprovação da versão final do manuscrito; participação efetiva na orientação da pesquisa; revisão crítica do manuscrito.
Conflito de interessesNenhum.
Agradecemos a todos os membros da equipe de Jianda Zhou pelo apoio especial. Agradecemos a ajuda do Departamento de Cirurgia Plástica e Reconstrutiva do Shanghai Ninth People's Hospital.
Como citar este artigo: Fang Y, Chen L, Wang X, Li X, Xiong W, Zhang X, et al. UVB irradiation differential regulate miRNAs expression in skin photoaging. An Bras Dermatol. 2022;97:458–66.
Trabalho realizado no Departamento de Cirurgia Plástica, The Third Xiangya Hospital, Central South University, Changsha, Hunan, China.