A leishmaniose é causada por um protozoário intracelular do gênero Leishmania. Lectina ligante de manose (MBL) é uma proteína do complemento do soro e reconhece antígenos lipoproteicos nos protozoários e na membrana plasmática da bactéria. Variantes de nucleotídeos na região promotora e éxon 1 do gene MBL podem influenciar sua expressão ou alterar sua estrutura molecular.
ObjetivoAvaliar por meio de revisão sistemática estudos de caso‐controle de associação genética de variantes no gene MBL2 e o risco de desenvolver leishmaniose.
MétodosEsta revisão pesquisou os bancos de dados PubMed, Science Direct, Cochrane Library, Scopus e Lilacs para publicações de caso‐controle com seis polimorfismos no gene lectina ligante de manose. Foi usada a estratégia: P=pacientes sob risco de leishmaniose; I=presença de polimorfismos; C=ausência de polimorfismos; O=ocorrência de leishmaniose. Quatro estudos de caso/controle consistindo em 791 pacientes com leishmaniose e 967 indivíduos saudáveis (controle) estão incluídos nesta metanálise. A associação de variantes no gene lectina ligante de manose e leishmaniose sob o modelo genético alélico, ‐550 (H vs. L), ‐221 (X vs. Y), +4 (Q vs. P), CD52 (A vs. D), CD54 (A vs. B), CD57 (A vs. C) e genótipo A/O (A vs. O) foi avaliado. Registro Internacional Prospectivo de Revisões Sistemáticas (PROSPERO): CRD42020201755.
ResultadosOs resultados da metanálise para qualquer modelo genético alélico não mostraram associação significativa para as variantes dentro do promotor, a região não traduzida e o éxon 1, bem como para o alelo selvagem A e alelo mutante O com a leishmaniose.
Limitações do estudoDeve‐se ter cautela ao interpretar esses resultados, pois se baseiam em poucos estudos, os quais apresentam resultados divergentes quando analisados separadamente.
ConclusõesEsta metanálise não mostrou associação significativa entre os polimorfismos rs11003125, rs7096206, rs7095891, rs5030737, rs1800450 e rs1800451 do gene lectina ligante de manose e leishmaniose em qualquer avaliação alélica e heterogênea.
A leishmaniose, uma doença transmitida através da picada da fêmea de um inseto hematófago da família dos flebótomos, causada por protozoários parasitas intracelulares pertencentes ao gênero Leishmania.1 A leishmaniose exibe uma variedade de características clínicas, como leishmaniose cutânea (LC), leishmaniose mucocutânea (LM) e leishmaniose visceral (LV).2 Doze milhões de pessoas em 98 países são vítimas de leishmaniose. Aproximadamente, 1,5 a 2,0 milhões e 500.000 novos casos de CL e LV, respectivamente, são observados anualmente. A leishmaniose causa cerca de 40 mil mortes por ano3 e é influenciada por diversos fatores, como origem genética do hospedeiro, aspectos nutricionais, Leishmania spp., meio ambiente e aspecto imunológico.4 Um estudo recente relatou que as variações genéticas do hospedeiro podem desempenhar papel fundamental na suscetibilidade à leishmaniose.5 Muitos genes foram investigados, evidenciando forte relação entre SNPs e risco de desenvolver leishmaniose, incluindo interferon‐gama (IFN‐G)6 e interleucina‐6 (IL‐6).7
A lectina de ligação à manose (MBL) é um receptor de reconhecimento de patógenos (PRR) e desempenha papel crítico na imunidade do hospedeiro. A MBL leva à ativação do sistema complemento.8,9 Essa proteína oligomérica consiste em subunidades estruturais formadas por três polipeptídios idênticos de 32 kD, cada um contendo uma ligação cruzada com a região N‐terminal da cisteína, colágeno ligado à região do pescoço e uma região do domínio C‐terminal que reconhece carboidratos em microrganismos.10
Por meio de múltiplos domínios da lectina, carboidratos como manose (carboidratos de seis carbonos) são encontrados na superfície de vários patógenos, incluindo Trypanosoma cruzi,11Plasmodium falciparum12 e Mycobacterium tuberculosis.13 Após o reconhecimento dessas moléculas pela lectina, as serina proteases são ativadas para facilitar a opsonização (fagocitose) pelos macrófagos e a lise da superfície do microrganismo.14 A forma infecciosa da leishmania (promastigota) é caracterizada pela presença de lipofosfoglicanos (GLP) e outras moléculas como a manose.15,16 Esses componentes atuam como padrões moleculares associados a patógenos (PAMPs) que são reconhecidos pelos componentes do complemento.17,18
O gene MBL2 está localizado no cromossomo 10 (10q11.2‐q21).19 Nesse gene, vários SNPs foram identificados, os quais são conhecidos por seu efeito funcional que influencia o desenvolvimento de doenças infecciosas.20–22 Os SNPs funcionais, localizados na região do promotor, como o SNP ‐550 H/L (substituição de G> C, rs11003125), ‐221 X/Y (substituição de C> G, rs7096206) e +4 Q/P (A substituição de C> T, rs7095891) localizada na região não traduzida, pode regular a taxa de transcrição do gene.23 No primeiro éxon existem três SNPs localizados no códon 52 CGT> TGT (rs5030737), códon 54 GGC> GAC (rs1800450) e códon 57 GGA> GAA (rs1800451), correspondendo a alterações de aminoácidos entre arginina para cisteína (Arg52Cys, alelo D), glicina para ácido aspártico (Gly54Asp, alelo B) e glicina para ácido glutâmico (Gly57Glu, alelo C) na região colágena da cadeia polipeptídica, respectivamente.24 Esses três polimorfismos formam o sistema AO, no qual o alelo selvagem é descrito como o alelo A, e o alelo O como um mutante. O genótipo A/O está correlacionado com baixos níveis da proteína e indetectável para o genótipo O/O.25
Resultados conflitantes são observados entre as variantes do gene MBL2 e a suscetibilidade à leishmaniose. Variantes, caracterizando altos níveis da proteína, foram associadas à suscetibilidade à LV na África,26 no nordeste do Brasil27 e na Índia.28 No entanto, um estudo realizado em indivíduos com CL no norte do Amazonas mostrou que todos os polimorfismos relacionados a baixos níveis de MBL tiveram uma forte associação com a susceptibilidade.29
Alguns estudos foram conduzidos anteriormente para avaliar os efeitos dos polimorfismos do gene MBL2 no progresso da infecção em leishmaniose, com resultados contraditórios, em virtude do pequeno tamanho de amostra, que carece de poder adequado para detectar os efeitos dos polimorfismos do gene MBL2 na leishmaniose.
Até o momento, nenhuma revisão sistemática foi realizada com variantes do gene MBL2 e leishmaniose. O uso da metanálise como ferramenta estatística que explora os fatores de risco associados a diferentes doenças genéticas pode fornecer uma conclusão confiável. Esta revisão sistemática incluiu estudos de caso‐controle de associação genética de variantes (rs11003125, rs7096206, rs7095891, rs5030737, rs1800450 e rs1800451) no gene MBL2 e risco de desenvolver leishmaniose. Esta revisão sistemática está no registro internacional prospectivo de revisões sistemáticas (PROSPERO): CRD42020201755.
Materiais e métodosPesquisa de banco de dadosEsta revisão sistemática foi conduzida de acordo com as recomendações do protocolo PRISMA (Preferred Reporting Items for Systematic reviews and Meta‐Analyzes).30 PubMed, Science Direct, Cochrane Library, Scopus e Lilacs foram pesquisados até dezembro de 2019 por três revisores independentes, sem restrição de idioma ou tempo.
Foi usada a estratégia: P=pacientes sob risco de leishmaniose; I=presença de polimorfismos; C=ausência de polimorfismos; O=ocorrência de leishmaniose. Os seguintes termos de pesquisa foram usados: (“Leishmaniasis” OR “Cutaneous Leishmaniasis” OR “Visceral Leishmaniasis” OR “Leishmania Infection” OR “Leishmania Infections”) AND (“Mannose‐binding Lectin” OR “Mannose‐binding Lectin 2” OR “MBL” OR “MBL2”) AND (“Polymorphism” OR “Polymorphisms” OR “Single Nucleotide Polymorphism” OR “Single Nucleotide Polymorphisms”). As referências citadas em artigos elegíveis foram pesquisadas manualmente para identificar publicações adicionais. A aprovação ética e o consentimento informado não foram necessários, uma vez que este estudo foi baseado em estudos publicados anteriormente e não teve contato direto com o paciente ou influências no atendimento ao paciente.
Seleção de estudosDois pesquisadores avaliaram independentemente todos os resultados da pesquisa. Os critérios de inclusão foram os seguintes: 1) estudo caso‐controle, 2) rs11003125 (‐550), rs7096206 (‐221), rs7095891 (+4), rs5030737 (códon 52), rs1800450 (CD54) e rs1800451 (CD57) polimorfismos, 3) estudos com dados de genotipagem suficientemente disponíveis para o cálculo dos Odds Ratios (OR) com Intervalos de Confiança de 95% (95% IC) e 4) o Equilíbrio de Hardy‐Weinberg (HWE). Os critérios de exclusão foram: 1) estudo não caso‐controle, 2) relatos de caso, 3) revisões, 4) estudos em animais, 5) editoriais, 6) estudos sem dados disponíveis, 7) estudos com metanálise, 8) outros polimorfismos e 9) dados duplicados. Posteriormente, todos os artigos selecionados foram checados por um terceiro pesquisador, que resolveu as divergências.
Extração de dadosDois pesquisadores extraíram independentemente os seguintes dados dos estudos incluídos: ano de publicação, primeiro autor, região do estudo, grupo étnico, forma clínica, número de amostras, idade e SNPs estudados. As divergências entre os pesquisadores foram discutidas e resolvidas pela consulta a um terceiro pesquisador.
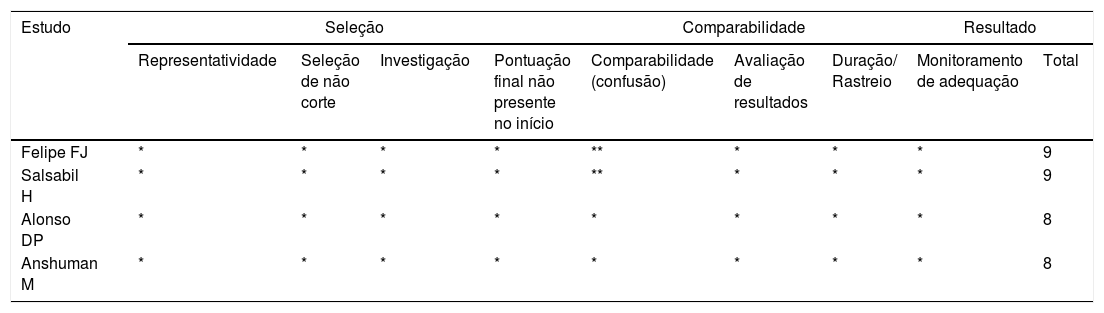
Avaliação de pontuação de qualidadeA escala Newcastle‐Ottawa (tabela 1) foi usada para avaliar a qualidade dos estudos elegíveis. Usando esse sistema, cada estudo incluído foi submetido a três julgamentos: 1) seleção de grupos de estudo; 2) comparabilidade dos grupos e 3) resultado de interesse (caso‐controle). Três pesquisadores calcularam independentemente a pontuação de cada publicação. Os escores variaram de 0 a 9. Os estudos com escore> 6 foram considerados de alta qualidade, enquanto escore < 6, de baixa qualidade. As divergências entre os pesquisadores foram discutidas em grupo e resolvidas em consenso.
Escala de Newcastle‐Ottawa dos estudos incluídos
| Estudo | Seleção | Comparabilidade | Resultado | ||||||
|---|---|---|---|---|---|---|---|---|---|
| Representatividade | Seleção de não corte | Investigação | Pontuação final não presente no início | Comparabilidade (confusão) | Avaliação de resultados | Duração/ Rastreio | Monitoramento de adequação | Total | |
| Felipe FJ | * | * | * | * | ** | * | * | * | 9 |
| Salsabil H | * | * | * | * | ** | * | * | * | 9 |
| Alonso DP | * | * | * | * | * | * | * | * | 8 |
| Anshuman M | * | * | * | * | * | * | * | * | 8 |
Cada item foi pontuado com pontuação máxima de um ponto (um *), com exceção da comparabilidade, que permitia dois pontos.
A metanálise avaliou a associação do gene MBL2 e leishmaniose sob o modelo genético alélico, ‐550 (H vs. L), ‐221 (X vs. Y), +4 (Q vs. P), CD52 (A vs. D), CD54 (A vs. B), CD57 (A vs. C) e genótipo A/O (A vs. O). O I2 foi utilizado para avaliar a heterogeneidade entre os estudos onde os valores 25%, 50% e 75% corresponderam a baixa, moderada e alta heterogeneidade, respectivamente. O modelo fixo foi usado quando I2 <50%, e o modelo aleatório foi usado quando I2> 50%. OR agrupados foram calculados usando o Mantel‐Haenszel, e a significância estatística de OR foi determinada usando estatística Z. Em ambos os modelos, o valor p=0,005 foi considerado estatisticamente significativo. O software RStudio (www.rstudio.com/products/rstudio/), versão 1.3.1 para Windows foi usado como programa estatístico para o estudo. Pacotes (“tidyverse”), (“meta”), (“metafor”).
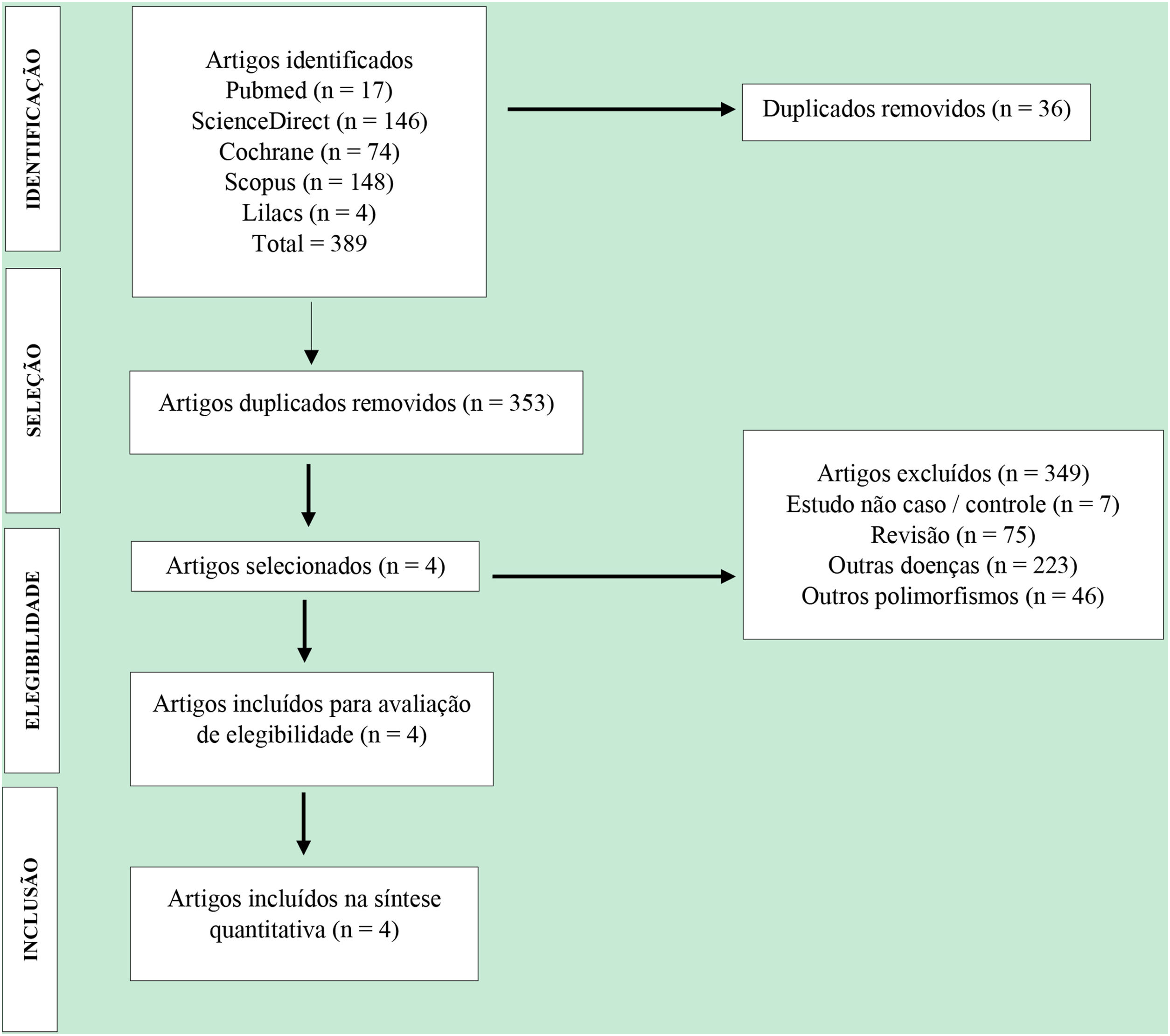
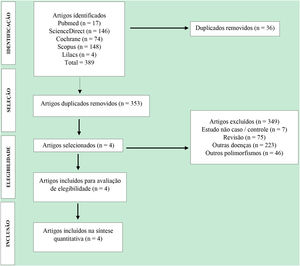
ResultadosCaracterísticas dos estudos incluídosForam pulicados 389 artigos identificados usando as bases de dados da literatura científica (fig. 1). Dentre os artigos selecionados, 35 foram removidos por duplicação, 349 artigos foram excluídos por não atenderem aos critérios de inclusão. Por fim, apenas quatro artigos,26–29 respeitando os critérios obrigatórios, foram incluídos na metanálise. Esses estudos foram publicados em inglês entre 2007 e 2015 (tabela 2).
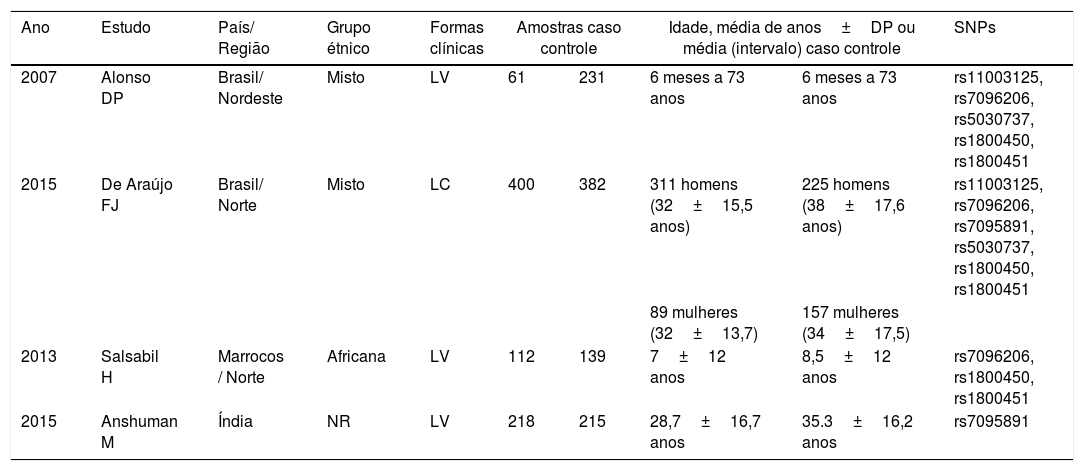
Leishmaniose. Características dos estudos incluídos na revisão sistemática
| Ano | Estudo | País/ Região | Grupo étnico | Formas clínicas | Amostras caso controle | Idade, média de anos±DP ou média (intervalo) caso controle | SNPs | ||
|---|---|---|---|---|---|---|---|---|---|
| 2007 | Alonso DP | Brasil/ Nordeste | Misto | LV | 61 | 231 | 6 meses a 73 anos | 6 meses a 73 anos | rs11003125, rs7096206, rs5030737, rs1800450, rs1800451 |
| 2015 | De Araújo FJ | Brasil/ Norte | Misto | LC | 400 | 382 | 311 homens (32±15,5 anos) | 225 homens (38±17,6 anos) | rs11003125, rs7096206, rs7095891, rs5030737, rs1800450, rs1800451 |
| 89 mulheres (32±13,7) | 157 mulheres (34±17,5) | ||||||||
| 2013 | Salsabil H | Marrocos / Norte | Africana | LV | 112 | 139 | 7±12 anos | 8,5±12 anos | rs7096206, rs1800450, rs1800451 |
| 2015 | Anshuman M | Índia | NR | LV | 218 | 215 | 28,7±16,7 anos | 35.3±16,2 anos | rs7095891 |
NR, não relatado.
Um estudo foi realizado em crianças africanas.26 Outros dois estudos foram de populações mistas do Nordeste do Brasil, compostas por 21% de europeus, 31% de africanos e 48% de ancestrais nativos americanos,27 e do norte do Brasil (estado do Amazonas), com uma população mista de 10% de africanos, 40% ancestrais europeus e 50% dos nativos americanos.29 O quarto estudo foi realizado na Índia, mas não especificou a etnia dos indivíduos estudados.28 Três estudos analisaram pacientes com LV26–28 e um analisou pacientes com CL.29Leishmania spp. identificadas foram: L. chagasi,27L. infantum,26L. guyanensis29 e L. donovani.28 Um estudo analisou todos os polimorfismos alvo desta metanálise,29 enquanto outro apenas cinco SNPs (rs11003125, rs7096206, rs5030737, rs1800450 e rs1800451).27 Um estudo analisou três SNPs (rs7096206, rs1800450 e rs1800451),26 e o estudo restante apenas um SNP (rs7095891).28 Dois estudos forneceram dados suficientes para realizar a análise do sistema A/O.27,29 Um total de 1.758 pessoas participaram desses estudos (791 pacientes e 967 controles). De acordo com a escala de Newcastle‐Ottawa, dois estudos obtiveram 9 pontos e dois com 8 pontos (tabela 1). A frequência dos genótipos e alelos estão organizados na tabela 3.
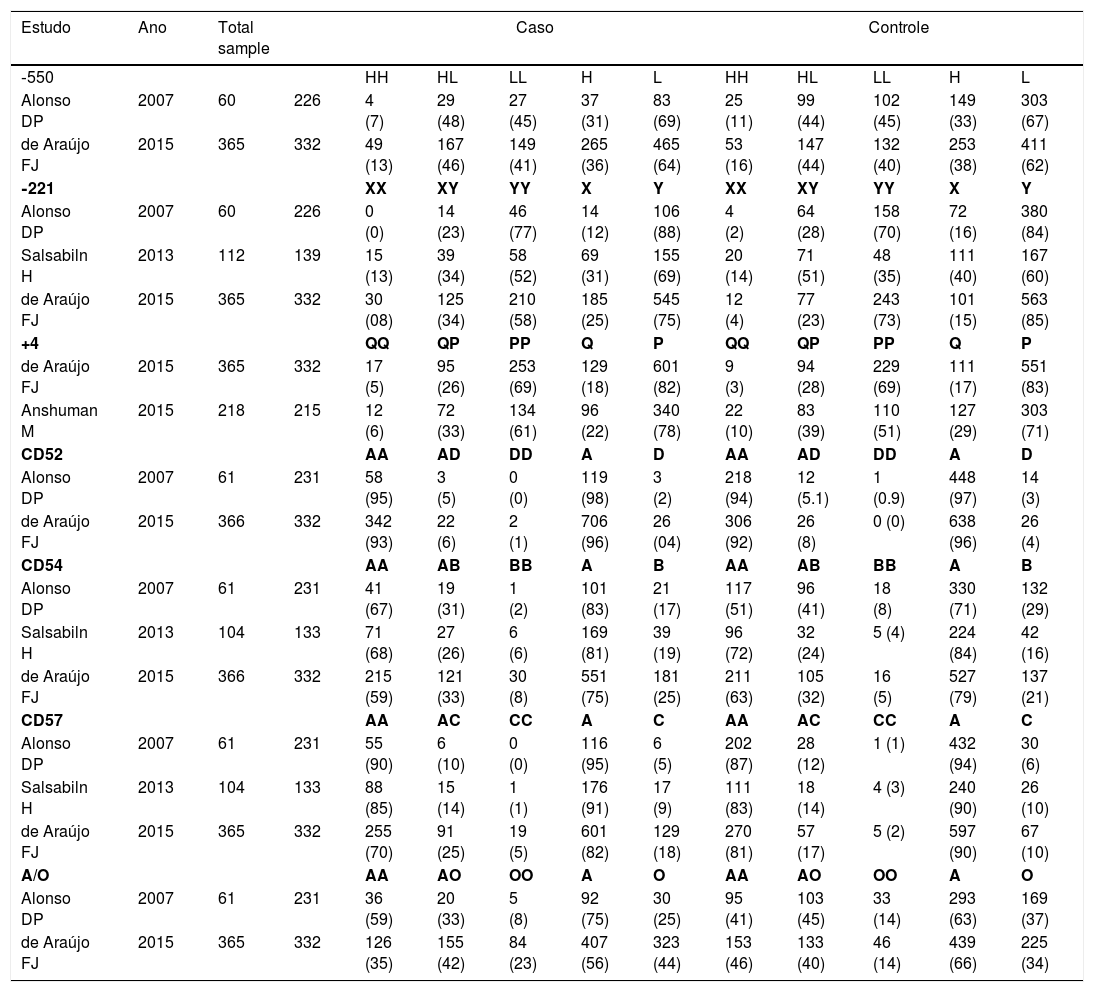
Modelo genético alélico adotado na metanálise para avaliar a associação de polimorfismos do gene MBL2 e leishmaniose
| Estudo | Ano | Total sample | Caso | Controle | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ‐550 | HH | HL | LL | H | L | HH | HL | LL | H | L | |||
| Alonso DP | 2007 | 60 | 226 | 4 (7) | 29 (48) | 27 (45) | 37 (31) | 83 (69) | 25 (11) | 99 (44) | 102 (45) | 149 (33) | 303 (67) |
| de Araújo FJ | 2015 | 365 | 332 | 49 (13) | 167 (46) | 149 (41) | 265 (36) | 465 (64) | 53 (16) | 147 (44) | 132 (40) | 253 (38) | 411 (62) |
| ‐221 | XX | XY | YY | X | Y | XX | XY | YY | X | Y | |||
| Alonso DP | 2007 | 60 | 226 | 0 (0) | 14 (23) | 46 (77) | 14 (12) | 106 (88) | 4 (2) | 64 (28) | 158 (70) | 72 (16) | 380 (84) |
| Salsabiln H | 2013 | 112 | 139 | 15 (13) | 39 (34) | 58 (52) | 69 (31) | 155 (69) | 20 (14) | 71 (51) | 48 (35) | 111 (40) | 167 (60) |
| de Araújo FJ | 2015 | 365 | 332 | 30 (08) | 125 (34) | 210 (58) | 185 (25) | 545 (75) | 12 (4) | 77 (23) | 243 (73) | 101 (15) | 563 (85) |
| +4 | QP | PP | Q | P | QP | PP | Q | P | |||||
| de Araújo FJ | 2015 | 365 | 332 | 17 (5) | 95 (26) | 253 (69) | 129 (18) | 601 (82) | 9 (3) | 94 (28) | 229 (69) | 111 (17) | 551 (83) |
| Anshuman M | 2015 | 218 | 215 | 12 (6) | 72 (33) | 134 (61) | 96 (22) | 340 (78) | 22 (10) | 83 (39) | 110 (51) | 127 (29) | 303 (71) |
| CD52 | AA | AD | DD | A | D | AA | AD | DD | A | D | |||
| Alonso DP | 2007 | 61 | 231 | 58 (95) | 3 (5) | 0 (0) | 119 (98) | 3 (2) | 218 (94) | 12 (5.1) | 1 (0.9) | 448 (97) | 14 (3) |
| de Araújo FJ | 2015 | 366 | 332 | 342 (93) | 22 (6) | 2 (1) | 706 (96) | 26 (04) | 306 (92) | 26 (8) | 0 (0) | 638 (96) | 26 (4) |
| CD54 | AA | AB | BB | A | B | AA | AB | BB | A | B | |||
| Alonso DP | 2007 | 61 | 231 | 41 (67) | 19 (31) | 1 (2) | 101 (83) | 21 (17) | 117 (51) | 96 (41) | 18 (8) | 330 (71) | 132 (29) |
| Salsabiln H | 2013 | 104 | 133 | 71 (68) | 27 (26) | 6 (6) | 169 (81) | 39 (19) | 96 (72) | 32 (24) | 5 (4) | 224 (84) | 42 (16) |
| de Araújo FJ | 2015 | 366 | 332 | 215 (59) | 121 (33) | 30 (8) | 551 (75) | 181 (25) | 211 (63) | 105 (32) | 16 (5) | 527 (79) | 137 (21) |
| CD57 | AA | AC | CC | A | C | AA | AC | CC | A | C | |||
| Alonso DP | 2007 | 61 | 231 | 55 (90) | 6 (10) | 0 (0) | 116 (95) | 6 (5) | 202 (87) | 28 (12) | 1 (1) | 432 (94) | 30 (6) |
| Salsabiln H | 2013 | 104 | 133 | 88 (85) | 15 (14) | 1 (1) | 176 (91) | 17 (9) | 111 (83) | 18 (14) | 4 (3) | 240 (90) | 26 (10) |
| de Araújo FJ | 2015 | 365 | 332 | 255 (70) | 91 (25) | 19 (5) | 601 (82) | 129 (18) | 270 (81) | 57 (17) | 5 (2) | 597 (90) | 67 (10) |
| A/O | AA | AO | OO | A | O | AA | AO | OO | A | O | |||
| Alonso DP | 2007 | 61 | 231 | 36 (59) | 20 (33) | 5 (8) | 92 (75) | 30 (25) | 95 (41) | 103 (45) | 33 (14) | 293 (63) | 169 (37) |
| de Araújo FJ | 2015 | 365 | 332 | 126 (35) | 155 (42) | 84 (23) | 407 (56) | 323 (44) | 153 (46) | 133 (40) | 46 (14) | 439 (66) | 225 (34) |
Os resultados da metanálise são mostrados na figura 2. Nenhuma das análises para qualquer modelo genético de alelo para as duas variantes (‐550 e ‐221) no promotor e as variantes +4 na região não traduzida mostraram associação com suscetibilidade ou resistência à leishmaniose (o alelo ‐550H: OR=0,92; 95% IC=0,76‐1,12; p=0,93; I2=0% e alelo ‐550L: OR=1,08; 95% IC=0,89‐1,32; p=0,93; I2=0%), o alelo ‐221X: OR=0,98; 95% IC=0,45‐2,13; p=0,01; I2=91% e alelo ‐221Y: OR=1,02; 95% IC=0,47‐2,22; p=0,01; I2=91%) e o alelo Q +4: OR=0,85; 95% IC=0,54‐1,33; p=0,03; I2=79% e alelo P: OR=1,17; 95% IC=0,75‐1,84;p=0,03; I2=79%). Resultados semelhantes foram obtidos para variantes localizadas no exon 1 CD52 (alelo A do alelo: OR=1,13; 95% IC=0,68‐1,87; p=0,87; I2=0% e alelo D do alelo: OR=0,89; 95% IC=0,53‐1,47; p=0,87; I2=0%), CD54 (alelo A: OR=1,04; 95% IC=0,62‐1,75; p=0,01; I2=79% e alelo B: OR=0,96; 95% IC=0,57‐1,61; p=0,01; I2=79%) e CD57 (alelo A: OR=0,85; 95% IC=0,44‐1,62; p=0,03; I2=72% e alelo C: OR=1,18; 95% IC=0,62‐2,26; p=0,03; I2=72%). A presença do alelo A selvagem (alelo A: OR=1,05; 95% IC=0,39‐2,81; p=0,01; I2=94%) e alelo O mutante (alelo O: OR=0,95; 95% IC=0,36‐2,56; p=0,01; I2=94%) também não foram associados à suscetibilidade ou resistência.
DiscussãoA MBL reconhece a presença de manose na superfície dos patógenos para promover a opsonização e a ativação do sistema complemento.31 A MBL desempenha papel fundamental na resposta imune inata,32 destacando sua concentração sérica como requisito para a predisposição ao desenvolvimento de doenças infecciosas humanas.33,34 As variantes do gene MBL2 foram associadas a risco aumentado de infecção por protozoários.35,36 No entanto, poucos estudos investigaram variantes genéticas no gene MBL2 na infecção por leishmania.26–29 Três estudos sugeriram que as variantes correlacionadas com baixos níveis circulantes de MBL são protetoras para VL,26,28 enquanto um estudo mostrou suscetibilidade ao CL.29
Os resultados conflitantes gerados pela maioria dos estudos tiveram um poder estatístico fraco em virtude do pequeno tamanho da amostra incluída. Para esclarecer resultados contraditórios em estudos de associação genética, a metanálise oferece um método poderoso para sintetizar dados obtidos de estudos independentes.37 Para abordar as limitações dos estudos de caso‐controle, a presente metanálise foi realizada para fornecer evidências estatísticas da associação entre os polimorfismos do gene MBL2 e a suscetibilidade à leishmaniose com ORs agrupados. Até o momento, esta é a primeira metanálise que aborda a associação entre os polimorfismos descritos e a leishmaniose. Metanálises anteriores sugeriram uma associação de polimorfismos nos genes IL2RA (receptor alfa da interleucina 2)38 e SLC11A1 (família transportadora de soluto 11 member a1)39 com os aspectos clínicos da leishmaniose.
Neste estudo, os dados de quatro artigos foram analisados de acordo com os alelos de baixa e alta produção da MBL. No entanto, as análises de metanálise não mostraram associação entre os alelos do gene MBL2 e a suscetibilidade à leishmaniose (fig. 1). Alta heterogenicidade foi observada para as variantes: ‐550 H/L (91%), +4 Q/P (79%), CD54 A/B (79%), CD57 A/C (72%) e A/O (94%). Isso pode ser explicado principalmente pelo fato de haver miscigenação étnica nos indivíduos dos estudos selecionados. Três estudos investigaram pacientes com VL,26–28 e um paciente investigado com LC.29 Em cada estudo, a espécie do agente etiológico foi diferente. É importante notar que o valor da heterogeneidade influencia o modelo estatístico adequado. Estudos com pequenos tamanhos de amostra podem mostrar resultados não confiáveis. Como consequência, o modelo aleatório deve ser sempre aplicado.40 Dentre os estudos selecionados, um analisou todos os seis polimorfismos alvo, os diplótipos, e também os haplótipos,29 com um número amostral elevado.
No entanto, deve‐se ter cautela na interpretação desses resultados, pois se baseiam em poucos estudos, os quais apresentam resultados divergentes quando analisados separadamente. Portanto, mais estudos são necessários para confirmar se as variantes que determinam os níveis séricos baixos são suscetíveis ou protetoras. Ressaltamos a importância do estudo de associação envolvendo marcadores genéticos na leishmaniose, para novos entendimentos sobre os mecanismos moleculares da doença. As variantes podem ser utilizadas como marcadores moleculares da predisposição do indivíduo a certos tipos de doenças ou como alvos terapêuticos no desenvolvimento de novos medicamentos.
ConclusãoEsta metanálise não mostrou associação significativa entre os polimorfismos rs11003125, rs7096206, rs7095891, rs5030737, rs1800450 e rs1800451 do gene MBL2 e leishmaniose.
Suporte financeiroNenhum.
Contribuição dos autoresWonei de Seixas Vital: Conceituação; análise formal; metodologia; supervisão; validação; redação – revisão e edição.
Felipe Jules de Araújo Santos: Conceituação; análise formal; metodologia; supervisão; validação; redação – revisão e edição.
Maurício Leandro Fernandes Gonçalves: Curadoria de dados; análise formal; metodologia; redação – rascunho original.
Claudia Dantas Comandolli Wyrepkowski: Curadoria de dados; análise formal; metodologia.
Rajendranath Ramasawmy: Curadoria de dados; análise formal; metodologia; redação; revisão final.
Silvania da Conceição Furtado: Conceituação; análise formal; metodologia; supervisão; validação; redação – revisão e edição.
Conflito de interessesNenhum.
Como citar este artigo: Vital WS, Santos FJA, Gonçalves MLF, Wyrepkowski CDC, Ramasawmy R, Furtado SC. Influence of the presence of mannose‐binding lectin polymorphisms on the occurrence of leishmaniasis: a systematic review and meta‐analysis. An Bras Dermatol. 2022;97:298–306.
Trabalho realizado na Universidade Federal do Amazonas, Manaus, AM, Brasil.