O vitiligo é caracterizado por resposta autoimune direcionada aos melanócitos, resultando na despigmentação da pele. Existem vários componentes genéticos envolvidos no desenvolvimento do vitiligo, dos quais vários polimorfismos genéticos são atualmente considerados fatores de risco. Por exemplo, as variantes do gene do antígeno 4 associado ao linfócito T citotóxico (CTLA4)+49A/G (rs231775) e CT60 (rs3087243) foram associadas à predisposição para doenças autoimunes em diferentes populações; entretanto, seu envolvimento no desenvolvimento do vitiligo permanece controverso.
ObjetivoAvaliar a associação entre vitiligo e as variantes do gene CTLA4+49A/G (rs231775) e CT60 (rs3087243) em população mexicana.
Métodos116 pacientes com vitiligo e 117 indivíduos controles do nordeste do México foram incluídos no estudo, com análise por PCR‐RFLP para determinar as variantes do gene CTLA4+49A/G (rs231775) e CT60 (rs3087243).
ResultadosNão foi observada diferença estatística para ambos os polimorfismos gênicos entre pacientes com vitiligo e controles (p> 0,05). Além disso, quando os parâmetros de atividade e história familiar de vitiligo, história pessoal de doenças autoimunes ou sexo foram analisados, não foram observadas diferenças (p> 0,05).
ConclusãoComo sugerido pela análise de uma população do nordeste mexicano, as variantes do gene CTLA4+49A/G (rs231775) e CT60 (rs3087243) não constituem fator de risco no desenvolvimento do vitiligo.
O vitiligo é um distúrbio autoimune caracterizado pela perda seletiva de melanócitos, que resulta na despigmentação da pele, e afeta cerca de 0,5% a 2% da população mundial, com prevalência global de aproximadamente 0,06% a 8,8%.1,2 O desenvolvimento do vitiligo tem forte componente genético, e vários polimorfismos genéticos têm sido associados à resposta autoimune e melanogênese tipicamente observadas nessa doença.2 Durante esse processo, os melanócitos são destruídos pelas células T, que constituem um dos principais mediadores da resposta autoimune, criando áreas de despigmentação.3 Um membro da superfamília de imunoglobulinas, o gene do antígeno 4 associado ao linfócito T citotóxico (CTLA4), encontrado no locus 2q33,4 codifica um receptor‐chave inibitório cuja função é a de um potente regulador negativo da resposta das células T durante a fase de priming da resposta imune.5 Portanto, o CTLA4 tem sido associado a várias doenças autoimunes, como doença autoimune da tireoide, doença de Graves, tireoidite de Hashimoto, diabetes tipo 1,6 psoríase,7 doença de Behçet8 e alopecia areata, embora a associação com esta última permaneça controversa.9
Duas variantes genéticas do CTLA4 foram associadas ao desenvolvimento do vitiligo:+49A/G (rs231775), uma variação missense do éxon 1 que leva à substituição de treonina por alanina no códon 17 (T17A); e CT60 A/G (rs3087243), localizado a 236 pb downstream de CTLA4 3’‐UTR.10 Entretanto, essas variantes e sua associação com o vitiligo variam amplamente e parecem ser fortemente dependentes da população analisada. 10–12
Assim, o presente estudo avaliou se as variantes do gene CTLA4+49 A/G e CT60 A/G estão associadas ao vitiligo em uma população mexicana.
Material e métodosAmostraPacientes com vitiligo e controles saudáveis foram recrutados do Departamento de Dermatologia do Hospital Universitário Dr. José Eleuterio Gonzalez, Universidad Autónoma de Nuevo León (UANL; Monterrey, México). Os indivíduos eram originários da região nordeste do México, incluindo os estados de Coahuila, Nuevo León, San Luis Potosí, Tamaulipas e Zacatecas. Um total de 116 pacientes com vitiligo (62 mulheres e 54 homens; média de idade de 43,06 ± 16,34 anos) e 117 controles saudáveis (75 mulheres e 42 homens; média de idade de 29,01±12,67 anos) foram incluídos neste estudo. Todos os pacientes foram avaliados por um dermatologista. A atividade do vitiligo foi determinada pelo intervalo de tempo entre a manifestação de novas áreas despigmentadas (vitiligo com estabilidade da lesão>1 ano) ou aumento das já existentes. O presente estudo foi aprovado pelo Comitê de Ética e Pesquisa do Hospital Universitário Dr. José Eleuterio Gonzalez – UANL (código DE08‐008). Todos os participantes forneceram seu consentimento informado.
Isolamento de DNAO DNA genômico foi isolado do sangue venoso periférico dos pacientes com vitiligo e controles. As amostras foram centrifugadas e a camada leucoplaquetária foi processada para isolamento do DNA seguindo o método de salting‐out – com o pellet de DNA ressuspenso em Tris‐EDTA (Ph 7,8) à concentração final de 0,1‐1,0μg/μL.
Genotipagem das variantes de CTLA4+49A/G (rs231775) e CT60 (rs3087243)A frequência alélica das variantes do gene CTLA4 rs231775 e rs3087243 foi caracterizada por PCR‐RFLP em termociclador MJ Mini PTC1148 (Bio‐Rad, Hercules; CA, EUA). Os primers de oligonucleotídeos para CTLA4 rs231775 (5’‐CCA CGG CTT CCT TTC TCG TA‐3’ e 5’‐AGT CTC ACT CAC CTT TGC AG‐3’) e CTLA4 rs3087243 (5’‐ATG AGT CAG CTT TGC ACC AGC CAT TAC‐3’ e 5’‐GAG GTG AAG AAC CTG TGT TAA ACA GCA TG‐3’) foram obtidos com IDT (IA; EUA).
De acordo com um protocolo publicado anteriormente, as enzimas BbvI e NlaIII (New England Biolabs, MA; EUA) foram usadas na análise de restrição.13 Os fragmentos do amplicon foram resolvidos por eletroforese em gel de agarose a 2,5%, corados com brometo de etídio e observados em um transiluminador UVP modelo 2UV High Performance (Upland, CA, EUA).
Análise estatísticaO tamanho da amostra foi definido com base na incidência relatada de vitiligo no México (4%).2 Considerando um poder estatístico de 99,0% (Z=2,33), um mínimo de 84 indivíduos é suficiente para análise genética precisa. O software SPSS v21.0 para Windows (IBM, IL; EUA) e o programa estatístico Epi‐INFO™ 7 (CDC, EUA) foram utilizados na análise estatística. O teste de equilíbrio de Hardy‐Weinberg foi obtido para os alelos usando teste de ajuste, enquanto a dependência genotípica entre pacientes e controles foi determinada com teste de χ2. O OR foi calculado a partir de tabelas de contingência 2×2. Um valor de p <0,05 foi considerado significante para todos os testes.
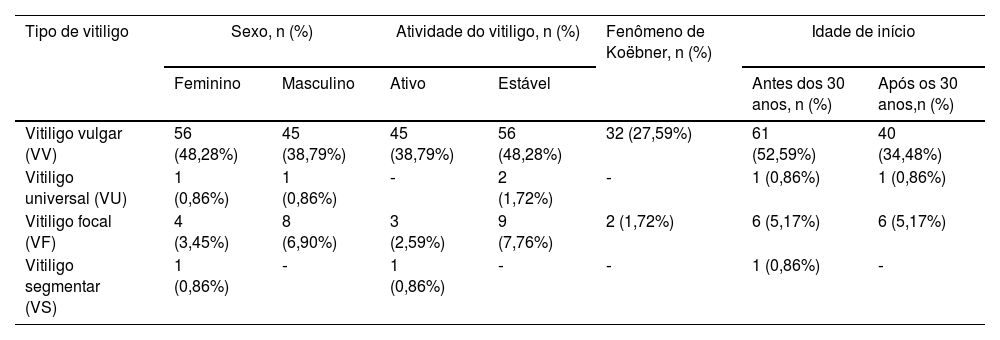
ResultadosParâmetros clínicosOs 116 pacientes com vitiligo incluídos neste estudo foram classificados em quatro categorias de acordo com a apresentação clínica da doença: 101 (87,07%) vitiligo vulgar (VV), 12 (10,35%) vitiligo focal (VF), 2 (1,72%) vitiligo universal (VU) e 1 (0,86%) vitiligo segmentar (VS; tabela 1). A idade de início, o aparecimento das lesões cutâneas após trauma (fenômeno de Koëbner) e as comorbidades comuns são apresentadas nas tabelas 1 e 2. Aproximadamente 47% dos pacientes tinham pelo menos um familiar com vitiligo; a idade de início antecedeu os 30 anos para 58,48% dos pacientes.
Parâmetros clínicos dos pacientes com vitiligo
| Tipo de vitiligo | Sexo, n (%) | Atividade do vitiligo, n (%) | Fenômeno de Koëbner, n (%) | Idade de início | |||
|---|---|---|---|---|---|---|---|
| Feminino | Masculino | Ativo | Estável | Antes dos 30 anos, n (%) | Após os 30 anos,n (%) | ||
| Vitiligo vulgar (VV) | 56 (48,28%) | 45 (38,79%) | 45 (38,79%) | 56 (48,28%) | 32 (27,59%) | 61 (52,59%) | 40 (34,48%) |
| Vitiligo universal (VU) | 1 (0,86%) | 1 (0,86%) | ‐ | 2 (1,72%) | ‐ | 1 (0,86%) | 1 (0,86%) |
| Vitiligo focal (VF) | 4 (3,45%) | 8 (6,90%) | 3 (2,59%) | 9 (7,76%) | 2 (1,72%) | 6 (5,17%) | 6 (5,17%) |
| Vitiligo segmentar (VS) | 1 (0,86%) | ‐ | 1 (0,86%) | ‐ | ‐ | 1 (0,86%) | ‐ |
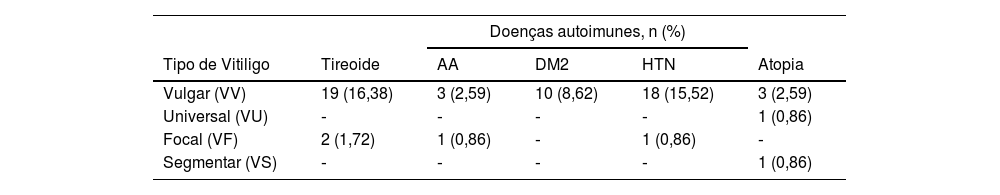
Dos pacientes com vitiligo, 21 (18,1%) tinham história pessoal de alterações tireoidianas (tabela 2); hipotireoidismo foi a mais prevalente (8,6%). Entretanto, diabetes mellitus tipo 2 (DM2, 8,62%) e hipertensão arterial (HTN, 16,38%) foram as comorbidades mais encontradas nesses pacientes.
Doenças autoimunes associadas ao vitiligo
| Doenças autoimunes, n (%) | |||||
|---|---|---|---|---|---|
| Tipo de Vitiligo | Tireoide | AA | DM2 | HTN | Atopia |
| Vulgar (VV) | 19 (16,38) | 3 (2,59) | 10 (8,62) | 18 (15,52) | 3 (2,59) |
| Universal (VU) | ‐ | ‐ | ‐ | ‐ | 1 (0,86) |
| Focal (VF) | 2 (1,72) | 1 (0,86) | ‐ | 1 (0,86) | ‐ |
| Segmentar (VS) | ‐ | ‐ | ‐ | ‐ | 1 (0,86) |
AA, alopecia areata; DM2, diabetes mellitus tipo 2; HTN, hipertensão arterial; VV, vitiligo vulgar; VU, vitiligo universal; VF, vitiligo focal; VS, vitiligo segmentar.
O estudo investigou se as variantes do gene CTLA4+49A/G (rs231775) e CT60 (rs3087243) estavam associadas ao vitiligo em amostra de pacientes e controles saudáveis mexicanos. Nenhum desvio do equilíbrio de Hardy‐Weinberg foi detectado para qualquer um dos polimorfismos de CTLA4 avaliados ([rs231775: pacientes com vitiligo Pearson 0,889, razão de verossimilhança 0,889 e teste exato de Fisher 1; controles Pearson 0,885, razão de verossimilhança 0,885 e teste exato de Fisher 1], [rs3087243: pacientes com vitiligo Pearson 0,837, razão de verossimilhança 0,837 e teste exato de Fisher 1; controles Pearson 0,721, razão de verossimilhança 0,721 e teste exato de Fisher 0,85]).
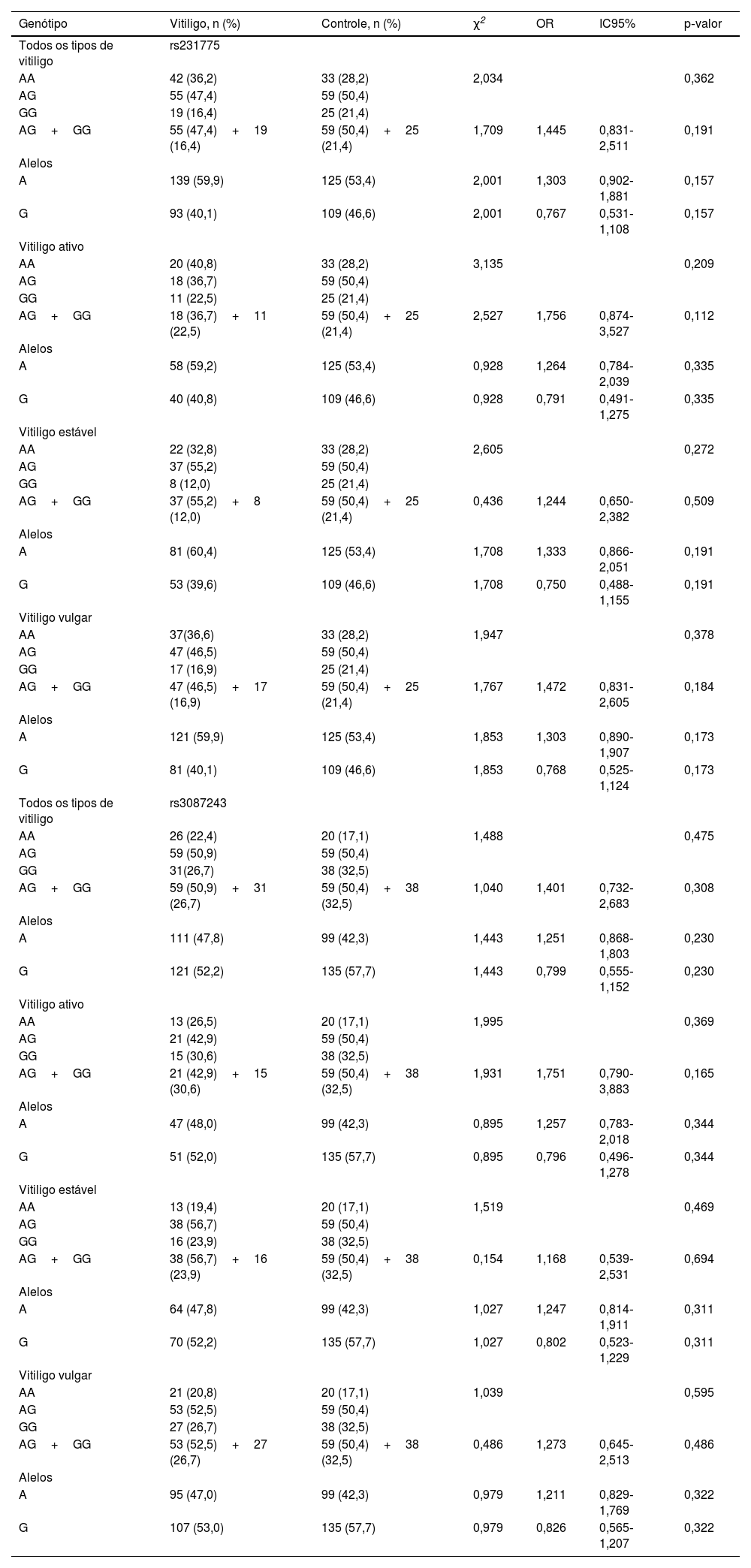
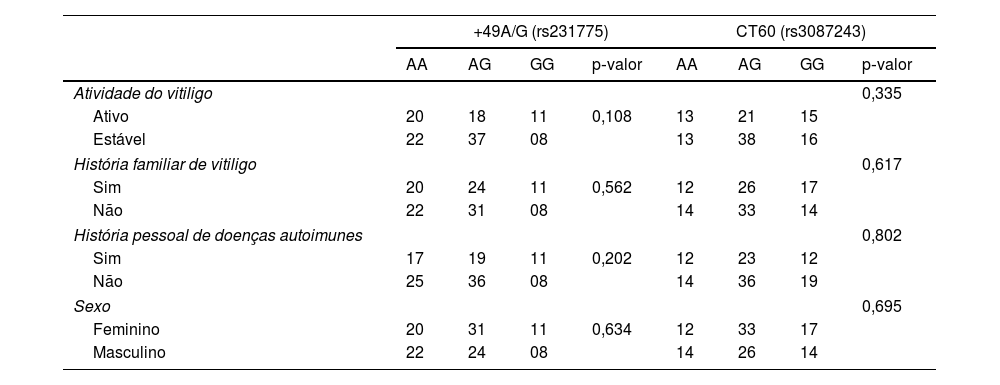
A comparação dos genótipos e/ou frequências alélicas de CTLA4+49A/G (rs231775) e CT60 (rs3087243) entre a coorte de casos e controles revelou que o genótipo AG heterozigoto (rs231775 e rs3087243) teve maior frequência em toda a coorte; entretanto, não foi observada correlação estatística entre os polimorfismos avaliados e o risco de desenvolver vitiligo (p> 0,05; tabela 3). Além disso, não foi encontrada nenhuma associação entre genótipos e atividade do vitiligo, história familiar, história pessoal de doenças autoimunes ou sexo nos pacientes analisados (p> 0,05; tabela 4).
Frequências alélicas e genotípicas das variantes do gene CTLA4+49A/G (rs231775) e CT60 (rs3087243) em pacientes com vitiligo e controles saudáveis
| Genótipo | Vitiligo, n (%) | Controle, n (%) | χ2 | OR | IC95% | p‐valor |
|---|---|---|---|---|---|---|
| Todos os tipos de vitiligo | rs231775 | |||||
| AA | 42 (36,2) | 33 (28,2) | 2,034 | 0,362 | ||
| AG | 55 (47,4) | 59 (50,4) | ||||
| GG | 19 (16,4) | 25 (21,4) | ||||
| AG+GG | 55 (47,4)+19 (16,4) | 59 (50,4)+25 (21,4) | 1,709 | 1,445 | 0,831‐2,511 | 0,191 |
| Alelos | ||||||
| A | 139 (59,9) | 125 (53,4) | 2,001 | 1,303 | 0,902‐1,881 | 0,157 |
| G | 93 (40,1) | 109 (46,6) | 2,001 | 0,767 | 0,531‐1,108 | 0,157 |
| Vitiligo ativo | ||||||
| AA | 20 (40,8) | 33 (28,2) | 3,135 | 0,209 | ||
| AG | 18 (36,7) | 59 (50,4) | ||||
| GG | 11 (22,5) | 25 (21,4) | ||||
| AG+GG | 18 (36,7)+11 (22,5) | 59 (50,4)+25 (21,4) | 2,527 | 1,756 | 0,874‐3,527 | 0,112 |
| Alelos | ||||||
| A | 58 (59,2) | 125 (53,4) | 0,928 | 1,264 | 0,784‐2,039 | 0,335 |
| G | 40 (40,8) | 109 (46,6) | 0,928 | 0,791 | 0,491‐1,275 | 0,335 |
| Vitiligo estável | ||||||
| AA | 22 (32,8) | 33 (28,2) | 2,605 | 0,272 | ||
| AG | 37 (55,2) | 59 (50,4) | ||||
| GG | 8 (12,0) | 25 (21,4) | ||||
| AG+GG | 37 (55,2)+8 (12,0) | 59 (50,4)+25 (21,4) | 0,436 | 1,244 | 0,650‐2,382 | 0,509 |
| Alelos | ||||||
| A | 81 (60,4) | 125 (53,4) | 1,708 | 1,333 | 0,866‐2,051 | 0,191 |
| G | 53 (39,6) | 109 (46,6) | 1,708 | 0,750 | 0,488‐1,155 | 0,191 |
| Vitiligo vulgar | ||||||
| AA | 37(36,6) | 33 (28,2) | 1,947 | 0,378 | ||
| AG | 47 (46,5) | 59 (50,4) | ||||
| GG | 17 (16,9) | 25 (21,4) | ||||
| AG+GG | 47 (46,5)+17 (16,9) | 59 (50,4)+25 (21,4) | 1,767 | 1,472 | 0,831‐2,605 | 0,184 |
| Alelos | ||||||
| A | 121 (59,9) | 125 (53,4) | 1,853 | 1,303 | 0,890‐1,907 | 0,173 |
| G | 81 (40,1) | 109 (46,6) | 1,853 | 0,768 | 0,525‐1,124 | 0,173 |
| Todos os tipos de vitiligo | rs3087243 | |||||
| AA | 26 (22,4) | 20 (17,1) | 1,488 | 0,475 | ||
| AG | 59 (50,9) | 59 (50,4) | ||||
| GG | 31(26,7) | 38 (32,5) | ||||
| AG+GG | 59 (50,9)+31 (26,7) | 59 (50,4)+38 (32,5) | 1,040 | 1,401 | 0,732‐2,683 | 0,308 |
| Alelos | ||||||
| A | 111 (47,8) | 99 (42,3) | 1,443 | 1,251 | 0,868‐1,803 | 0,230 |
| G | 121 (52,2) | 135 (57,7) | 1,443 | 0,799 | 0,555‐1,152 | 0,230 |
| Vitiligo ativo | ||||||
| AA | 13 (26,5) | 20 (17,1) | 1,995 | 0,369 | ||
| AG | 21 (42,9) | 59 (50,4) | ||||
| GG | 15 (30,6) | 38 (32,5) | ||||
| AG+GG | 21 (42,9)+15 (30,6) | 59 (50,4)+38 (32,5) | 1,931 | 1,751 | 0,790‐3,883 | 0,165 |
| Alelos | ||||||
| A | 47 (48,0) | 99 (42,3) | 0,895 | 1,257 | 0,783‐2,018 | 0,344 |
| G | 51 (52,0) | 135 (57,7) | 0,895 | 0,796 | 0,496‐1,278 | 0,344 |
| Vitiligo estável | ||||||
| AA | 13 (19,4) | 20 (17,1) | 1,519 | 0,469 | ||
| AG | 38 (56,7) | 59 (50,4) | ||||
| GG | 16 (23,9) | 38 (32,5) | ||||
| AG+GG | 38 (56,7)+16 (23,9) | 59 (50,4)+38 (32,5) | 0,154 | 1,168 | 0,539‐2,531 | 0,694 |
| Alelos | ||||||
| A | 64 (47,8) | 99 (42,3) | 1,027 | 1,247 | 0,814‐1,911 | 0,311 |
| G | 70 (52,2) | 135 (57,7) | 1,027 | 0,802 | 0,523‐1,229 | 0,311 |
| Vitiligo vulgar | ||||||
| AA | 21 (20,8) | 20 (17,1) | 1,039 | 0,595 | ||
| AG | 53 (52,5) | 59 (50,4) | ||||
| GG | 27 (26,7) | 38 (32,5) | ||||
| AG+GG | 53 (52,5)+27 (26,7) | 59 (50,4)+38 (32,5) | 0,486 | 1,273 | 0,645‐2,513 | 0,486 |
| Alelos | ||||||
| A | 95 (47,0) | 99 (42,3) | 0,979 | 1,211 | 0,829‐1,769 | 0,322 |
| G | 107 (53,0) | 135 (57,7) | 0,979 | 0,826 | 0,565‐1,207 | 0,322 |
Correlação entre os polimorfismos do gene CTLA4+49A/G (rs231775) e CT60 (rs3087243) com a atividade de vitiligo, história familiar, história pessoal de doenças autoimunes e sexo
| +49A/G (rs231775) | CT60 (rs3087243) | |||||||
|---|---|---|---|---|---|---|---|---|
| AA | AG | GG | p‐valor | AA | AG | GG | p‐valor | |
| Atividade do vitiligo | 0,335 | |||||||
| Ativo | 20 | 18 | 11 | 0,108 | 13 | 21 | 15 | |
| Estável | 22 | 37 | 08 | 13 | 38 | 16 | ||
| História familiar de vitiligo | 0,617 | |||||||
| Sim | 20 | 24 | 11 | 0,562 | 12 | 26 | 17 | |
| Não | 22 | 31 | 08 | 14 | 33 | 14 | ||
| História pessoal de doenças autoimunes | 0,802 | |||||||
| Sim | 17 | 19 | 11 | 0,202 | 12 | 23 | 12 | |
| Não | 25 | 36 | 08 | 14 | 36 | 19 | ||
| Sexo | 0,695 | |||||||
| Feminino | 20 | 31 | 11 | 0,634 | 12 | 33 | 17 | |
| Masculino | 22 | 24 | 08 | 14 | 26 | 14 | ||
O vitiligo é uma doença complexa, na qual os melanócitos são destruídos progressiva e seletivamente.14 Múltiplos fatores internos e externos têm sido associados ao desenvolvimento do vitiligo, incluindo alterações autoimunes, fatores genéticos, trauma epidérmico, estresse emocional e infecções, entre outros fatores de risco, que podem agir independentemente uns dos outros ou em combinação.2,15 Além disso, acredita‐se que o vitiligo possa ter padrão de herança não mendeliana, penetrância incompleta, múltiplos loci de suscetibilidade e heterogeneidade genética.16
O vitiligo tem sido associado a múltiplas variantes genéticas, muitas das quais estão envolvidas nas vias da resposta imune, principalmente em relação às regiões gênicas de classe I e classe II do antígeno leucocitário humano (HLA, do inglês human leukocyte antigen) do complexo principal de histocompatibilidade (MHC, do inglês major histocompatibility complex) e genes candidatos não MHC.17 Em relação a esses últimos, o gene CTLA4 codifica um correceptor de células T expresso por células T CD4+e CD8+, que está envolvido na ativação de células T; além disso, esse correceptor é um regulador negativo crítico da resposta das células T, desempenhando assim papel de proteção essencial contra a autoimunidade.18 A resposta autoimune mediada pelas células T CD8+é fundamental durante o processo de despigmentação do vitiligo, pois essas células são diretamente responsáveis pela destruição dos melanócitos, criando as áreas de despigmentação da pele típicas da doença.3
A associação entre as variantes do gene CTLA4+49G/A e CT60 e o desenvolvimento de diferentes doenças autoimunes foi sugerida anteriormente.19,20 Entretanto, os resultados foram inconsistentes em doenças dermatológicas; por exemplo, em casos de psoríase, sua participação ou falta de influência foi relatada.21 Uma condição semelhante foi observada durante o desenvolvimento da alopecia areata (AA), na qual um estudo de associação anterior propôs o envolvimento de polimorfismos do CTLA4 como fator de risco em seu desenvolvimento em uma população europeia;22 no entanto, outro estudo revelou que esses polimorfismos eram irrelevantes para o desenvolvimento dessa doença em uma população mexicana.9
Na América Latina, as variantes genéticas do CTLA4 só foram correlacionadas com o desenvolvimento de obesidade no nordeste do Brasil,23 diabetes mellitus tipo 1 no Chile,24 desenvolvimento do inibidor do FVIII em pacientes com hemofilia tipo A (HA) na Argentina,25 leishmaniose cutânea difusa na Venezuela26 e vírus da hepatite C27 e artrite reumatoide no México.28
Embora a participação, ou falta de influência, de ambos os polimorfismos genéticos tenha sido relatada para o vitiligo, um estudo de metanálise, incluindo 1.252 casos em quatro populações europeias, três asiáticas e duas turcas revelou que a variante do gene CTLA4 CT60 A/G confere suscetibilidade ao vitiligo apenas na população europeia.10
Em estudos anteriores, observamos o papel de variantes genéticas do TNF‐α29 e PTPN22 (https://www.sciencedirect.com/science/article/pii/S0365059620300921 ‐ bib0195)30 como fatores de risco no desenvolvimento de formas ativas de vitiligo em população mexicana. Ambos os genes estão relacionados com a regulação de mecanismos imunológicos. No entanto, a influência das variantes do gene CTLA4 no desenvolvimento do vitiligo não foi analisada em pacientes mexicanos. Talvez sem surpresa, este estudo não mostrou diferença significante na presença dos polimorfismos rs231775 ou rs3087243 na correlação com o desenvolvimento de vitiligo, sugerindo que essas variantes genéticas não constituem fator de risco para a doença em população do nordeste mexicano.
ConclusãoAs variantes do gene CTLA4 rs231775 e rs3087243 não constituem fator de risco para o desenvolvimento de vitiligo na população do nordeste mexicano. Além disso, essas variantes genéticas não têm correlação com a história pessoal de doenças autoimunes, história familiar de vitiligo ou o sexo dos pacientes.
Suporte financeiroNenhum.
Contribuição dos autoresMauricio Salinas‐Santander: Concepção, desenho e planejamento do estudo; obtenção, análise e interpretação dos dados; redação do manuscrito; revisão crítica da literatura e revisão crítica do manuscrito; aprovação da versão final do manuscrito.
Víctor Suárez‐Valencia: Análise e interpretação dos dados; redação do manuscrito; revisão crítica da literatura e revisão crítica do manuscrito; aprovação da versão final do manuscrito.
Mayela del Ángel‐Martínez: Interpretação dos dados; redação do manuscrito; revisão crítica da literatura e revisão crítica do manuscrito; aprovação da versão final do manuscrito.
David Kubelis‐Lopez: Interpretação dos dados; revisão crítica do manuscrito; aprovação da versão final do manuscrito.
Natalia Zapata‐Salazar: Interpretação dos dados; revisão crítica do manuscrito; aprovação da versão final do manuscrito.
Jorge Ocampo‐Garza: Revisão crítica do manuscrito; aprovação da versão final do manuscrito.
Jorge Ocampo‐Candiani: Revisão crítica do manuscrito; aprovação da versão final do manuscrito.
Conflito de interessesNenhum.
Os autores expressam sua gratidão aos participantes deste estudo. Agradecimentos ao Dr. Daniel Díaz por sua gentil assistência na edição e revisão deste manuscrito.
Como citar este artigo: Salinas‐Santander MA, Suárez‐Valencia VJ, del Ángel‐Martínez M, Kubelis‐Lopez DE, Zapata‐Salazar NA, Ocampo‐Garza JA, et al. Association between the CTLA4+49A/G (rs231775) and CT60 (rs3087243) gene variants with vitiligo: Study on a Mexican population. An Bras Dermatol. 2022;97:710–5.
Trabalho realizado no Departamento de Dermatologia, Dr. José E. González University Hospital, Universidad Autónoma de Nuevo León, Monterrey, México.