Polimorfismos do gene da filagrina 2 (rs 12568784 e rs 16899374) estão associados com dermatite atópica persistente em pacientes negros. A filagrina 2 é uma proteína com função semelhante à filagrina e também codificada no complexo de diferenciação da epiderme no cromossomo 1q21.
ObjetivoAvaliar os polimorfismos no gene da filagrina 2 (rs 12568784 e rs 16899374) em crianças e adultos com dermatite atópica e verificar a associação desses com a gravidade do quadro clínico, presença de outras doenças alérgicas e fatores sociodemográficos.
MétodoO estudo foi feito com pacientes do ambulatório de dermatite atópica e grupo controle. Foram usados questionários para avaliar etnia, sexo, faixa etária, histórico familiar, ScoringAtopic Dermatitis (SCORAD), entre outros parâmetros. A genotipagem do gene da filagrina 2 foi feita através de reação em cadeia da polimerase em tempo real.
ResultadosForam avaliados 48 pacientes e 83 controles. Não foi encontrada uma correlação entre as variáveis estudadas dos pacientes com dermatite atópica e os polimorfismos. Também não houve diferença significativa entre a prevalência dos polimorfismos nos pacientes e no grupo controle p > 0,05.
Limitações do estudoO uso exclusivo de informações da etnia autodeclarada e o número da amostra.
ResultadosOs resultados deste trabalho podem ser um incentivo para o estudo de polimorfismos na dermatite atópica, considerando‐se a característica multiétnica da população brasileira.
ConclusãoEste é um trabalho inédito no Brasil e o primeiro estudo no mundo a ter um grupo controle para avaliar alterações no gene da filagrina 2.
A dermatite atópica (DA) é uma doença inflamatória da pele frequente e que afeta aproximadamente 20% das crianças, compromete a qualidade de vida do paciente e da família. Mutações no gene que codifica a proteína epidérmica filagrina demonstram ser um fator importante para o desenvolvimento dessa doença.1 A filagrina também está associada com a persistência e coexistência de outras doenças alérgicas.2
A filagrina (FILAGGRIN: FILamentAGGRegatingproteIN) é uma proteína encontrada nos corneócitos responsável por agregar a queratina na formação do estrato córneo.3 É produzida a partir de um precursor, a pró‐filagrina, que se localiza na camada granulosa da epiderme nos grânulos de querato hialina. A pró‐filagrina é subsequentemente desfosforilada, resulta na formação de monómeros de filagrina que têm propriedades de agregação de filamentos de queratina. A degradação da filagrina gera os fatores de hidratação natural (Natural Mosturizing Factors − NMFs), que incluem aminoácidos livres, ácido urocânico (UCA) e ácido pirrolidônico carboxílico (PCA). Os NMFs promovem captação e aprisionamento da água e manutenção do pH ácido da pele, essencial para o processo de hidratação e manutenção da barreira cutânea.4
O gene que codifica a filagrina está localizado no cromossomo 1q21, em uma região denominada complexo de diferenciação epidérmica.3 De acordo com a literatura; 27,5% dos brancos americanos; 48% dos europeus; 31,4% dos chineses e 20% dos japoneses portadores de dermatite atópica apresentam mutações no gene da filagrina.5
Atualmente 47 mutações com perda de função já foram identificadas no gene que codifica a filagrina em pacientes com DA. Perfis distintos de mutações foram identificados em pacientes da Europa e da Ásia.6 Entre as mutações descritas, as mais comumente encontradas nos brancos são a R501X e a 2282del4 (compreendem 50% do total), além da R2447X e da S3247X.7 Na população japonesa, as principais mutações foram a 3321delA e a S2554X.8 Essas mutações parecem variar de acordo com grupos étnicos, por exemplo, na população negra estudos demonstraram uma prevalência bem menor que nos brancos.9 Gao et al.10 encontraram a R501X em 3,2% [U1] de 187 pacientes negros com dermatite atópica e nenhuma associação com 2282del4. Já Margolis et al.7 avaliaram o genótipo de 370 negros com DA e encontraram mutações em apenas 1% dos pacientes8 (R501X 3,2%; 2282del4 0,5%; S3247X 3% e R2447X 1,4%).
A filagrina 2 é uma proteína com função semelhante à filagrina e também codificada na região denominada complexo de diferenciação da epiderme. De acordo com Makino et al.,11 a expressão da proteína filagrina 2 está diminuída em portadores de DA. Há relato de uma associação entre polimorfismos no gene da filagrina 2 (rs 12568784 e rs 1689937411) e DA persistente em pacientes negros portadores de dermatite atópica.12
Não há estudos sobre a prevalência de filagrina e filagrina 2 na população brasileira. O objetivo deste estudo foi avaliar os polimorfismos no gene da filagrina 2 (rs 12568784 e rs 16899374) em crianças e adultos com dermatite atópica e verificar a associação desses com a gravidade do quadro clínico, presença de outras doenças alérgicas e fatores sociodemográficos.
MétodosEstudo transversal, envolveu pacientes de ambos os sexos, entre um e 27 anos, com diagnóstico de dermatite atópica, de acordo com os critérios da United Kingdom Working Party (UK),13 recrutados nos ambulatórios de dermatite atópica. Foram excluídos aqueles com outras doenças crônicas da pele (psoríase, entre outras) ou doenças sistêmicas que afetam a pele, como imunodeficiências primárias, lúpus eritematoso sistêmico, entre outras.
De acordo com a ordem de chegada ao ambulatório, os pacientes responderam a um questionário com as seguintes variáveis: sexo, idade, etnia, início da DA, história familiar de doenças alérgicas e presença de asma, alergia alimentar e rinite. Usou‐se o questionário ISAAC, validado para nossa linguagem,14,15 para avaliar a presença de asma e rinite. Além disso, foi feita análise da gravidade da DA pelo Scoring Atopic Dermatitis (SCORAD). Os níveis de gravidade da DA foram definidos como; leve, SCORAD até 25 pontos; moderado, entre 25–50 pontos; e grave, acima de 50 pontos.16
Após a assinatura de termos de consentimento, que relataram os principais objetivos, riscos e benefícios da pesquisa, por parte dos responsáveis e de assentimento por parte dos pacientes adolescentes, amostras de sangue venoso foram coletadas em tubos contendo EDTA. A fim de aumentar a confiabilidade do estudo em relação à análise das mutações, foram analisadas amostras controle. Devido à dificuldade ética de criar um grupo controle pediátrico com coleta de sangue, optou‐se por amostras de um mesmo grupo populacional provenientes de um banco de medula óssea, as quais foram pareadas por sexo e etnia.
Para a determinação da variação genética da filagrina 2, o DNA genômico dos indivíduos participantes do estudo foi purificado com auxílio do kit QIAamp DNA Blood Mini kit (Qiagen, Valencia, CA), de acordo com as instruções do fabricante. O princípio desse kit baseia‐se na extração de DNA genômico de células sanguíneas nucleadas e posterior ligação do DNA a uma matriz de fibra de vidro contida em uma coluna de MicroSpin. Resumidamente, após a digestão das membranas celulares pela proteinase K, o material nucleico foi transferido para uma membrana de sílica, onde sofreu várias lavagens com soluções alcoólicas. A eluição foi feita pela adição de água ultrapura. O DNA genômico foi quantificado através da medida da absorbância na densidade óptica de 260 nm (OD260) com o espectrofotômetro NanoDrop Lite (ThermoScientific). A preparação final foi armazenada a ‐20°C.
A genotipagem do gene da filagrina 2 (rs 16833974 e rs 12568784) foi feita através de PCR em tempo real, usou‐se sonda TaqMan® de uso comercial (ABI assay; Applied Biosystems) marcada com os fluoroforos VIC e FAM para cada alelo, sendo C_34256909_10 para rs16833974 e C_11261511 para rs12568784. Resumidamente, a reação foi feita em um volume final de 10 uL, com 40 ng de DNA genômico, Taqman SNP GenotypingAssays 40X e TaqmanGenotyping Master Mix, de acordo com as instruções do fabricante. Inicialmente as amostras foram submetidas a uma etapa de leitura pré‐PCR a 60°C por 30 segundos, seguida pela etapa de desnaturação a 95°C durante 10 minutos. Em seguida foram submetidas à etapa de amplificação, que consistia em 40 ciclos de 95°C por 15 segundos e 60°C por 1 minuto, para anelamento do primer e extensão dos fragmentos. Após a amplificação dos fragmentos de DNA, as amostras foram submetidas à etapa final de leitura pós‐PCR a 60°C durante 30 segundos. A reação da PCR foi feita com auxílio do termociclador ABI StepOnePlus Real‐Time PCR System (Applied Biosystems, Foster City, CA). Os resultados da análise de discriminação alélica foram plotados em um gráfico de dispersão que contrastou a fluorescência de cada sonda.
Análise estatísticaUsou‐se uma amostra de 48 pacientes com um grupo controle maior (1,7 vez) de 83 pacientes, para uma melhor análise estatística. Fez‐se uso das estatísticas de frequência, frequência relativa e teste qui‐quadrado linear para apresentação das variáveis em tabelas descritivas, com objetivo de compreender o perfil da amostra e também do controle. Com o intuito de verificar possíveis associações usou‐se o teste estatístico qui‐quadrado, ou o exato de Fisher, para identificar evidências que comprovem significância de determinadas variáveis em relação à mutação genética estudada e nível de Scoring Atopic Dermatitis (SCORAD).
Usou‐se o nível de significância de 0,05; p‐valor < 0,05 para avaliação das associações entre a variável dependente (mutações no gene da filagrina 2) e as demais variáveis de interesse do estudo. Os programas Microsoft Excel 2010 e Software R, versão 3.3.1 (R Core Team 2015, Viena, Áustria), foram empregados para organização, criação de tabelas e análise estatística dos dados. O estudo seguiu os princípios da Declaração de Helsinki e foi aprovado pelo comitê de ética sob parecer n° 36934514.9.0000.5259.
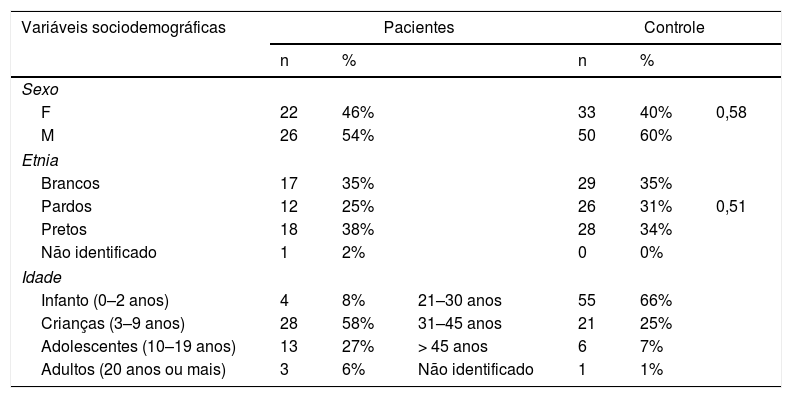
ResultadosDe novembro de 2015 a fevereiro de 2016 foram coletados os dados de 48 pacientes, 54% do sexo masculino. O grupo controle foi constituído por 83 amostras. A prevalência total do polimorfismo rs 16833974 no grupo de pacientes é 16,7% (14,6% heterozigoto e 2,1% homozigoto) e no controle 15,7% (heterozigotos). Já a prevalência total do polimorfismo rs 12568784 é: 43,7% dos pacientes (35,4% heterozigotos e 8,3% homozigotos) e dos controles 46,9% (37,3% heterozigoto e 9,6% homozigoto). As principais características da amostra e do grupo controle são descritas na tabela 1
Distribuição dos dados sociodemográficos dos pacientes e controles
| Variáveis sociodemográficas | Pacientes | Controle | ||||
|---|---|---|---|---|---|---|
| n | % | n | % | |||
| Sexo | ||||||
| F | 22 | 46% | 33 | 40% | 0,58 | |
| M | 26 | 54% | 50 | 60% | ||
| Etnia | ||||||
| Brancos | 17 | 35% | 29 | 35% | ||
| Pardos | 12 | 25% | 26 | 31% | 0,51 | |
| Pretos | 18 | 38% | 28 | 34% | ||
| Não identificado | 1 | 2% | 0 | 0% | ||
| Idade | ||||||
| Infanto (0–2 anos) | 4 | 8% | 21–30 anos | 55 | 66% | |
| Crianças (3–9 anos) | 28 | 58% | 31–45 anos | 21 | 25% | |
| Adolescentes (10–19 anos) | 13 | 27% | > 45 anos | 6 | 7% | |
| Adultos (20 anos ou mais) | 3 | 6% | Não identificado | 1 | 1% | |
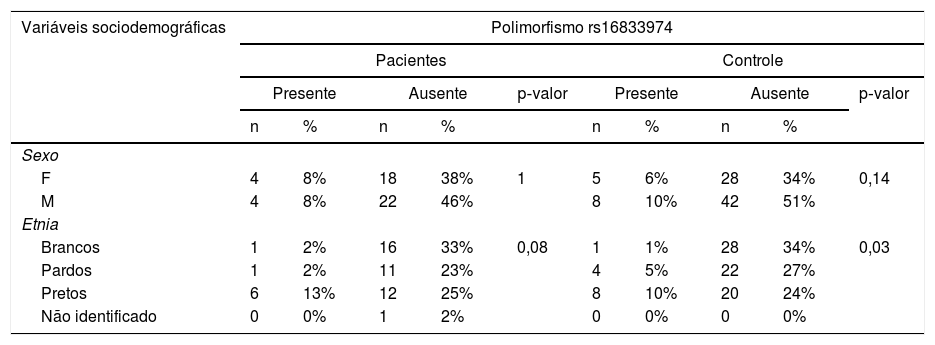
A distribuição do polimorfismo genético de rs 16833974 e rs 12568784 de acordo com as variáveis sociodemográficas da amostra e do grupo controle é demonstrada respectivamente na tabela 2.
Distribuição do polimorfismo rs16833974 e rs12568784 de acordo com as variáveis sociodemográficas da amostra e do grupo controle
| Variáveis sociodemográficas | Polimorfismo rs16833974 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Pacientes | Controle | |||||||||
| Presente | Ausente | p‐valor | Presente | Ausente | p‐valor | |||||
| n | % | n | % | n | % | n | % | |||
| Sexo | ||||||||||
| F | 4 | 8% | 18 | 38% | 1 | 5 | 6% | 28 | 34% | 0,14 |
| M | 4 | 8% | 22 | 46% | 8 | 10% | 42 | 51% | ||
| Etnia | ||||||||||
| Brancos | 1 | 2% | 16 | 33% | 0,08 | 1 | 1% | 28 | 34% | 0,03 |
| Pardos | 1 | 2% | 11 | 23% | 4 | 5% | 22 | 27% | ||
| Pretos | 6 | 13% | 12 | 25% | 8 | 10% | 20 | 24% | ||
| Não identificado | 0 | 0% | 1 | 2% | 0 | 0% | 0 | 0% | ||
| Variáveis sociodemográficas | Polimorfismo rs12568784 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Pacientes | Controle | |||||||||
| Presente | Ausente | p‐valor | Presente | Ausente | p‐valor | |||||
| n | % | n | % | n | % | n | % | |||
| Sexo | ||||||||||
| F | 11 | 23% | 11 | 23% | 0,56 | 19 | 23% | 14 | 17% | 0,17 |
| M | 10 | 21% | 16 | 33% | 20 | 24% | 30 | 36% | ||
| Etnia | ||||||||||
| Brancos | 7 | 15% | 10 | 21% | 0,38 | 10 | 12% | 19 | 23% | 0,51 |
| Pardos | 3 | 6% | 9 | 19% | 17 | 20% | 9 | 11% | ||
| Pretos | 10 | 21% | 8 | 17% | 12 | 14% | 16 | 19% | ||
| Não identificado | 1 | 2% | 0 | 0% | 0 | 0% | 0 | 0% | ||
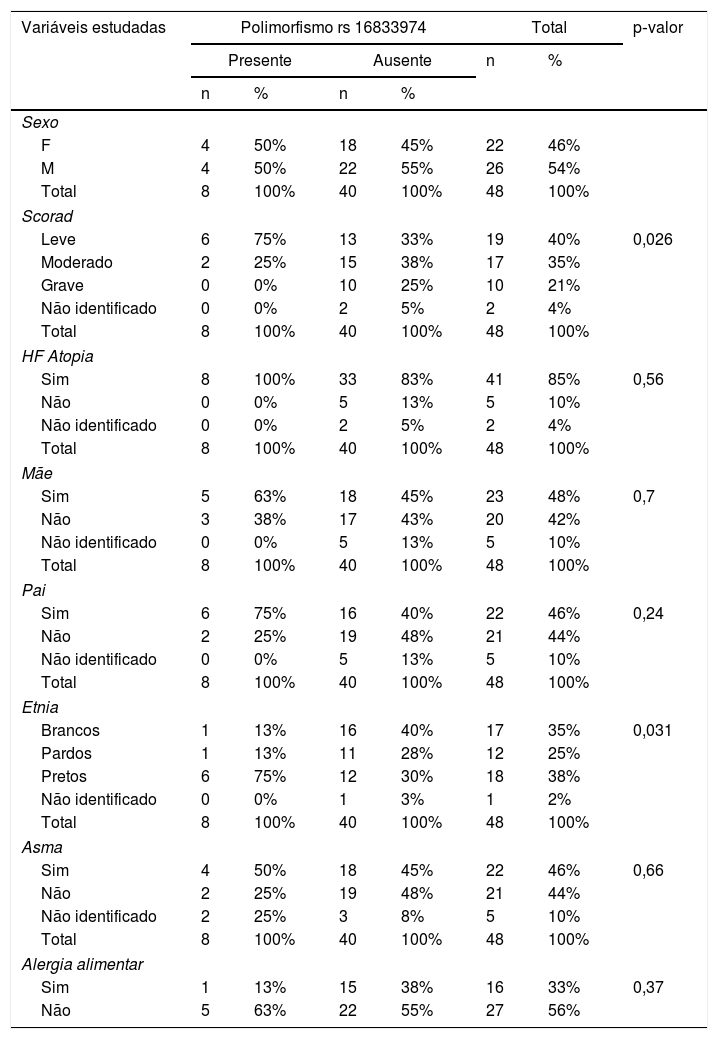
As principais variáveis estudadas foram: sexo, etnia, SCORAD, história familiar de atopia e doenças alérgicas associadas. Quanto ao polimorfismo rs 16833974, foram encontradas diferenças estatísticas quanto à variável etnia, quanto mais escura a cor da pele, maior a chance de se ter o polimorfismo, p‐valor = 0,031 (tabela 3).
Variáveis estudadas na amostra e polimorfismo rs16833974
| Variáveis estudadas | Polimorfismo rs 16833974 | Total | p‐valor | ||||
|---|---|---|---|---|---|---|---|
| Presente | Ausente | n | % | ||||
| n | % | n | % | ||||
| Sexo | |||||||
| F | 4 | 50% | 18 | 45% | 22 | 46% | |
| M | 4 | 50% | 22 | 55% | 26 | 54% | |
| Total | 8 | 100% | 40 | 100% | 48 | 100% | |
| Scorad | |||||||
| Leve | 6 | 75% | 13 | 33% | 19 | 40% | 0,026 |
| Moderado | 2 | 25% | 15 | 38% | 17 | 35% | |
| Grave | 0 | 0% | 10 | 25% | 10 | 21% | |
| Não identificado | 0 | 0% | 2 | 5% | 2 | 4% | |
| Total | 8 | 100% | 40 | 100% | 48 | 100% | |
| HF Atopia | |||||||
| Sim | 8 | 100% | 33 | 83% | 41 | 85% | 0,56 |
| Não | 0 | 0% | 5 | 13% | 5 | 10% | |
| Não identificado | 0 | 0% | 2 | 5% | 2 | 4% | |
| Total | 8 | 100% | 40 | 100% | 48 | 100% | |
| Mãe | |||||||
| Sim | 5 | 63% | 18 | 45% | 23 | 48% | 0,7 |
| Não | 3 | 38% | 17 | 43% | 20 | 42% | |
| Não identificado | 0 | 0% | 5 | 13% | 5 | 10% | |
| Total | 8 | 100% | 40 | 100% | 48 | 100% | |
| Pai | |||||||
| Sim | 6 | 75% | 16 | 40% | 22 | 46% | 0,24 |
| Não | 2 | 25% | 19 | 48% | 21 | 44% | |
| Não identificado | 0 | 0% | 5 | 13% | 5 | 10% | |
| Total | 8 | 100% | 40 | 100% | 48 | 100% | |
| Etnia | |||||||
| Brancos | 1 | 13% | 16 | 40% | 17 | 35% | 0,031 |
| Pardos | 1 | 13% | 11 | 28% | 12 | 25% | |
| Pretos | 6 | 75% | 12 | 30% | 18 | 38% | |
| Não identificado | 0 | 0% | 1 | 3% | 1 | 2% | |
| Total | 8 | 100% | 40 | 100% | 48 | 100% | |
| Asma | |||||||
| Sim | 4 | 50% | 18 | 45% | 22 | 46% | 0,66 |
| Não | 2 | 25% | 19 | 48% | 21 | 44% | |
| Não identificado | 2 | 25% | 3 | 8% | 5 | 10% | |
| Total | 8 | 100% | 40 | 100% | 48 | 100% | |
| Alergia alimentar | |||||||
| Sim | 1 | 13% | 15 | 38% | 16 | 33% | 0,37 |
| Não | 5 | 63% | 22 | 55% | 27 | 56% | |
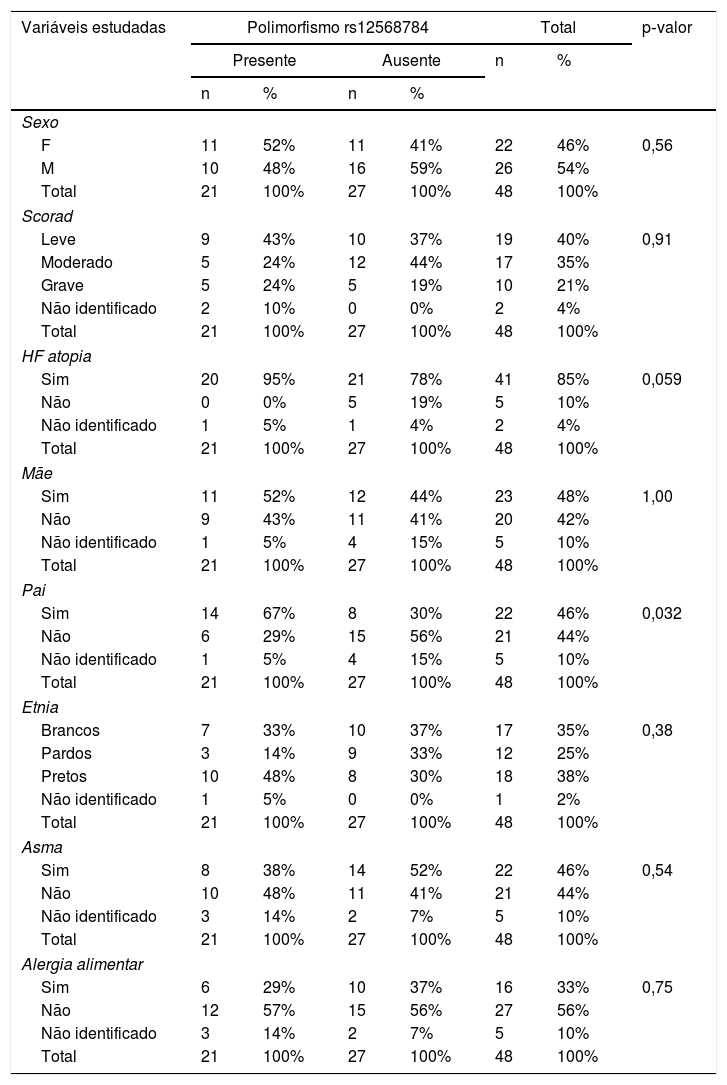
Foram encontradas diferenças estatisticamente significantes, p > 0,05, de acordo com a tabela 4.
Variáveis estudadas na amostra e polimorfismo rs1256878
| Variáveis estudadas | Polimorfismo rs12568784 | Total | p‐valor | ||||
|---|---|---|---|---|---|---|---|
| Presente | Ausente | n | % | ||||
| n | % | n | % | ||||
| Sexo | |||||||
| F | 11 | 52% | 11 | 41% | 22 | 46% | 0,56 |
| M | 10 | 48% | 16 | 59% | 26 | 54% | |
| Total | 21 | 100% | 27 | 100% | 48 | 100% | |
| Scorad | |||||||
| Leve | 9 | 43% | 10 | 37% | 19 | 40% | 0,91 |
| Moderado | 5 | 24% | 12 | 44% | 17 | 35% | |
| Grave | 5 | 24% | 5 | 19% | 10 | 21% | |
| Não identificado | 2 | 10% | 0 | 0% | 2 | 4% | |
| Total | 21 | 100% | 27 | 100% | 48 | 100% | |
| HF atopia | |||||||
| Sim | 20 | 95% | 21 | 78% | 41 | 85% | 0,059 |
| Não | 0 | 0% | 5 | 19% | 5 | 10% | |
| Não identificado | 1 | 5% | 1 | 4% | 2 | 4% | |
| Total | 21 | 100% | 27 | 100% | 48 | 100% | |
| Mãe | |||||||
| Sim | 11 | 52% | 12 | 44% | 23 | 48% | 1,00 |
| Não | 9 | 43% | 11 | 41% | 20 | 42% | |
| Não identificado | 1 | 5% | 4 | 15% | 5 | 10% | |
| Total | 21 | 100% | 27 | 100% | 48 | 100% | |
| Pai | |||||||
| Sim | 14 | 67% | 8 | 30% | 22 | 46% | 0,032 |
| Não | 6 | 29% | 15 | 56% | 21 | 44% | |
| Não identificado | 1 | 5% | 4 | 15% | 5 | 10% | |
| Total | 21 | 100% | 27 | 100% | 48 | 100% | |
| Etnia | |||||||
| Brancos | 7 | 33% | 10 | 37% | 17 | 35% | 0,38 |
| Pardos | 3 | 14% | 9 | 33% | 12 | 25% | |
| Pretos | 10 | 48% | 8 | 30% | 18 | 38% | |
| Não identificado | 1 | 5% | 0 | 0% | 1 | 2% | |
| Total | 21 | 100% | 27 | 100% | 48 | 100% | |
| Asma | |||||||
| Sim | 8 | 38% | 14 | 52% | 22 | 46% | 0,54 |
| Não | 10 | 48% | 11 | 41% | 21 | 44% | |
| Não identificado | 3 | 14% | 2 | 7% | 5 | 10% | |
| Total | 21 | 100% | 27 | 100% | 48 | 100% | |
| Alergia alimentar | |||||||
| Sim | 6 | 29% | 10 | 37% | 16 | 33% | 0,75 |
| Não | 12 | 57% | 15 | 56% | 27 | 56% | |
| Não identificado | 3 | 14% | 2 | 7% | 5 | 10% | |
| Total | 21 | 100% | 27 | 100% | 48 | 100% | |
Quanto ao polimorfismo rs12568784, também foram estudadas as seguintes variáveis: sexo, etnia, SCORAD, histórico familiar de atopia e doenças alérgicas associadas (alergia alimentar, asma e rinite) e não foi encontrada significância estatística entre as variáveis, exceto a variável parental. O teste apresentou p‐valor = 0,032, menor do que o nível de significância de 0,05 (5%). Visto isso, é possível afirmar a existência de evidências estatísticas que comprovem alguma relação de causa entre polimorfismo e atopia (Pai). De acordo com os dados da tabela pode‐se perceber uma redução no polimorfismo rs 12568784 na presença de Atopia (Pai) (tabela 4).
Foram feitos 46 cruzamentos de interesses no total, buscou‐se verificar significância estatística entre polimorfismo e história familiar, sexo, etnia, gravidade da doença, atopia (asma, rinite, alergia familiar) e comparação com o controle quanto à frequência da mutação, etnia e sexo. Buscamos fazer avaliação separadamente dos polimorfismos heterozigotos e homozigotos, porém nenhuma associação foi encontrada.
DiscussãoA relação entre polimorfismos do gene da filagrina 2 e dermatite atópica foi recentemente relatada.11 De acordo com a literatura, o polimorfismo da filagrina 2 pode influenciar a gravidade do quadro.11 Neste estudo, buscamos analisar alguns pontos dessa associação de forma ampla, através da análise exploratória e correlação de dados clínicos e demográficos.
É importante considerar que o grupo controle usado no estudo foi de conveniência, com amostras de banco de medula óssea, visto a dificuldade de organizar um grupo controle de crianças. Foi feito pareamento de sexo e etnia entre os grupos. Este é o primeiro trabalho que usa controles para avaliar polimorfismos da filagrina 2 e DA. Não foram encontradas diferenças estatísticas entre a prevalência dos polimorfismos no grupo de pacientes com DA e no grupo controle. Além disso, também não houve diferença quanto ao nível de gravidade em pacientes portadores do polimorfismo.
Da mesma forma, quando se analisaram as correlações entre a idade no início da DA e os polimorfismos, não houve diferenças estatísticas. Assim como descrito por Margolis et al.,12 também não foi encontrada associação do polimorfismo com outros doenças atópicas avaliadas (asma, rinite, alergia alimentar).
A história familiar é um importante fator de risco para o desenvolvimento da DA. Aproximadamente 70% dos pacientes com DA apresentam história familiar de doenças atópicas. A probabilidade de desenvolvimento da DA é de cerca de duas a três vezes maior em crianças com um dos pais atópicos e aumenta para três a cinco vezes se ambos os pais forem atópicos.17 De um modo geral, a história materna de DA é mais preditiva para o desenvolvimento de DA em relação à atopia paterna.18 Em nosso estudo, 85% dos pacientes apresentaram história familiar positiva para atopia, corroboraram a literatura.
De um modo geral, não foram observadas diferenças estatísticas entre história familiar de atopia e polimorfismos de filagrina 2, exceto para aumento da frequência da mutação do polimorfismo rs 12568784 na presença de atopia paterna. Esse dado pode referir‐se apenas a um achado estatístico, em função do reduzido número amostral e possivelmente sem plausibilidade biológica. Mais estudos são necessários para elucidar esse achado.
De acordo com Margolis et al.,12 os polimorfismos do gene da filagrina 2, rs 12568784 e rs 16833974, estão associadas com a persistência da DA em negros e estão ausentes, ou são raramente encontrados, em indivíduos de origem europeia. Na população do estudo, houve significância estatística entre o polimorfismo rs 16833974 e os pacientes com DA autodeclarados negros, o que corroborou a literatura.
A população brasileira tem um alto grau de miscigenação e segue diferentes padrões étnicos quando comparada a grupos populacionais de outros países e continentes, como China, Estados Unidos, Japão e Europa, onde já existem estudos com prevalência da filagrina.5 Portanto, é difícil estabelecer uma classificação por etnias em nosso país. O uso exclusivo de informações da etnia autodeclarada não é o melhor método para classificação étnica19 e é uma limitação do estudo.
Os genomas da maioria dos brasileiros são miscigenados e os marcadores genéticos são capazes de fornecer novos e valiosos insights sobre a estrutura atual da população brasileira.19
Perspectivas futuras de análises de ancestralidade das amostras sanguíneas de nosso estudo poderão permitir um melhor esclarecimento acerca da associação entre a filagrina 2 e DA, visto que a prevalência das mutações do gene da filagrina apresenta diferenças étnicas tanto na população geral quanto em indivíduos portadores de DA.20
O pequeno tamanho amostral pode ter limitado a significância de nossos resultados e talvez uma maior amostra e/ou o estudo de ancestralidade poderá contribuir para o esclarecimento entre indivíduos com DA e os polimorfismo da filagrina 2. A feitura do estudo proteômico da filagrina 2, já que diferenças estruturais podem se dever a controles pós‐transcricionais, também contribuiria para uma melhor avaliação da filagrina 2.
Até o momento, não há estudos de polimorfismos do gene da filagrina 2 e DA na América Latina, o que reforçando a relevância de nosso estudo e o ineditismo dessa iniciativa no Brasil. Além disso, este é o primeiro estudo em nível mundial que usa um grupo controle para avaliar alterações no gene da filagrina 2.
ConclusãoMuito pouco se sabe sobre o perfil de polimorfismos de filagrinas 2 associados à DA na altamente miscigenada população brasileira. Os resultados deste trabalho podem ser um considerável incentivo às pesquisas sobre polimorfismos e DA no país, colaborar para uma melhor compreensão, não apenas de formas de tratamento da doença, mas também do perfil genético da população.
Quanto ao polimorfismo rs16833974, foram encontradas diferenças estatisticamente significantes em relação à variável etnia. Quanto mais escura a cor da pele, maior a chance de ocorrer o polimorfismo, valor de p=0,031.
Suporte financeiroFundo de Apoio à Dermatologia (Funaderm), da Sociedade Brasileira de Dermatologia.
Contribuição dos autoresAmanda Hertz: Concepção e planejamento do estudo; obtenção, análise e interpretação dos dados; revisão crítica da literatura.
Luna Azulay‐Abulafia: Participação efetiva na orientação da pesquisa.
Adriana Paulino do Nascimento: Concepção e planejamento do estudo; obtenção, análise e interpretação dos dados.
Cintya Yumi Ohara: Concepção e planejamento do estudo.
Fabio Chigres Kuschnir: Revisão crítica da literatura; revisão crítica do manuscrito.
Luís Cristovão Porto: Concepção e planejamento do estudo; obtenção, análise e interpretação dos dados.
Conflitos de interesseNenhum.
Como citar este artigo: Hertz A, Azulay‐Abulafia L, Nascimento AP, Ohara CY, Kuschnir FC, Porto LC. Analysis of filaggrin 2 gene polymorphisms in patients with atopic dermatitis. An Bras Dermatol. 2020;95:173–179.
Trabalho realizado no Ambulatório de Dermatite Atópica, Policlínica Piquet Carneiro, Universidade do Estado do Rio de Janeiro, RJ, Brasil.