O vitiligo é doença autoimune da pele que resulta em máculas brancas localizadas ou disseminadas. Uma característica comum de vários protocolos de classificação existentes é a distribuição da doença em dois subtipos principais, vitiligo não segmentar (VNS) e vitiligo segmentar (VS). O VS é caracterizado pela despigmentação que se espalha em um ou mais segmentos da pele, enquanto o VNS é disseminado. Várias observações clínico‐epidemiológicas sugerem que o VS tem fisiopatologia autoimune distinta em relação ao VNS. Além disso, o padrão de distribuição clínica das lesões de VS assemelha‐se muito a outras doenças com mosaicismo de melanócitos. Essas observações levam à suposição de que o VS seja causado por reação autoimune localizada direcionada ao mosaicismo de melanócitos epidérmicos. Aqui, os autores propõem exemplos de abordagens experimentais para avaliar o mosaicismo em pacientes com VS.
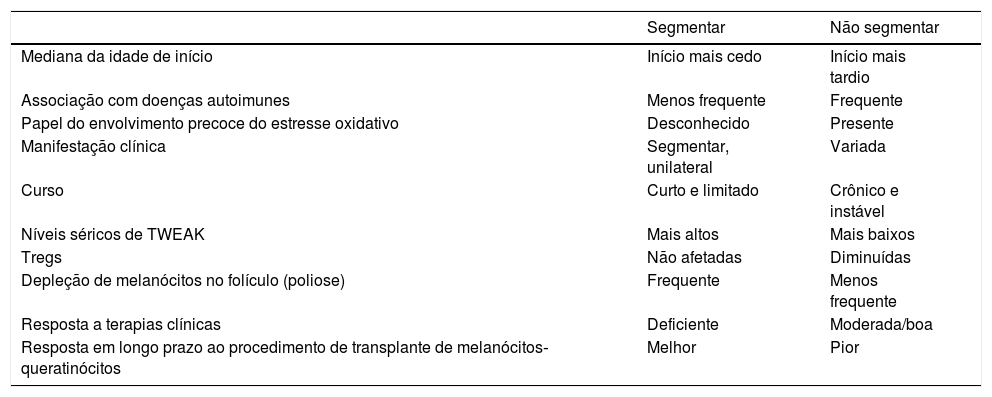
De acordo com consenso internacional, o vitiligo é classificado em três grupos principais: vitiligo segmentar (VS), não segmentar (VNS) e misto (coexistência de VS e VNS).1 O consenso descreve o VS como despigmentação que se espalha dentro do segmento, uni, bi ou pluri‐segmentar; entretanto, essa distribuição pode ocasionalmente ser bilateral.1,2 O padrão de distribuição segmentar não é a única diferença entre o VS e VNS (tabela 1). A mediana da idade de início do VS é de 16 anos, em média oito a dez anos antes da mediana da idade de início do VNS.3–5 Além disso, a despigmentação no VS geralmente tem progressão rápida, com curso de tempo limitado entre seis a 24 meses, raramente se estendendo após esse período, enquanto o VNS é crônico, apresentando evolução incerta ao longo da vida.1,6 Em contraste com o VNS, o VS apresenta envolvimento precoce dos melanócitos dos folículos pilosos; até 50% dos pacientes com VS apresentam poliose na área afetada.1 A prevalência de distúrbios autoimunes concomitantes (p. ex., tireoidite) é menor no VS.3,7 VS e VNS também diferem em relação à resposta ao tratamento: em geral, os pacientes com VS têm resposta deficiente à fototerapia em comparação com o VNS, possivelmente em decorrência da depleção da reserva folicular no primeiro.8,9 Por outro lado, pacientes com VS têm resposta excelente e em longo prazo a intervenções cirúrgicas, como transplante de melanócitos‐queratinócitos.10,11 O sucesso em longo prazo da terapia de transplante no VS sugere defeito limitado do metabolismo dos melanócitos‐queratinócitos.10
Características do vitiligo segmentar e não segmentar
| Segmentar | Não segmentar | |
|---|---|---|
| Mediana da idade de início | Início mais cedo | Início mais tardio |
| Associação com doenças autoimunes | Menos frequente | Frequente |
| Papel do envolvimento precoce do estresse oxidativo | Desconhecido | Presente |
| Manifestação clínica | Segmentar, unilateral | Variada |
| Curso | Curto e limitado | Crônico e instável |
| Níveis séricos de TWEAK | Mais altos | Mais baixos |
| Tregs | Não afetadas | Diminuídas |
| Depleção de melanócitos no folículo (poliose) | Frequente | Menos frequente |
| Resposta a terapias clínicas | Deficiente | Moderada/boa |
| Resposta em longo prazo ao procedimento de transplante de melanócitos‐queratinócitos | Melhor | Pior |
Os relatos sugerem diferenças nos mecanismos biológicos subjacentes à patogênese do VS em comparação com o VNS. Por exemplo, os níveis séricos de TWEAK (indutor fraco de apoptose semelhante ao fator de necrose tumoral) foram significantemente mais altos em pacientes com VS em comparação com pacientes com VNS.12 Além disso, foi demonstrado que o TWEAK é um biomarcador com 100% de sensibilidade e 80,1% de especificidade para diferenciar VS de VNS.12 Em contraste com o VNS, o estresse oxidativo sistêmico tem contribuição fraca e limitada para a patogênese do VS.13,14 No VS, aumento significante de marcadores induzidos por estresse (p. ex., mitocondrial, HSP70 e CXCL16) foi observado apenas na pele perilesional, sugerindo mecanismo patogênico localizado que promove a despigmentação. A análise imunofenotípica de células imunes circulantes em pacientes com VS identificou células T reguladoras (Tregs) inalteradas em comparação com controles saudáveis, enquanto os pacientes com VNS apresentaram níveis reduzidos de Tregs.15 Coletivamente, várias evidências indicam imunidade sistêmica inalterada em pacientes com VS e apontam para uma reação citotóxica localizada direcionada aos melanócitos epidérmicos.15 Em conjunto, essas diferenças e a notável semelhança do padrão de distribuição do VS com as doenças melanocíticas em mosaico (como lentiginose segmentar e nevo epidérmico verrucoso) levam à hipótese do envolvimento do mosaicismo somático na patogênese do VS.16,17
Como testar a hipóteseMosaicismo designa indivíduos que englobam pelo menos duas populações de células derivadas de um único zigoto, mas com genótipos ou perfis epigenéticos distintos.18 A apresentação fenotípica de uma doença causada por mosaicismo genético é condicionada pelo tipo de variação e fase de desenvolvimento em que ocorreu uma mutação somática.19 As variantes que levam ao mosaicismo genético compreendem duplicações cromossômicas, translocações de segmentos, variação do número de cópias (VNC), variantes de nucleotídeo único (SNV – single‐nucleotide variants) ou alterações epigenéticas, como alterações transcriptômicas decorrentes de inserções por retrotransposição. A fase embrionária e o estado de diferenciação celular onde ocorreu uma mutação de novo ou retrotransposição delineiam a extensão dos tecidos/células envolvidos no mosaicismo. A hipótese a ser testada sugere que o mosaicismo genético no VS ocorreu em algum momento durante a diferenciação da pele/melanócitos. Diferentes abordagens podem ser aplicadas para testar a hipótese de mosaicismo no VS, cada uma visando detectar um tipo distinto de mosaicismo. Aqui são propostos exemplos adaptando desenhos aplicados para estudar a resposta do hospedeiro a infecções, detectar mutações somáticas no câncer e avaliar o desenvolvimento embrionário.20–23
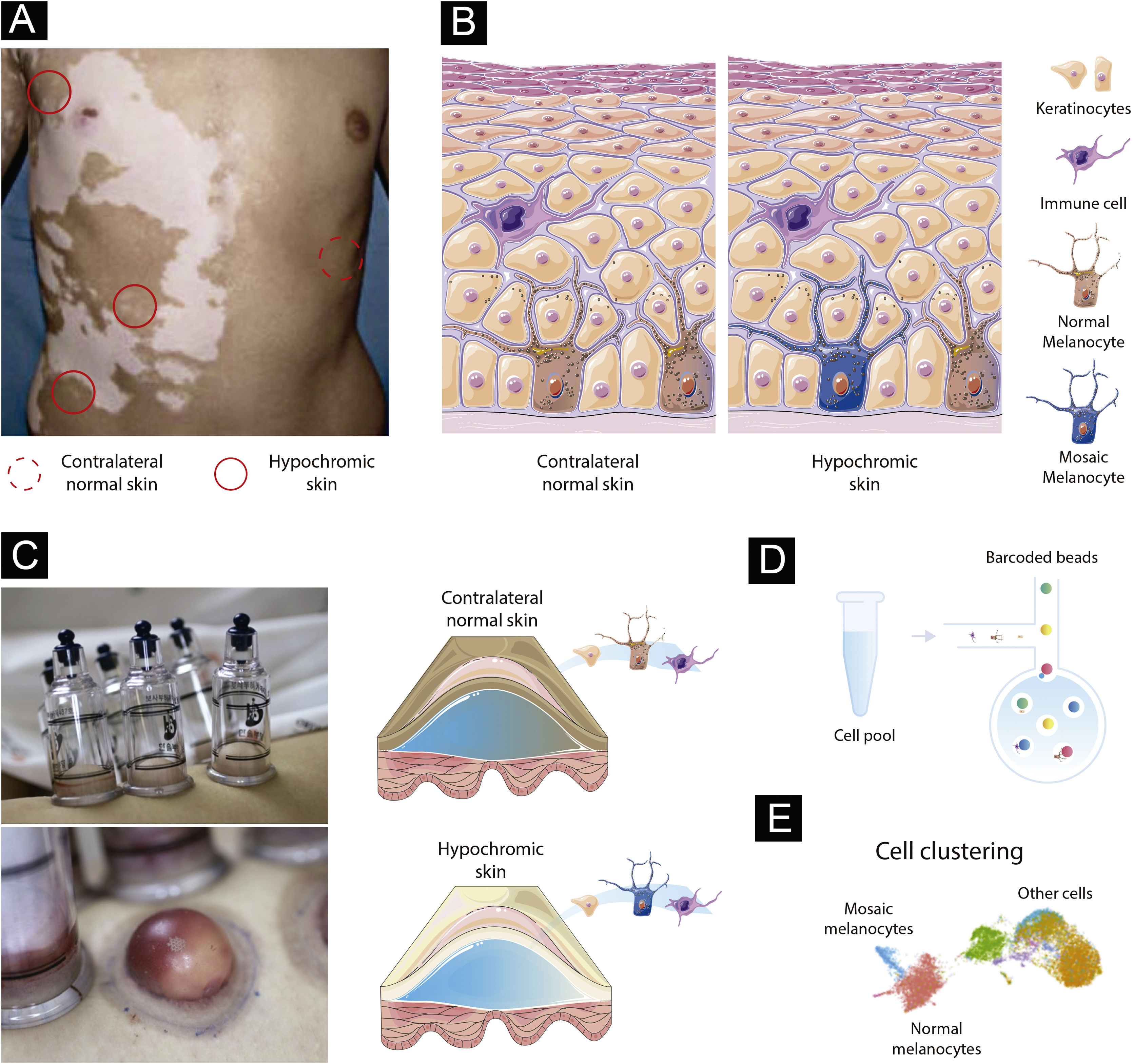
Contraste pareado de pele perilesional e pele saudável contralateral no VSA detecção de mosaicismo em doenças humanas pode ser um desafio, pois o número de células de mosaico no tecido alvo pode ser pequeno. Além disso, os melanócitos em mosaico provavelmente estariam ausentes nas lesões de VS existentes, pois a perda de melanócitos é a causa do vitiligo. Portanto, métodos de alta resolução ao nível de célula única podem ser necessários para detectar populações de células sub‐representadas.24 Para detectar possíveis células em mosaico, seria necessário avaliar o tecido obtido de regiões hipocrômicas da pele de indivíduos no estágio inicial do VS (fig. 1A). Uma vantagem de estudar a hipótese do mosaicismo no VS é a possibilidade de usar controles internos, uma vez que o VS geralmente é restrito a um segmento unilateral: se a mutação de novo ocorreu durante a diferenciação da pele em um segmento, a pele saudável contralateral poderia ser usada para estabelecer o perfil “normal” do melanócito (fig. 1A). Essa estratégia permitiria controlar os efeitos de confusão causados pela variabilidade interindividual ao combinar vários casos de VS.25
Desenho experimental para testar a hipótese do mosaicismo no vitiligo segmentar. (A) Representação de paciente com vitiligo segmentar. Áreas hipocrômicas destacadas com um círculo vermelho completo seriam avaliadas quanto à presença de melanócitos em mosaico. Amostra de pele contralateral marcada com círculo pontilhado seria usada como controle interno. (B) Composição cutânea para pele contralateral e hipocrômica. A pele hipocrômica inclui melanócitos em mosaico residuais. (C) Técnica de bolhas de sucção subepidérmica de pele normal e afetada. A tripsinização do teto da bolha possibilita o desprendimento de queratinócitos e melanócitos, que podem estar junto com células imunoinfiltradas, utilizadas nos experimentos com células únicas. (D) Código de barras de célula única. O conjunto contendo a suspensão de células para a pele normal e afetada seria carregado no canal de célula única. Células individuais seriam incorporadas em gotículas de óleo e marcadas com esferas com código de barras exclusivos, o que possibilitaria a aplicação de diferentes conjuntos de abordagens de célula única, incluindo scWGS, scRNA e scATAC. (E) Cluster de células utilizando dados ômicos. A análise de clusters agruparia células que compartilham estados semelhantes e identificaria melanócitos em mosaico, bem como outros tipos de células incluídos no tecido da bolha.
Os melanócitos representam ∼2,8% da população de células na epiderme e existem aproximadamente 1.200 melanócitos por mm2 de pele, independentemente da etnia do indivíduo (fig. 1B).26 Para capturar um número representativo de melanócitos em mosaico viáveis para a análise de célula única, áreas hipocrômicas de VS ativo de início precoce precisariam ser identificadas utilizando a lâmpada de Wood. A amostragem de queratinócitos‐melanócitos dessas áreas pode ser realizada com uma técnica de enxertia epidérmica por bolhas de sucção com tripsinização subsequente do teto da bolha para destacar as células em suspensão (fig. 1C).27 Uma vantagem de coletar epiderme usando a bolha de sucção é a ampliação da área amostrada, o que aumenta a probabilidade de capturar melanócitos em mosaico viáveis remanescentes – é uma técnica quase sem cicatrizes, o que facilita a inclusão de pacientes.28 Para avaliar melanoblastos foliculares não capturados pelo método da bolha de sucção, a técnica por punch também pode ser utilizada. A biópsia por punch é mais invasiva do que a bolha, e a proporção geral de melanoblastos capturados seria pequena. No entanto, refinamentos metodológicos de análise de célula única podem permitir em um futuro próximo o estudo de populações de células menores.
Identificando células em mosaico no vitiligo segmentar por meio de variações genéticas e epigenéticasPara avaliar a presença de melanócitos em mosaico, a seleção por amostragem celular ativada por fluorescência pode ser usada para separar a fração de melanócitos em tecido de bolhas ou melanoblastos em biópsias por punch. Em seguida, seria realizado o sequenciamento do genoma completo (WGS – whole genome sequencing) utilizando DNA extraído de melanócitos da pele hipocrômica e contralateral dos mesmos indivíduos. As variantes detectadas em tecidos contralaterais seriam utilizadas para excluir variantes germinativas. Algoritmos projetados para detectar células tumorais, como MuTect2,29 ou específicos para células em mosaico, como MosaicForecast30 e DeepMosaic,31 poderiam ser usados para detectar mutações somáticas. A limitação da abordagem em massa é a incapacidade de separar as células em mosaico dos melanócitos regulares e avaliar sua interação com outras células. Abordar essa limitação exigiria tecnologias de sequenciamento de célula única (sc – single‐cell) avaliando o perfil de sequência transcriptômica, epigenética ou de DNA de células individuais (fig. 1D).32 Os scWGS possibilitam a avaliação da variação estrutural, VNC e SNV em células individuais. Um método scWGS de alto rendimento desenvolvido para detectar mutações subclonais em câncer pode ser usado para testar a hipótese de mosaicismo de melanócitos.22 No VS, as células do mosaico compartilhariam um estado (isto é, variantes genéticas específicas) que poderia ser usado para agrupar células em clusters e definir a proporção e o tipo de variantes genéticas presentes nas células do mosaico (fig. 1E).33 O mosaicismo epigenético no VS pode ser testado usando abordagem multiômica com medidas transcriptômicas (scRNA) e de acessibilidade da cromatina (scATAC). Abordagens multiômicas estão sendo utilizadas atualmente para estudar um conjunto diversificado de doenças.34 Análises de scRNA e scATAC podem ser usadas para identificar células de mosaico por meio de semelhanças transcriptômicas e epigenômicas com ferramentas como Seurat,35 Monocle36 e Cicero.37 Abordagem semelhante de scRNA aplicada para estudar embriões humanos foi capaz de detectar com sucesso o mosaicismo.20 Uma vantagem da célula única em comparação com as abordagens em massa é a capacidade de estudar a relação entre as células presentes no tecido da bolha ou nas biópsias por punch. De fato, o estudo de scRNA de tecido de bolhas mostrou a recuperação de melanócitos de lesões de VNS.24 A aplicação da abordagem de célula única para estudar o VS pode testar a hipótese do mosaicismo ao avaliar o perfil imunológico local, como demonstrado para o VNS.24
Suporte financeiroNenhum.
Contribuição dos autoresGerson Dellatorre: Concepção do estudo, redação e aprovação final do manuscrito.
Vinicius Medeiros Fava: Concepção do estudo, redação e aprovação final do manuscrito.
Marcelo Távora Mira: Redação e aprovação final do manuscrito.
Caio Cesar Silva de Castro: Concepção do estudo, redação e aprovação final do manuscrito.
Conflito de interessesNenhum.