O carcinoma espinocelular cutâneo (CECC) é um dos tipos de câncer de pele mais comuns em todo o mundo. Portanto, a identificação de biomarcadores associados à progressão do CECC poderia auxiliar na detecção precoce do carcinoma espinocelular de alto risco e no desenvolvimento de novas estratégias terapêuticas.
ObjetivoEste estudo teve como objetivo investigar os padrões de expressão da proteína silent mating type information regulation 2 homolog 6 (SIRT6) em CECC e seu significado clínico.
MétodosO nível de expressão da proteína SIRT6 nos tecidos foi detectado por imuno‐histoquímica, e a correlação entre a expressão da SIRT6 e parâmetros clinicopatológicos em pacientes com CECC foi analisada. A expressão relativa de SIRT6 na linhagem celular de CECC e em amostras de tecido cutâneo foi determinada por western blotting e PCR. O efeito do silenciamento da SIRT6 na proliferação celular foi avaliado utilizando o kit‐8 de contagem de células. A cicatrização de feridas, o método transwell e a citometria de fluxo foram utilizados para investigar a migração, invasão e distribuição/apoptose do ciclo celular de células de CECC após o silenciamento da SIRT6, respectivamente. Western blot foi utilizado para detectar a expressão de transição epitelial‐mesenquimal (TEM), ciclo, apoptose e outras proteínas relacionadas.
ResultadosA alta expressão de SIRT6 foi correlacionada com a localização do tecido neoplásico e o estadiamento de Broder em pacientes com CECC. O silenciamento da SIRT6 inibiu a proliferação, migração, invasão e TEM de células de CECC e promoveu sua apoptose; as células foram bloqueadas na fase G1.
Limitações do estudoNenhum experimento com animais foi realizado para verificar adicionalmente os resultados.
ConclusãoA redução da expressão de SIRT6 pode inibir a ocorrência e o desenvolvimento de CECC.
O carcinoma espinocelular cutâneo (CECC) está consolidado como o segundo câncer de pele não melanoma mais comum, com incidência crescente variando de 20% a 50% de todos os tipos de câncer de pele.1 A progressão do CECC é determinada por uma combinação de suscetibilidade genética e fatores de risco ambientais, incluindo exposição à radiação ultravioleta, agentes cancerígenos e medicamentos imunossupressores.2 Apesar do risco relativamente baixo de metástases e da sobrevida favorável em longo prazo após o tratamento, a incidência de CECC aumentou proporcionalmente nos últimos anos, resultando em morbidade, mortalidade e ônus econômico significantes no longo prazo para os pacientes.3–5 Portanto, uma compreensão mais profunda da patogênese do CECC é crucial para melhorar o diagnóstico e a eficácia do tratamento.6
As sirtuínas, que pertencem à família das histonas desacetilases, estão envolvidas em várias funções celulares, como envelhecimento, resposta ao estresse, metabolismo e homeostase energética.7–9 As sirtuínas de mamíferos consistem em sete membros da família (SIRT1‐7), que exibem diferentes localização subcelular, atividades catalíticas e funções biológicas.10 A SIRT6, localizada principalmente no núcleo, funciona como desacetilase dependente de NAD+ e ADP‐ribosiltransferase altamente conservadas.11,12 A SIRT6 tem sido implicada em diabetes, doenças cardiovasculares, envelhecimento e câncer.13–16 Evidências emergentes sugerem forte ligação entre SIRT6 e o início e progressão do mieloma múltiplo (MM) e vários outros tipos de neoplasias.17,18 Seu duplo papel de suprimir ou promover o câncer foi observado em diferentes tipos de câncer, potencialmente influenciado por vias de sinalização e contextos tecido‐específicos.19,20 Entretanto, o impacto preciso da SIRT6 no CECC permanece em grande parte indefinido. A hipótese dos autores é que a SIRT6 desempenha papel fundamental na patogênese e progressão do CECC, justificando avaliação de seus padrões de expressão, mecanismos funcionais e potenciais implicações clínicas.
Materiais e métodosMateriaisReagentesForam utilizados os seguintes reagentes: linhagem celular de carcinoma espinocelular cutâneo humano A431 (obtida de Zhongqiao Xinzhou, Xangai) e linhagem celular SCL‐1 (Enzyme Research, Xangai); a linhagem de queratinócitos humanos normais imortalizados HaCaT (Shanghai Fuheng Biotechnology Co., Ltd.); leite desnatado, clorofórmio e isopropanol (adquirido da Shanghai Shenggong Co., Ltd.); anticorpos primários contra SIRT6 e β‐tubulina, anticorpos secundários contra imunoglobulina IgG de camundongo (adquiridos de Abcam, EUA); anticorpo monoclonal de N‐caderina anti‐humano de camundongo e anticorpo monoclonal de vimentina anti‐humano de camundongo (Proteintech, Wuhan); soro bovino fetal e meio DMEM com alto teor de glicose (adquirido de Gibco, EUA); kit de análise BCA, cristal violeta e lipofectamina 2000 (adquiridos de Biyuntian Biotechnology Co., Ltd.); membrana de fluoreto de polivinilideno (PVDF; adquirida de Millipore, EUA); reagente trizol, kit de tecido puro para preparação de RNA, kit de síntese PrimeScript RT e kit SYBR Green qRT‐PCR Master Mix (adquiridos de Thermo, EUA); kit de ensaio CCK‐8 (Proteintech, Wuhan); gel de matriz e câmaras transwell de 8μm (Corning, EUA). Todos os primers foram sintetizados pela Shanghai Shenggong Co., Ltd.
Amostras de tecidoBlocos de tecido de 70 pacientes com CECC, de 25 pacientes com doença de Bowen (DB) e de 60 pacientes com ceratose actínica (CA) foram coletados de janeiro de 2017 a dezembro de 2022. Além disso, 20 amostras de pele normal de cirurgia plástica foram coletadas como controles normais. Foram coletadas amostras frescas de CECC e de pele normal (TN), 20 de cada. Além disso, nenhum dos pacientes deste estudo recebeu qualquer tratamento antes da cirurgia, incluindo quimioterapia ou radioterapia. Todos os pacientes participantes do estudo forneceram consentimento informado, e este estudo foi aprovado pelo Comitê de Ética do hospital (número de aprovação: 2021KT085).
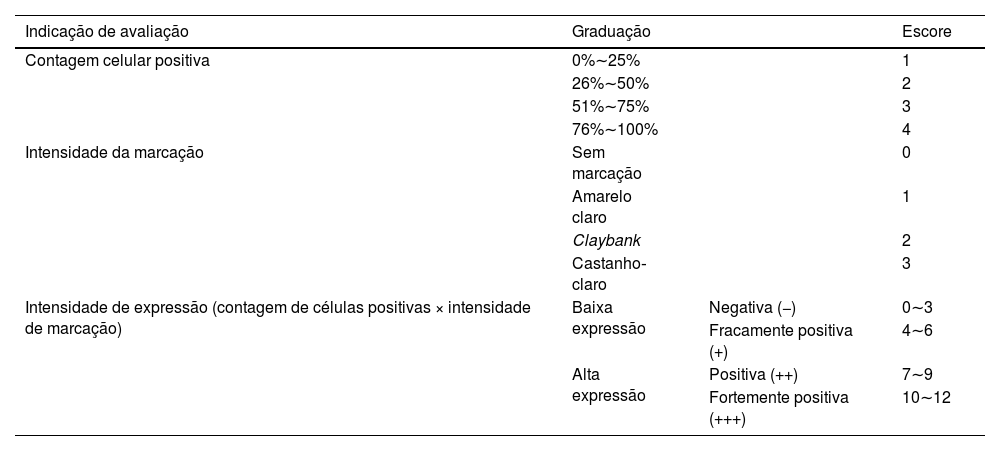
MétodosImuno‐histoquímica para análise de expressão da proteínaAmostras de tecido de CECC foram desparafinizadas e tratadas com peróxido de hidrogênio por 5 minutos para inibir a atividade da peroxidase endógena. Foi adicionada solução bloqueadora de soro de cabra, e a recuperação antigênica foi realizada com solução de citrato. Amostras de tecido de CECC foram incubadas com anticorpo anti‐SIRT6 de camundongo (1:200) durante a noite a 4°C. Após duas lavagens com PBS por 3 minutos cada, as amostras foram incubadas com IgG de cabra anticamundongo conjugado com HRP (1:2000) por 1,5 hora à temperatura ambiente. Após duas lavagens com PBS durante 3 minutos cada, o anticorpo primário foi detectado utilizando um kit de substrato de diaminobenzidina (DAB). Imagens de marcação imuno‐histoquímica foram observadas ao microscópio e vários campos foram selecionados aleatoriamente para observação em grande aumento. Os resultados da marcação imuno‐histoquímica foram interpretados usando um método de escore semiquantitativo comumente utilizado21 (tabela 1).
Resultados da marcação para SIRT6 avaliados utilizando método de escore semiquantitativo de 12 pontos
| Indicação de avaliação | Graduação | Escore | |
|---|---|---|---|
| Contagem celular positiva | 0%∼25% | 1 | |
| 26%∼50% | 2 | ||
| 51%∼75% | 3 | ||
| 76%∼100% | 4 | ||
| Intensidade da marcação | Sem marcação | 0 | |
| Amarelo claro | 1 | ||
| Claybank | 2 | ||
| Castanho‐claro | 3 | ||
| Intensidade de expressão (contagem de células positivas × intensidade de marcação) | Baixa expressão | Negativa (−) | 0∼3 |
| Fracamente positiva (+) | 4∼6 | ||
| Alta expressão | Positiva (++) | 7∼9 | |
| Fortemente positiva (+++) | 10∼12 |
As linhagens celulares utilizadas neste estudo incluíram a linhagem celular de carcinoma espinocelular da pele humana (A431, SCL‐1) e a linhagem de queratinócitos normais humanos imortalizados HaCaT. O meio utilizado para células A431, SCL‐1 e HaCaT foi meio DMEM com alto teor de glicose. Todas as células foram cultivadas a 37°C com 5% de CO2 e 10% de soro bovino fetal no meio.
Western blotA lise celular foi realizada utilizando tampão RIPA, e a proteína foi extraída em gelo. A concentração de proteína foi determinada utilizando um kit de ensaio BCA. Amostras de proteína de cada grupo foram carregadas em gel a 12,5% e submetidas à eletroforese a 80V por 30 minutos, seguida de 110V por 1 hora. A transferência para membrana PVDF foi realizada a 100V em banho de gelo por 1,5 hora. A membrana foi então bloqueada com leite desnatado a 5% durante 2 horas, seguida de incubação com anticorpo primário a 4°C durante a noite e incubação com anticorpo secundário por 2 horas. Os anticorpos secundários utilizados foram HRP de cabra anticamundongo (diluição 1:1000) e HRP de cabra anticoelho (diluição 1:1000). Por fim, as imagens foram capturadas e armazenadas utilizando sistema de imagem por quimiluminescência Bio‐Rad.
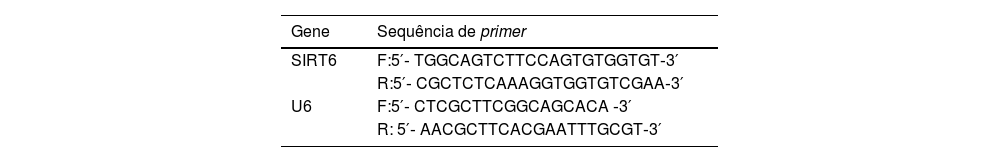
Reação em cadeia da polimerase via transcriptase reversa quantitativa (qRT‐PCR)Pellets celulares foram coletados e tratados com reagente trizol para lise, seguido de extração com clorofórmio/isopropanol para separar e precipitar RNA. O RNA total foi posteriormente extraído por precipitação com etanol. A transcrição reversa de 1μg de RNA em cDNA foi realizada utilizando o kit de síntese PrimeScript RT. A qRT‐PCR foi realizada utilizando o kit SYBR Green qRT‐PCR Master Mix, com U6 sendo utilizado como referência interna. As sequências de primers estão listadas na tabela 2.
Infecção celular e triagemO shSIRT6 lentiviral e o controle shNC foram fornecidos por Youxi Weinan Biotechnology Company (Sanming, China). Os lentivírus foram envelopados usando um sistema de plasmídeo triplo. O shRNA foi inserido no vetor de expressão gênica induzível por doxiciclina (DOX) Tet‐pLKO.1‐puro. Após transfecção durante 36 horas, os lentivírus foram colhidos. Células A431 e células SCL‐1 foram infectadas com os lentivírus por 24 horas e linhagens celulares transduzidas estáveis foram selecionadas usando puromicina (0,8μg/mL para células A431; 0,4μg/mL para células SCL‐1). A expressão gênica foi induzida com 1μg/mL de DOX e células transfectadas com vetor vazio serviram como células de controle normal (CN). As células foram divididas em quatro grupos: grupo CN, grupo CN+DOX, grupo shSIRT6 e grupo shSIRT6+DOX.
Ensaio Cell Counting Kit‐8 (CCK‐8) para proliferação celularAs células foram semeadas a 2.000 células/100μL em placa de 96 poços com quatro poços replicados por grupo. A placa foi incubada a 37°C com 5% de CO2 e umidade saturada até as células atingirem 80% de confluência. Cada grupo de células foi tratado de maneira diferente e incubado por mais 24 horas nas mesmas condições. O meio de cultura original em cada poço foi descartado e foram adicionados 100μL de meio de cultura contendo 10% de CCK‐8. A placa foi incubada em incubadora de células por 1 hora e a absorbância a 450nm foi medida utilizando leitor de microplacas. Os valores de absorbância (OD) foram medidos após 12, 24, 48 e 72 horas para avaliar a proliferação celular.
Ensaio TranswellTranswell revestido com Matrigel foi utilizado para realizar ensaios de invasão e migração celular para cada grupo. Foram adicionados 50μL de Matrigel (2mg/mL) à câmara superior de transwell e incubados a 37°C por 30 minutos. Em seguida, 4 × 103 células foram semeadas em meio DMEM isento de soro. A câmara inferior foi preenchida com 600μL de meio contendo 10% de soro fetal bovino como quimioatraente. Após incubação por 24 horas, as células não invasoras foram removidas com um cotonete. As células invadidas na membrana inferior foram fixadas com paraformaldeído a 4% por 30 minutos à temperatura ambiente e coradas com cristal violeta a 0,1% por 20 minutos. As células invadidas e migradas foram fotografadas e contadas ao microscópio (Carl Zeiss, Alemanha). Três campos aleatórios foram selecionados para contagem de células e a média foi calculada.
Ensaio cell scratchAs células da fase logarítmica foram colhidas, centrifugadas e ressuspensas em meio de cultura. As células foram contadas e cada grupo de células foi semeado em uma placa de seis poços (6,0 × 105 células/poço) para cultura. Quando a confluência celular atingiu aproximadamente 95% no dia seguinte, um arranhão foi feito no centro de cada poço usando uma ponta de pipeta de 200μL. Após lavagem com PBS, foi adicionado meio de cultura fresco. A imagem da hora 0 foi obtida utilizando um microscópio óptico invertido e as células foram adicionalmente incubadas. As imagens foram obtidas após 24 e 48 horas. A taxa de cicatrização foi calculada usando o software ImageJ.
Análise de citometria de fluxo de apoptose celular usando coloração dupla de anexina V/iodeto de propídio (IP)O sobrenadante da cultura foi coletado de cada grupo de células e as células foram lavadas com PBS, tripsinizadas e coletadas. As células foram duplamente coradas utilizando o Annexin V‐FITC Apoptosis Detection Kit. A coloração foi realizada em câmara escura à temperatura ambiente por 10 minutos. A citometria de fluxo foi utilizada para análise quantitativa e a taxa de apoptose foi calculada.
Análise do ciclo celularAs linhagens celulares de CECC humanos A431 e SCL‐1 foram mantidas em meio de cultura DMEM suplementado com 5% de FBS em placa de cultura de 10cm. Após tratamento nas condições especificadas, as células foram coletadas e suspensas em PBS e fixadas em etanol frio a 70% durante a noite a 4°C. As células foram então incubadas com 50μg/mL de iodeto de propídio (IP) e 100μg/mL de RNase A (1:1) por 20 minutos em temperatura ambiente no escuro. Por fim, o sinal de coloração das células CECC foi avaliado utilizando o citômetro de fluxo FACSCalibur™ (BD Biosciences, Franklin Lakes, New Jersey, EUA), e a distribuição das porcentagens do ciclo celular foi analisada utilizando o software Cell Quest versão 3.3 (BD Biosciences) de acordo com as instruções do fabricante.
Análise estatísticaA análise estatística foi realizada utilizando o software SPSS 23.0. Os dados quantitativos foram expressos como média ± desvio padrão. O teste de Qui‐Quadrado foi utilizado para comparar dados categóricos e análise de variância (ANOVA) foi utilizada para comparar taxas positivas entre múltiplos grupos. Testes t de amostras independentes foram usados para comparações pareadas. O valor de p<0,05 foi considerado estatisticamente significante.
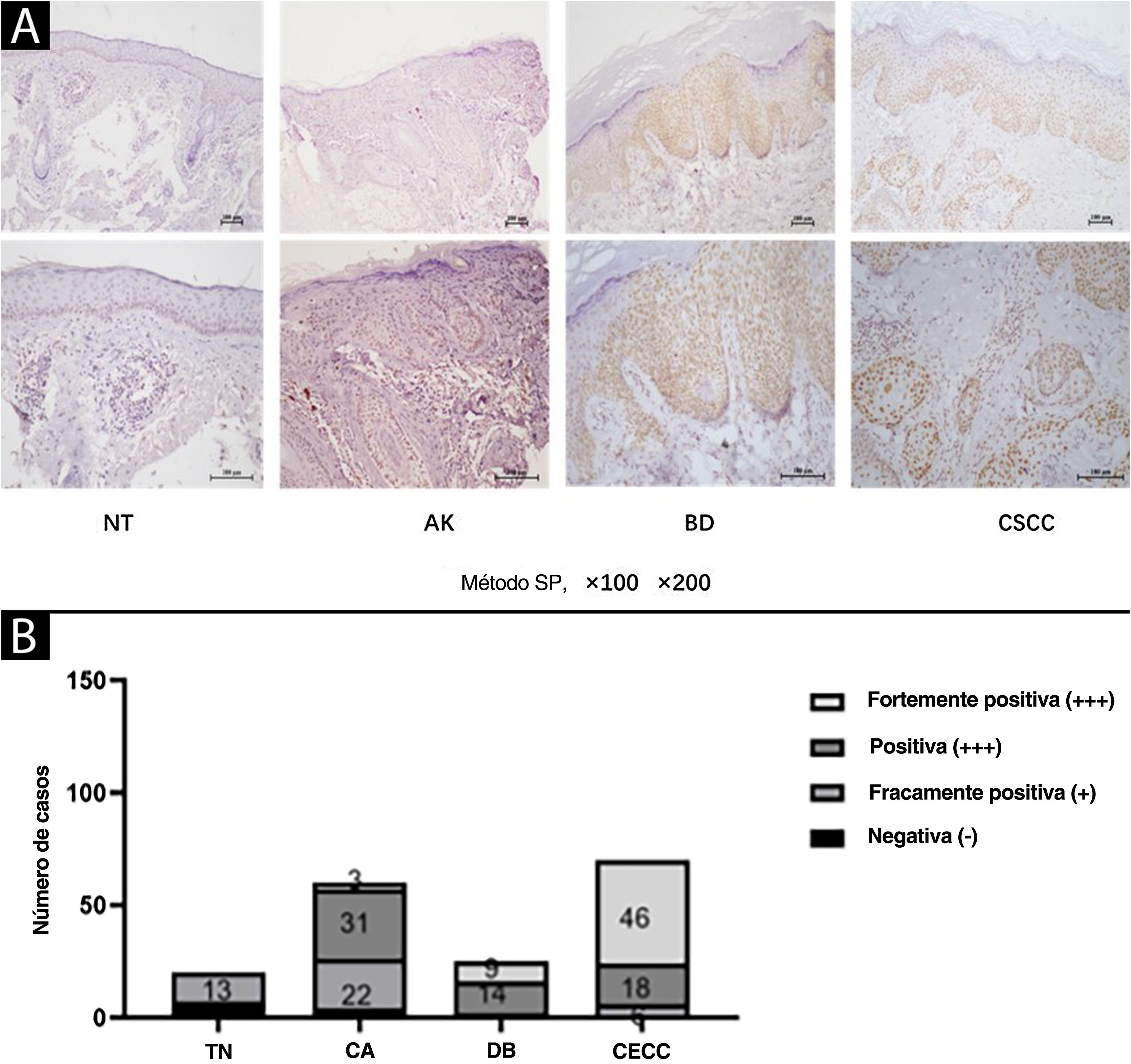
ResultadosExpressão elevada de SIRT6 em tecidos de QA, DB e CECC em comparação com tecido cutâneo normalA expressão de SIRT6 em diferentes tecidos foi analisada por marcação imuno‐histoquímica. Os resultados revelaram que a expressão da proteína SIRT6 era predominantemente observada no núcleo celular, exibindo uma gama de tonalidades desde amarelo claro até coloração marrom‐acastanhada, enquanto nenhuma expressão significante foi observada no citoplasma (fig. 1A).
Expressão de SIRT6 em tecido cutâneo normal (TN), ceratose actínica (CA), doença de Bowen (DB) e carcinoma espinocelular cutâneo (CECC). (A) Imagens representativas (ampliadas por 100× e 200×) da marcação imuno‐histoquímica para SIRT6 em tecidos TN, CA, DB e CECC usando o método SP. (B) Gráfico de barras ilustrando a intensidade da expressão de SIRT6: n(TN)=20 amostras (0 amostras fortemente positivas, 0 amostras positivas, 13 amostras fracamente positivas e sete amostras negativas); n(CA)=60 amostras (três amostras fortemente positivas, 31 amostras positivas, 22 amostras fracamente positivas e quatro amostras negativas); n(DB)=25 amostras (nove amostras fortemente positivas, 14 amostras positivas, duas amostras fracamente positivas e 0 amostras negativas); n(CECC)=70 amostras (46 amostras fortemente positivas, 18 amostras positivas, seis amostras fracamente positivas e 0 amostras negativas). Nota: O nome completo do método SP é método conjugado estreptomicina peroxidase.
Uma avaliação quantitativa dos níveis de expressão foi realizada utilizando sistema de escore semiquantitativo. Os resultados demonstraram a seguinte distribuição nos tecidos examinados: em 20 amostras de tecido cutâneo normal (TN), não houve expressão de SIRT6 fortemente positiva, positiva, fracamente positiva ou negativa; em 60 amostras de tecido de QA, a distribuição foi de três amostras, 31 amostras, 22 amostras e quatro amostras, respectivamente; em 25 amostras de tecido de DB, a distribuição foi de nove amostras, 14 amostras, duas amostras e 0 amostras, respectivamente; e entre 70 amostras de tecido de CECC, houve 46 amostras, 18 amostras, seis amostras e 0 amostras, respectivamente (fig. 1B). As taxas de expressão elevada de SIRT6 nos tecidos de TN, QA, DB e CECC foram 0%, 56,67%, 92,00% e 91,43%, respectivamente (tabela 3). Comparações pareadas revelaram maior expressão estatisticamente significante de SIRT6 em tecidos de QA, DB e CECC em comparação com TN (p<0,05), enquanto não houve diferença estatisticamente significante na expressão de SIRT6 entre tecidos de DB e CECC.
Diferenças na expressão de SIRT6 em tecidos TN, CA, DB e CECC
| Grupo | Expressão of SIRT6 | Total | Taxa de alta expressão | |
|---|---|---|---|---|
| Baixa expressão (−∼+) | Alta expressão (++∼+++) | |||
| TN | 20 | 0 | 20 | 0 |
| CA | 26 | 34 | 60 | 56,67% |
| DB | 2 | 23 | 25 | 92,00% |
| CECC | 6 | 64 | 70 | 91,43% |
TN, tecido cutâneo normal; CA, ceratose actínica; DB, doença de Bowen; CECC, carcinoma espinocelular cutâneo.
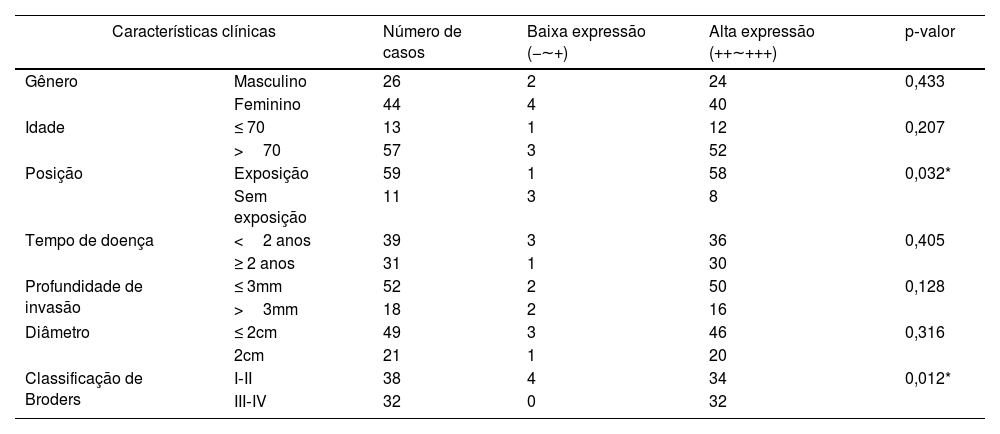
A análise da correlação entre a expressão de SIRT6 e parâmetros clínico‐patológicos em pacientes com CECC revelou que a elevada expressão de SIRT6 estava associada a tecido neoplásico localizado em áreas expostas ao sol (p<0,05) e maior grau na classificação Broders (p<0,05). Entretanto, não foram observadas correlações significantes entre a expressão de SIRT6 e dados clínico‐patológicos, como gênero, idade, duração da doença, profundidade de invasão e diâmetro do tumor (p>0,05). A tabela 4 mostra detalhes adicionais.
Relação entre a expressão de SIRT6 e características clínicas de pacientes com CECC
| Características clínicas | Número de casos | Baixa expressão (−∼+) | Alta expressão (++∼+++) | p‐valor | |
|---|---|---|---|---|---|
| Gênero | Masculino | 26 | 2 | 24 | 0,433 |
| Feminino | 44 | 4 | 40 | ||
| Idade | ≤ 70 | 13 | 1 | 12 | 0,207 |
| >70 | 57 | 3 | 52 | ||
| Posição | Exposição | 59 | 1 | 58 | 0,032* |
| Sem exposição | 11 | 3 | 8 | ||
| Tempo de doença | <2 anos | 39 | 3 | 36 | 0,405 |
| ≥ 2 anos | 31 | 1 | 30 | ||
| Profundidade de invasão | ≤ 3mm | 52 | 2 | 50 | 0,128 |
| >3mm | 18 | 2 | 16 | ||
| Diâmetro | ≤ 2cm | 49 | 3 | 46 | 0,316 |
| 2cm | 21 | 1 | 20 | ||
| Classificação de Broders | I‐II | 38 | 4 | 34 | 0,012* |
| III‐IV | 32 | 0 | 32 | ||
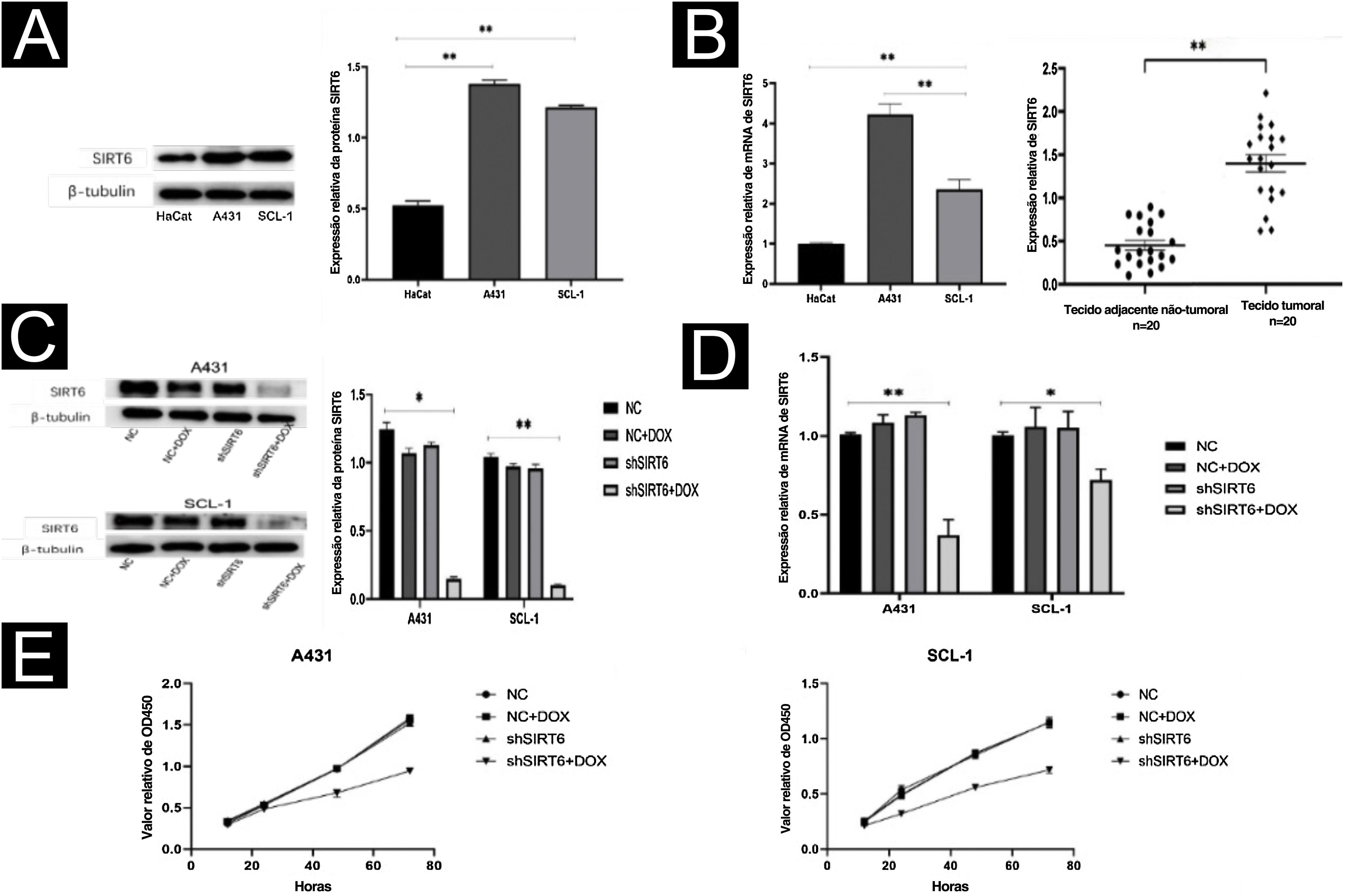
Pela análise WB e qRT‐PCR, observou‐se que o nível de expressão da proteína SIRT6 foi maior nas linhagens celulares de CECC (A431, SCL‐1) em comparação aos queratinócitos humanos imortalizados (HaCaT; fig. 2A). Do mesmo modo, o nível de expressão de mRNA de SIRT6 foi maior nas células A431 e SCL‐1 do que nas células HaCaT. A análise qRT‐PCR de 20 pares de CECC e tecidos adjacentes normais confirmou a alta expressão de SIRT6 em tecidos de CECC (fig. 2B), consistente com os resultados da análise imuno‐histoquímica. Para investigar melhor o papel funcional da SIRT6 no CECC, linhagens celulares de CECC com silenciamento do gene SIRT6 foram estabelecidas por transdução lentiviral. Controle negativo (CN), grupo CN+DOX e grupo controle shSIRT6 foram incluídos como CN. A eficiência do silenciamento de SIRT6 em células A431 e SCL‐1 foi validada por análise de Western blot (fig. 2C) e qRT‐PCR (fig. 2D), demonstrando regulação negativa da proteína SIRT6 e expressão de mRNA no grupo shSIRT6 em comparação com o grupo controle. Experimentos subsequentes foram realizados utilizando linhagens celulares silenciadas e grupos controle. Os resultados do ensaio CCK‐8 mostraram que o silenciamento de SIRT6 inibiu significantemente a proliferação de células A431 e SCL‐1 (fig. 2E).
O silenciamento de SIRT6 inibiu a proliferação de células de CECC. (A) Análise de Western blot da expressão da proteína SIRT6 em células A431, SCL‐1 e HaCaT. (B) Análise qRT‐PCR da expressão de mRNA de SIRT6 em células A431, SCL‐1 e HaCaT, bem como em 20 amostras de CECC e pele normal adjacente. (C‐D) Validação dos efeitos de silenciamento da proteína e mRNA de SIRT6 em células A431 e SCL‐1. (E) Ensaio CCK‐8 de proliferação de células de CECC, medindo a absorbância a 450nm em 12, 24, 48 e 72 horas; *p<0,05, **p<0,01.
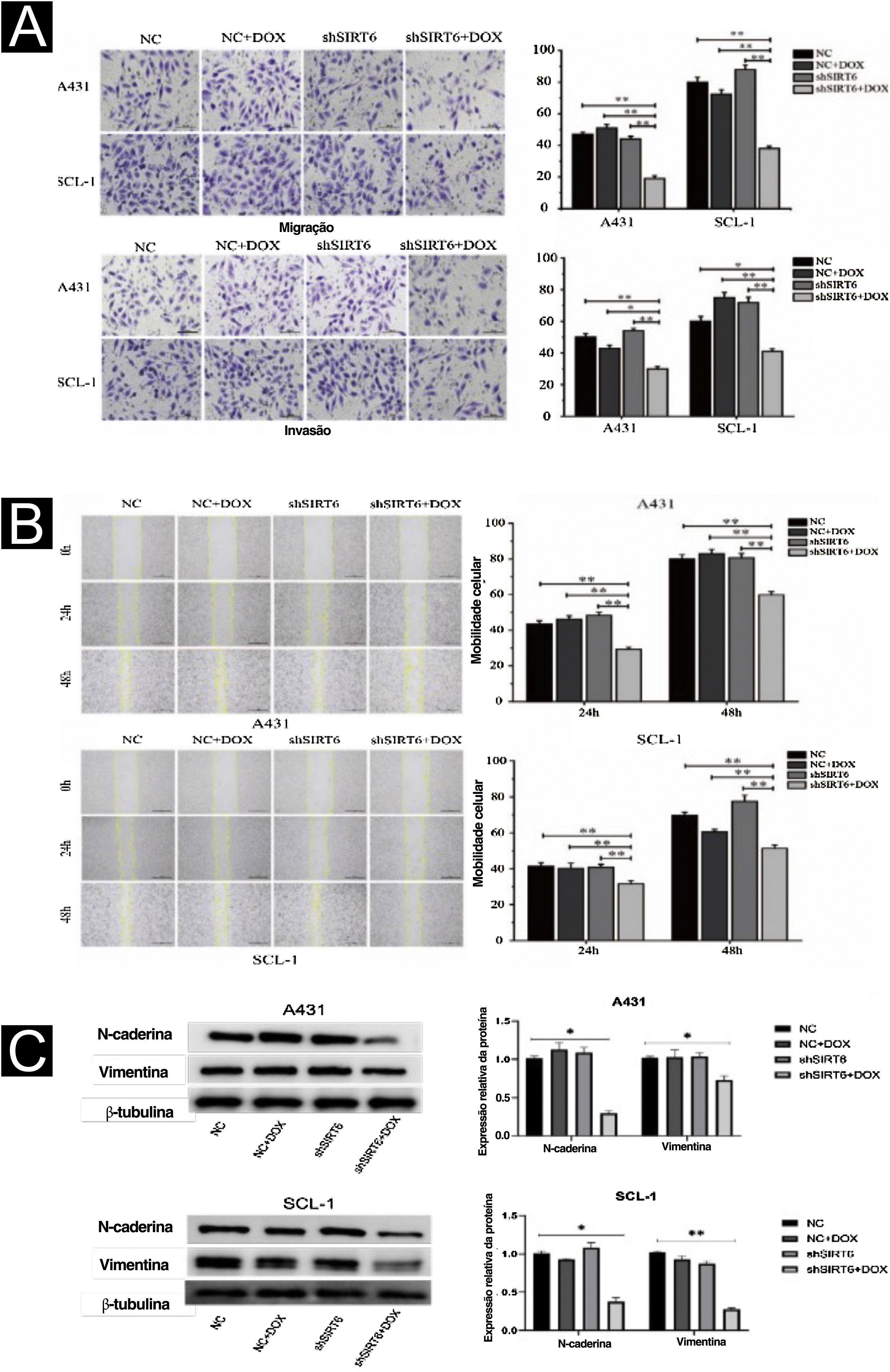
Os efeitos de SIRT6 na migração e invasão de células CECC foram investigados posteriormente utilizando o ensaio Transwell e o ensaio scratch. Os resultados mostraram que, em comparação com os grupos controle (CN, CN + DOX e controle shSIRT6), o grupo silenciado shSIRT6 + DOX exibiu migração e invasão celular reduzidas (fig. 3A) e taxa de cicatrização de feridas reduzida (fig. 3B) em células A431 e SCL‐1. Esses resultados indicam que o silenciamento de SIRT6 inibe significantemente as capacidades de migração e invasão das células de CECC. A análise de Western blot foi realizada para detectar a expressão de marcadores de transição epitelial‐mesenquimal (TEM) em células de CECC. Em comparação com os grupos de controle (controle CN, CN + DOX e shSIRT6), o grupo shSIRT6 + DOX de células A431 e SCL‐1 apresentou expressão regulada negativamente dos marcadores mesenquimais N‐caderina e vimentina (fig. 3C). Esses resultados indicam que o silenciamento de SIRT6 promove a expressão dos marcadores de transição epitelial‐mesenquimal N‐caderina e vimentina em células CECC, sugerindo que a inibição da migração e invasão de células CECC por silenciamento de SIRT6 possa estar associada à supressão do processo TEM.
Inibição da migração e invasão de células de CECC por silenciamento de SIRT6. (A) Resultados do ensaio Transwell e análise estatística representando os efeitos da SIRT6 na migração e invasão celular em células A431 e SCL‐1. Comparação do número de células migradas e invadidas nos grupos SIRT6+DOX, NC, NC+DOX e grupo controle shSIRT6. (B) Resultados do ensaio scratch e análise estatística demonstrando os efeitos da SIRT6 na taxa de migração celular em células A431 e SCL‐1. Comparação da taxa de migração em 24 e 48 horas nos grupos SIRT6+DOX, NC, NC+DOX e grupo shSIRT6. (C) Resultados da análise de Western blot e análise estatística representando os efeitos da SIRT6 na expressão de marcadores de transição epitelial‐mesenquimal (EMT) em células de CECC. Comparação dos níveis de expressão dos marcadores mesenquimais N‐caderina e vimentina em células A431 e SCL‐1. Nota: *p<0,05, **p<0,01.
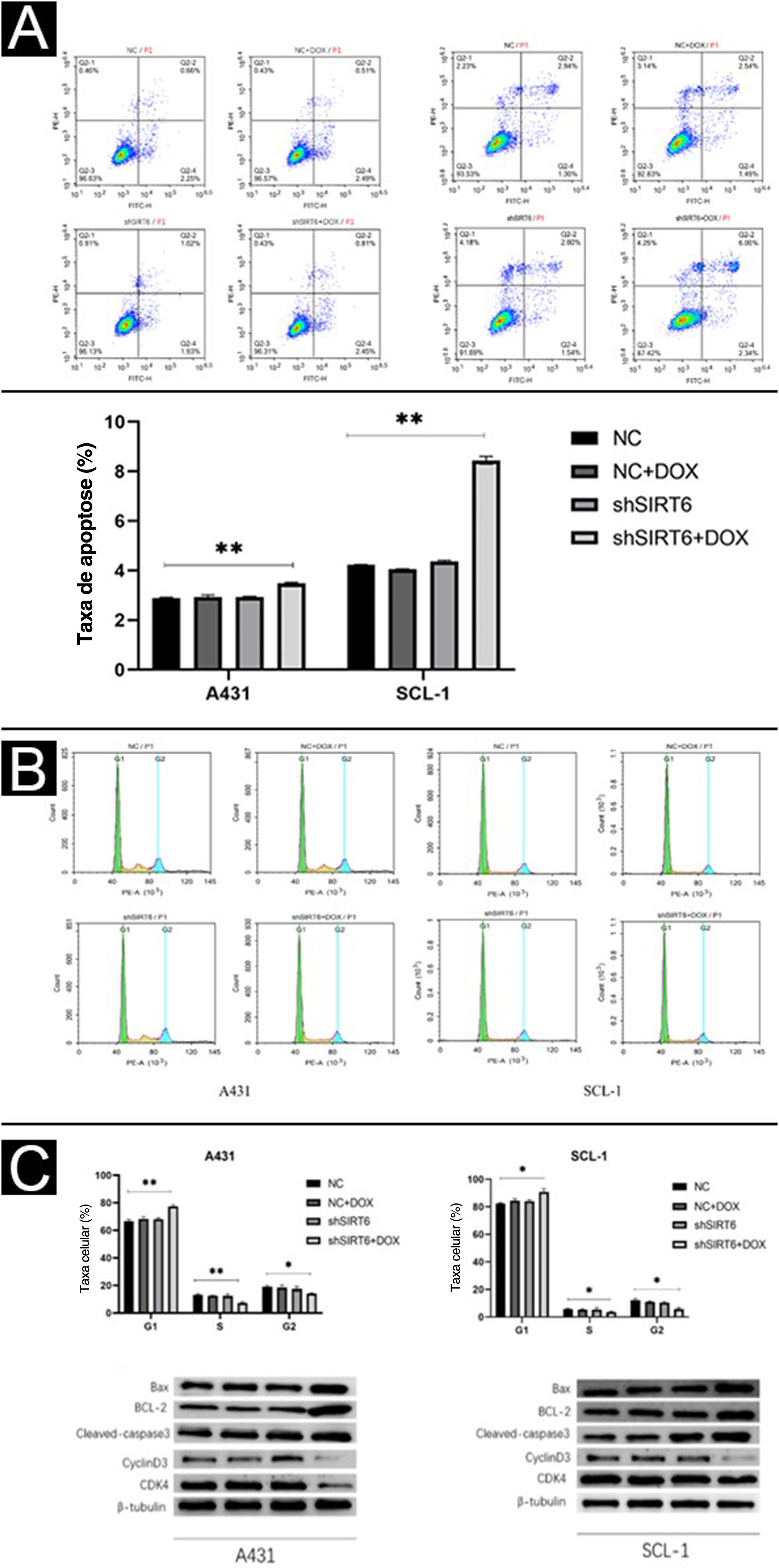
Utilizando citometria de fluxo, foi investigado também o efeito da SIRT6 na apoptose e na progressão do ciclo celular em células de CECC. Os resultados mostram que os grupos de células A431 e SCL‐1 tratados com shSIRT6 + DOX exibem taxas apoptóticas aumentadas (fig. 4A) e bloqueio na fase G0/G1 do ciclo celular (fig. 4B), indicando que a regulação negativa de SIRT6 pode aumentar a apoptose e induzir a parada do ciclo celular na fase G0/G1 em células de CECC. Para avaliar a expressão de marcadores relacionados à apoptose (Bax, BCL‐2, caspase‐3 clivada) e marcadores relacionados ao ciclo celular (CyclinD1, CDK4) em células de CECC, foi realizada análise por Western blot. Em comparação com os grupos de controle CN, CN + DOX e shSIRT6, os grupos de células A431 e SCL‐1 tratados com shSIRT6 + DOX exibiram níveis aumentados de expressão da proteína Bax, BCL‐2 e caspase‐3 clivada, enquanto os níveis de proteína CyclinD1 e CDK4 estavam diminuídos (fig. 4C). Esses resultados indicam que a regulação negativa da SIRT6 promove a apoptose e induz a parada do ciclo celular na fase G0/G1 em células de CECC.
O silenciamento de SIRT6 pode aumentar a apoptose e a parada do ciclo celular na fase G0/G1 das células de CECC. (A‐B) A análise de citometria de fluxo mostrou a taxa de apoptose e a distribuição do ciclo celular de células A431 e SCL‐1 transfectadas com shSIRT6+DOX e NC, NC+DOX e shSIRT6 no grupo controle. A taxa de apoptose foi medida por citometria de fluxo. O quadrante superior direito representava células apoptóticas tardias, e o quadrante inferior direito representava células apoptóticas precoces. A taxa de apoptose=razão apoptótica tardia + razão apoptótica precoce. (C) Western blots mostraram proteínas relacionadas à apoptose (Bax, BCL‐2, Cleaved‐caspase3) e ciclinas (ciclinaD1 e CDK4) em células A431 e SCL‐1 transfectadas com shSIRT6+DOX e controle NC, NC+DOX, shSIRT6 Flat, *p<0,05, **p<0,01.
O CECC é neoplasia maligna proliferativa originada das células queratinizadas dos anexos ou da epiderme. Embora a maioria dos CECC possa ser erradicada com sucesso por meio de excisão cirúrgica, aproximadamente 14% a 15% dos pacientes com CECC primário ainda apresentam infiltração subclínica.22 O crescimento invasivo do CECC pode romper a membrana basal da epiderme e invadir a derme mais profunda, anexos da pele, vasos sanguíneos, vasos linfáticos, nervos etc., levando ao desenvolvimento do CECC de alto risco. Apesar dos avanços significantes nas opções de tratamento, como cirurgia de Mohs, cetuximabe e panitumumabe, que melhoraram muito o prognóstico dos pacientes com CECC, o prognóstico para CECC de alto e muito alto risco permanece ruim, com taxas de recorrência em cinco anos variando de 30% a 50%.23 Portanto, é necessário explorar ainda mais os mecanismos subjacentes à ocorrência e progressão do CECC e identificar mais alvos terapêuticos.
A SIRT6 está desregulada em vários tipos de câncer, e seu papel na tumorigênese depende do tipo e do contexto do tumor. A expressão de SIRT6 é regulada positivamente no câncer de mama, câncer de pâncreas, melanoma primário e carcinoma de pulmão de células não pequenas,24,25 sugerindo papel pró‐oncogênico nesses tipos de câncer. Por outro lado, a SIRT6 é regulada negativamente em tecidos de câncer de ovário, e sua baixa expressão está intimamente associada à infiltração neoplásica e metástases.26 Estudos demonstraram que a SIRT6 inibe o crescimento do câncer gástrico ao suprimir o transdutor de sinal/JAK2 e o ativador da via da transcrição‐3.27 Além disso, foi relatado que SIRT6 pode suprimir a ocorrência e o desenvolvimento de câncer pancreático.28 Portanto, a SIRT6 também desempenha um papel na inibição do início e desenvolvimento de neoplasias malignas, com forte heterogeneidade em diferentes tipos de tumores. Os resultados deste estudo mostram que a expressão de SIRT6 está aumentada em tecidos tumorais de CECC em comparação com tecidos cutâneos normais, sugerindo que a alta expressão de SIRT6 possa estar envolvida na ocorrência e desenvolvimento do CECC. O nível de expressão da proteína SIRT6 no CECC é maior do que na lesão pré‐cancerosa de CA e tecido cutâneo normal, sugerindo que a SIRT6 possa desempenhar papel importante na progressão do tecido cutâneo normal para lesões pré‐cancerosas e posteriormente para carcinoma espinocelular cutâneo. No entanto, a expressão de SIRT6 foi ligeiramente maior na DB do que no tecido de CECC, o que pode estar relacionado ao número limitado de amostras de tecido de DB. A alta expressão de SIRT6 em tecidos de CECC está associada à localização do tecido neoplásico em áreas expostas ao sol e ao estadiamento de Broders, mas não ao gênero, idade, diâmetro do tumor etc., consistente com achados em melanoma e carcinoma de pulmão de células não pequenas.29 Além disso, descobriu‐se que a SIRT6 é altamente expressa em células de CECC e que o silenciamento da SIRT6 inibe a capacidade de proliferação, migração e invasão das células de CECC. A invasão e a migração são características críticas das neoplasias malignas, e o silenciamento da SIRT6 reduziu significantemente a proliferação, migração e invasão de células de CECC, fornecendo evidências do papel da SIRT6 no crescimento e motilidade das células de CECC.
O silenciamento de SIRT6 induz a apoptose celular e inibe a transição do ciclo celular G1/S. A apoptose celular é processo complexo que envolve múltiplas vias de sinalização, e a regulação positiva de proteínas relacionadas à apoptose (incluindo Bax, BCL‐2, caspase‐3 clivada) após o silenciamento de SIRT6 confirma seu envolvimento na regulação da apoptose do CECC. Também foi observado que o silenciamento de SIRT6 leva à parada do ciclo celular G0/G1, como demonstrado pela análise do ciclo celular. Além disso, a regulação negativa de ciclina D1 e CDK4, que codificam proteínas que regulam o ciclo celular, está associada à progressão do tumor.30 A ciclina D1 é proteína‐chave na regulação do ciclo celular e é suficiente para impulsionar a progressão desse ciclo.31,32 Este estudo demonstra o envolvimento da SIRT6 na regulação da apoptose e do ciclo celular em células de CECC, influenciando assim os níveis de expressão de apoptose e proteínas relacionadas ao ciclo celular. Com base nisso, especula‐se que a SIRT6 possa estar envolvida no comportamento biológico das células do CECC como uma molécula funcional. Entretanto, existem várias limitações neste estudo. Primeiro, as amostras clínicas de tecidos do estudo são limitadas; estudos futuros devem aumentar o tamanho da amostragem e utilizar múltiplos métodos experimentais para esclarecer ainda mais o padrão de expressão de SIRT6. Em segundo lugar, em virtude da complexidade da regulação e desenvolvimento tumoral, os mecanismos específicos ainda estão sujeitos a investigação adicional, e experimentos subsequentes in vivo devem ser realizados para explorar o impacto da SIRT6 no comportamento biológico do CECC. Além disso, as neoplasias são doenças complexas com elevada heterogeneidade, e sua ocorrência e desenvolvimento não são determinados por um único fator independente. Portanto, a identificação no CECC de moléculas que possam interagir sinergicamente ou antagonicamente com a SIRT6 continua sendo um foco para pesquisas futuras.
ConclusãoEste estudo descobriu que a SIRT6 é altamente expressa no CECC, o que pode desempenhar papel como fator cancerígeno, fornecendo certa base teórica para tratamentos futuros.
Suporte financeiroEste trabalho recebeu suporte financeiro da Nantong Municipal Health Commission (MB2021019), Basic Research Project of Nantong Science e Technology Bureau (JCZ2022021), e Natural Science Foundation of Jiangsu Province (BK20211108).
Contribuição dos autoresChen Sai: Concepção e planejamento do estudo; obtenção, análise e interpretação dos dados; análise estatística; elaboração e redação do manuscrito; revisão crítica do manuscrito; aprovação da versão final do manuscrito.
Chen Hongxia: Obtenção, análise e interpretação dos dados; análise estatística; elaboração e redação do manuscrito; revisão crítica do manuscrito; aprovação da versão final do manuscrito.
Wang Xu: Obtenção, análise e interpretação dos dados; análise estatística; elaboração e redação do manuscrito; revisão crítica do manuscrito; aprovação da versão final do manuscrito.
Zhang Dongmei: Participação efetiva na orientação da pesquisa; revisão crítica da literatura; aprovação da versão final do manuscrito.
Zhang Li: Obtenção, análise e interpretação dos dados; revisão crítica da literatura; aprovação da versão final do manuscrito.
Jiawei Cheng: Obtenção, análise e interpretação dos dados; revisão crítica da literatura; aprovação da versão final do manuscrito.
Zhang Qi: Obtenção, análise e interpretação dos dados; revisão crítica da literatura; aprovação da versão final do manuscrito.
Zhixiang Hua: Participação efetiva na orientação da pesquisa; revisão crítica da literatura; aprovação da versão final do manuscrito.
Miao Xu: Participação efetiva na orientação da pesquisa; revisão crítica da literatura; aprovação da versão final do manuscrito.
Shi Jian: Participação efetiva na orientação da pesquisa; obtenção, análise e interpretação dos dados; análise estatística; elaboração e redação do manuscrito; revisão crítica do manuscrito; aprovação da versão final do manuscrito.
Conflito de interessesNenhum.
Os autores deste artigo são muito gratos à Nantong Municipal Health Commission (MB2021019), ao Nantong Municipal Science and Technology Bureau (JCZ2022021) e à Natural Science Foundation of Jiangsu Province (BK20211108) por financiar o projeto.
Como citar este artigo: Chen S, Chen H, Wang X, Zhang D, Zhang L, Cheng J, et al. Expression analysis and biological regulation of silencing regulatory protein 6 (SIRT6) in cutaneous squamous cell carcinoma. An Bras Dermatol. 2024;99:535–45.
Trabalho realizado no Nantong First People's Hospital, Nantong, China.