Melasma é hipermelanose que afeta áreas fotoexpostas, especialmente em mulheres adultas, que inflige importante impacto na qualidade de vida por acometer áreas visíveis e ser recidivante, apesar dos tratamentos. Sua fisiopatologia não é totalmente compreendida, mas resulta da interação entre fatores de exposição (p. ex., radiação solar e hormônios sexuais) e predisposição genética. Diversos estímulos dérmicos foram apontados na manutenção da melanogênese no melasma, envolvendo atividade de fibroblastos, endotélio e mastócitos, que promovem elastonização do colágeno, dano estrutural à membrana basal, liberação de fatores de crescimento (p. ex., sSCF, bFGF, NGF, HGF) e mediadores inflamatórios (p. ex., ET1, IL1, VEGF, TGFb).1–3
Este estudo objetivou explorar proteínas diferencialmente expostas na pele com melasma em relação à pele adjacente fotoexposta, não afetada.
Realizou‐se estudo transversal envolvendo 20 mulheres com melasma facial, sem tratamentos específicos por 30 dias. Duas biópsias foram realizadas (mesmo pesquisador), uma no limite do melasma facial e outra na pele não afetada, a 2 cm de distância da primeira, conforme padronizado anteriormente.1,3 Realizou‐se a extração mecânica de proteínas, sua digestão enzimática e espectrometria de massa. O projeto foi aprovado pelo Comitê de Ética institucional (n° 1.411.931).
As amostras foram analisadas em duplicata no sistema nanoACQUITY‐UPLC acoplado a espectrômetro de massa Xevo‐Q‐TOF‐G2, cujos resultados foram processados no software ProteinLynx GlobalServer 3.03v. As proteínas foram indentificadas por meio do algoritmo de contagem de íons, cujos padrões espectrais foram pesquisados no banco Homo sapiens, no catálogo UniProt (https://www.uniprot.org/).
Todas as proteínas identificadas com > 95% de similaridade foram incluídas na análise. As intensidades dos picos de íons foram normalizadas, escalonadas e comparadas entre as topografias por um algoritmo bayesiano (método Monte Carlo), que retorna um valor p ≤ 0,05 para proteínas sub‐reguladas e ≥ 0,95 para as super‐reguladas, corrigido pelo procedimento de Benjamini‐Hochberg.4
O principal desfecho do estudo foi a diferença entre as intensidades dos picos iônicos das proteínas (melasma: M; perilesional: P). O tamanho do efeito foi estimado pela razão dessas quantidades entre as topografias (M/P). Proteínas com razão M/P ≤ 0,5 ou ≥ 2,0 foram consideradas neste estudo.
As proteínas identificadas e suas funções biológicas foram diagramadas em mapa térmico e agrupadas por meio de procedimento de cluster (método Ward).
A idade média (desvio‐padrão) das pacientes foi de 42,8 (8,9) anos; 70% eram dos fototipos III‐IV e 25% exerciam profissões expostas ao sol. A idade de início do melasma foi de 29,3 (7,5) anos; 55% das mulheres referiam histórico familiar e 30% usavam anticoncepcional.
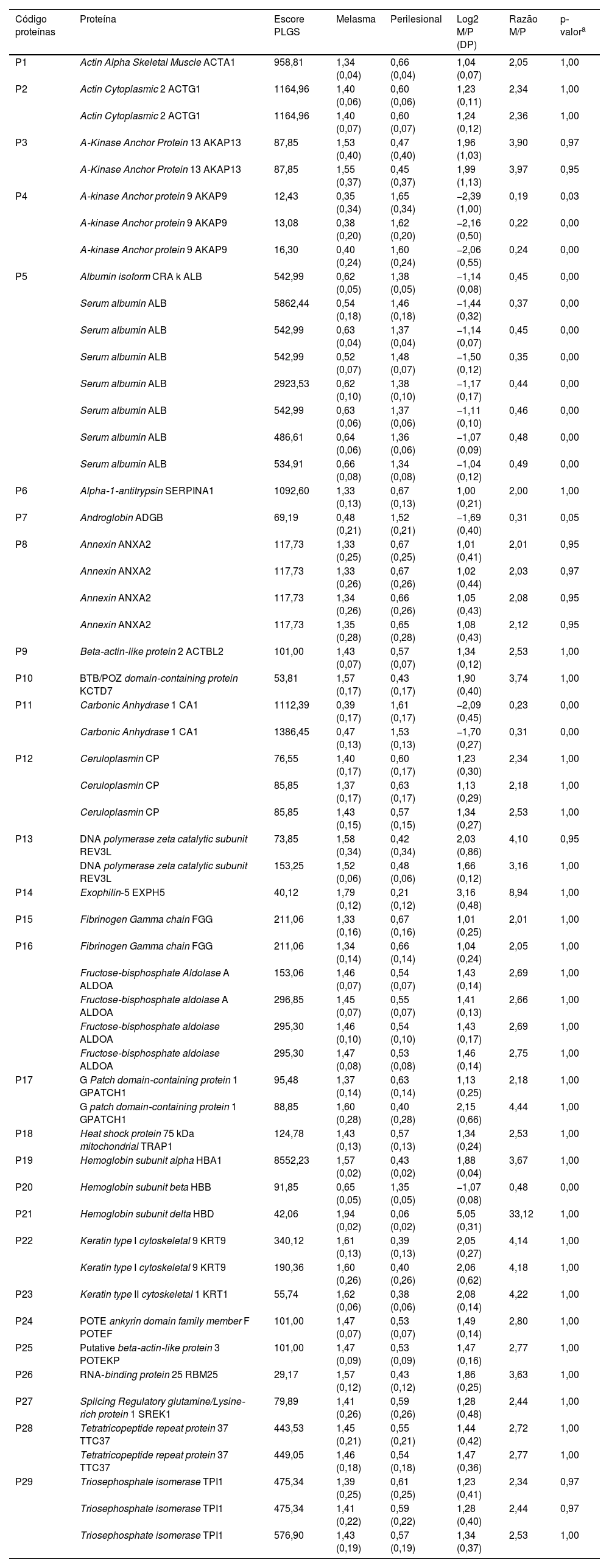
Validaram‐se 256 proteínas nas amostras de pele, e as 29 proteínas diferencialmente quantificadas entre as topografias estão expostas na tabela 1. As maiores discrepâncias ocorreram para as proteínas HBD, EXPH5, KRT1, KRT9, REV3L (M/S > 4,00); e ACAP9, ADGB, CA1 (M/S < 0,33).
Proteínas e isoformas identificadas nas amostras de peles de melasma facial (M) e adjacente fotoexposta (P) (n = 40) com diferença ente os grupos (p ≤ 0,05 ou ≥ 0,95) e razão M/P ≥ 2,0 ou ≤ 0,5
| Código proteínas | Proteína | Escore PLGS | Melasma | Perilesional | Log2 M/P (DP) | Razão M/P | p‐valora |
|---|---|---|---|---|---|---|---|
| P1 | Actin Alpha Skeletal Muscle ACTA1 | 958,81 | 1,34 (0,04) | 0,66 (0,04) | 1,04 (0,07) | 2,05 | 1,00 |
| P2 | Actin Cytoplasmic 2 ACTG1 | 1164,96 | 1,40 (0,06) | 0,60 (0,06) | 1,23 (0,11) | 2,34 | 1,00 |
| Actin Cytoplasmic 2 ACTG1 | 1164,96 | 1,40 (0,07) | 0,60 (0,07) | 1,24 (0,12) | 2,36 | 1,00 | |
| P3 | A‐Kinase Anchor Protein 13 AKAP13 | 87,85 | 1,53 (0,40) | 0,47 (0,40) | 1,96 (1,03) | 3,90 | 0,97 |
| A‐Kinase Anchor Protein 13 AKAP13 | 87,85 | 1,55 (0,37) | 0,45 (0,37) | 1,99 (1,13) | 3,97 | 0,95 | |
| P4 | A‐kinase Anchor protein 9 AKAP9 | 12,43 | 0,35 (0,34) | 1,65 (0,34) | −2,39 (1,00) | 0,19 | 0,03 |
| A‐kinase Anchor protein 9 AKAP9 | 13,08 | 0,38 (0,20) | 1,62 (0,20) | −2,16 (0,50) | 0,22 | 0,00 | |
| A‐kinase Anchor protein 9 AKAP9 | 16,30 | 0,40 (0,24) | 1,60 (0,24) | −2,06 (0,55) | 0,24 | 0,00 | |
| P5 | Albumin isoform CRA k ALB | 542,99 | 0,62 (0,05) | 1,38 (0,05) | −1,14 (0,08) | 0,45 | 0,00 |
| Serum albumin ALB | 5862,44 | 0,54 (0,18) | 1,46 (0,18) | −1,44 (0,32) | 0,37 | 0,00 | |
| Serum albumin ALB | 542,99 | 0,63 (0,04) | 1,37 (0,04) | −1,14 (0,07) | 0,45 | 0,00 | |
| Serum albumin ALB | 542,99 | 0,52 (0,07) | 1,48 (0,07) | −1,50 (0,12) | 0,35 | 0,00 | |
| Serum albumin ALB | 2923,53 | 0,62 (0,10) | 1,38 (0,10) | −1,17 (0,17) | 0,44 | 0,00 | |
| Serum albumin ALB | 542,99 | 0,63 (0,06) | 1,37 (0,06) | −1,11 (0,10) | 0,46 | 0,00 | |
| Serum albumin ALB | 486,61 | 0,64 (0,06) | 1,36 (0,06) | −1,07 (0,09) | 0,48 | 0,00 | |
| Serum albumin ALB | 534,91 | 0,66 (0,08) | 1,34 (0,08) | −1,04 (0,12) | 0,49 | 0,00 | |
| P6 | Alpha‐1‐antitrypsin SERPINA1 | 1092,60 | 1,33 (0,13) | 0,67 (0,13) | 1,00 (0,21) | 2,00 | 1,00 |
| P7 | Androglobin ADGB | 69,19 | 0,48 (0,21) | 1,52 (0,21) | −1,69 (0,40) | 0,31 | 0,05 |
| P8 | Annexin ANXA2 | 117,73 | 1,33 (0,25) | 0,67 (0,25) | 1,01 (0,41) | 2,01 | 0,95 |
| Annexin ANXA2 | 117,73 | 1,33 (0,26) | 0,67 (0,26) | 1,02 (0,44) | 2,03 | 0,97 | |
| Annexin ANXA2 | 117,73 | 1,34 (0,26) | 0,66 (0,26) | 1,05 (0,43) | 2,08 | 0,95 | |
| Annexin ANXA2 | 117,73 | 1,35 (0,28) | 0,65 (0,28) | 1,08 (0,43) | 2,12 | 0,95 | |
| P9 | Beta‐actin‐like protein 2 ACTBL2 | 101,00 | 1,43 (0,07) | 0,57 (0,07) | 1,34 (0,12) | 2,53 | 1,00 |
| P10 | BTB/POZ domain‐containing protein KCTD7 | 53,81 | 1,57 (0,17) | 0,43 (0,17) | 1,90 (0,40) | 3,74 | 1,00 |
| P11 | Carbonic Anhydrase 1 CA1 | 1112,39 | 0,39 (0,17) | 1,61 (0,17) | −2,09 (0,45) | 0,23 | 0,00 |
| Carbonic Anhydrase 1 CA1 | 1386,45 | 0,47 (0,13) | 1,53 (0,13) | −1,70 (0,27) | 0,31 | 0,00 | |
| P12 | Ceruloplasmin CP | 76,55 | 1,40 (0,17) | 0,60 (0,17) | 1,23 (0,30) | 2,34 | 1,00 |
| Ceruloplasmin CP | 85,85 | 1,37 (0,17) | 0,63 (0,17) | 1,13 (0,29) | 2,18 | 1,00 | |
| Ceruloplasmin CP | 85,85 | 1,43 (0,15) | 0,57 (0,15) | 1,34 (0,27) | 2,53 | 1,00 | |
| P13 | DNA polymerase zeta catalytic subunit REV3L | 73,85 | 1,58 (0,34) | 0,42 (0,34) | 2,03 (0,86) | 4,10 | 0,95 |
| DNA polymerase zeta catalytic subunit REV3L | 153,25 | 1,52 (0,06) | 0,48 (0,06) | 1,66 (0,12) | 3,16 | 1,00 | |
| P14 | Exophilin‐5 EXPH5 | 40,12 | 1,79 (0,12) | 0,21 (0,12) | 3,16 (0,48) | 8,94 | 1,00 |
| P15 | Fibrinogen Gamma chain FGG | 211,06 | 1,33 (0,16) | 0,67 (0,16) | 1,01 (0,25) | 2,01 | 1,00 |
| P16 | Fibrinogen Gamma chain FGG | 211,06 | 1,34 (0,14) | 0,66 (0,14) | 1,04 (0,24) | 2,05 | 1,00 |
| Fructose‐bisphosphate Aldolase A ALDOA | 153,06 | 1,46 (0,07) | 0,54 (0,07) | 1,43 (0,14) | 2,69 | 1,00 | |
| Fructose‐bisphosphate aldolase A ALDOA | 296,85 | 1,45 (0,07) | 0,55 (0,07) | 1,41 (0,13) | 2,66 | 1,00 | |
| Fructose‐bisphosphate aldolase ALDOA | 295,30 | 1,46 (0,10) | 0,54 (0,10) | 1,43 (0,17) | 2,69 | 1,00 | |
| Fructose‐bisphosphate aldolase ALDOA | 295,30 | 1,47 (0,08) | 0,53 (0,08) | 1,46 (0,14) | 2,75 | 1,00 | |
| P17 | G Patch domain‐containing protein 1 GPATCH1 | 95,48 | 1,37 (0,14) | 0,63 (0,14) | 1,13 (0,25) | 2,18 | 1,00 |
| G patch domain‐containing protein 1 GPATCH1 | 88,85 | 1,60 (0,28) | 0,40 (0,28) | 2,15 (0,66) | 4,44 | 1,00 | |
| P18 | Heat shock protein 75 kDa mitochondrial TRAP1 | 124,78 | 1,43 (0,13) | 0,57 (0,13) | 1,34 (0,24) | 2,53 | 1,00 |
| P19 | Hemoglobin subunit alpha HBA1 | 8552,23 | 1,57 (0,02) | 0,43 (0,02) | 1,88 (0,04) | 3,67 | 1,00 |
| P20 | Hemoglobin subunit beta HBB | 91,85 | 0,65 (0,05) | 1,35 (0,05) | −1,07 (0,08) | 0,48 | 0,00 |
| P21 | Hemoglobin subunit delta HBD | 42,06 | 1,94 (0,02) | 0,06 (0,02) | 5,05 (0,31) | 33,12 | 1,00 |
| P22 | Keratin type I cytoskeletal 9 KRT9 | 340,12 | 1,61 (0,13) | 0,39 (0,13) | 2,05 (0,27) | 4,14 | 1,00 |
| Keratin type I cytoskeletal 9 KRT9 | 190,36 | 1,60 (0,26) | 0,40 (0,26) | 2,06 (0,62) | 4,18 | 1,00 | |
| P23 | Keratin type II cytoskeletal 1 KRT1 | 55,74 | 1,62 (0,06) | 0,38 (0,06) | 2,08 (0,14) | 4,22 | 1,00 |
| P24 | POTE ankyrin domain family member F POTEF | 101,00 | 1,47 (0,07) | 0,53 (0,07) | 1,49 (0,14) | 2,80 | 1,00 |
| P25 | Putative beta‐actin‐like protein 3 POTEKP | 101,00 | 1,47 (0,09) | 0,53 (0,09) | 1,47 (0,16) | 2,77 | 1,00 |
| P26 | RNA‐binding protein 25 RBM25 | 29,17 | 1,57 (0,12) | 0,43 (0,12) | 1,86 (0,25) | 3,63 | 1,00 |
| P27 | Splicing Regulatory glutamine/Lysine‐rich protein 1 SREK1 | 79,89 | 1,41 (0,26) | 0,59 (0,26) | 1,28 (0,48) | 2,44 | 1,00 |
| P28 | Tetratricopeptide repeat protein 37 TTC37 | 443,53 | 1,45 (0,21) | 0,55 (0,21) | 1,44 (0,42) | 2,72 | 1,00 |
| Tetratricopeptide repeat protein 37 TTC37 | 449,05 | 1,46 (0,18) | 0,54 (0,18) | 1,47 (0,36) | 2,77 | 1,00 | |
| P29 | Triosephosphate isomerase TPI1 | 475,34 | 1,39 (0,25) | 0,61 (0,25) | 1,23 (0,41) | 2,34 | 0,97 |
| Triosephosphate isomerase TPI1 | 475,34 | 1,41 (0,22) | 0,59 (0,22) | 1,28 (0,40) | 2,44 | 0,97 | |
| Triosephosphate isomerase TPI1 | 576,90 | 1,43 (0,19) | 0,57 (0,19) | 1,34 (0,37) | 2,53 | 1,00 |
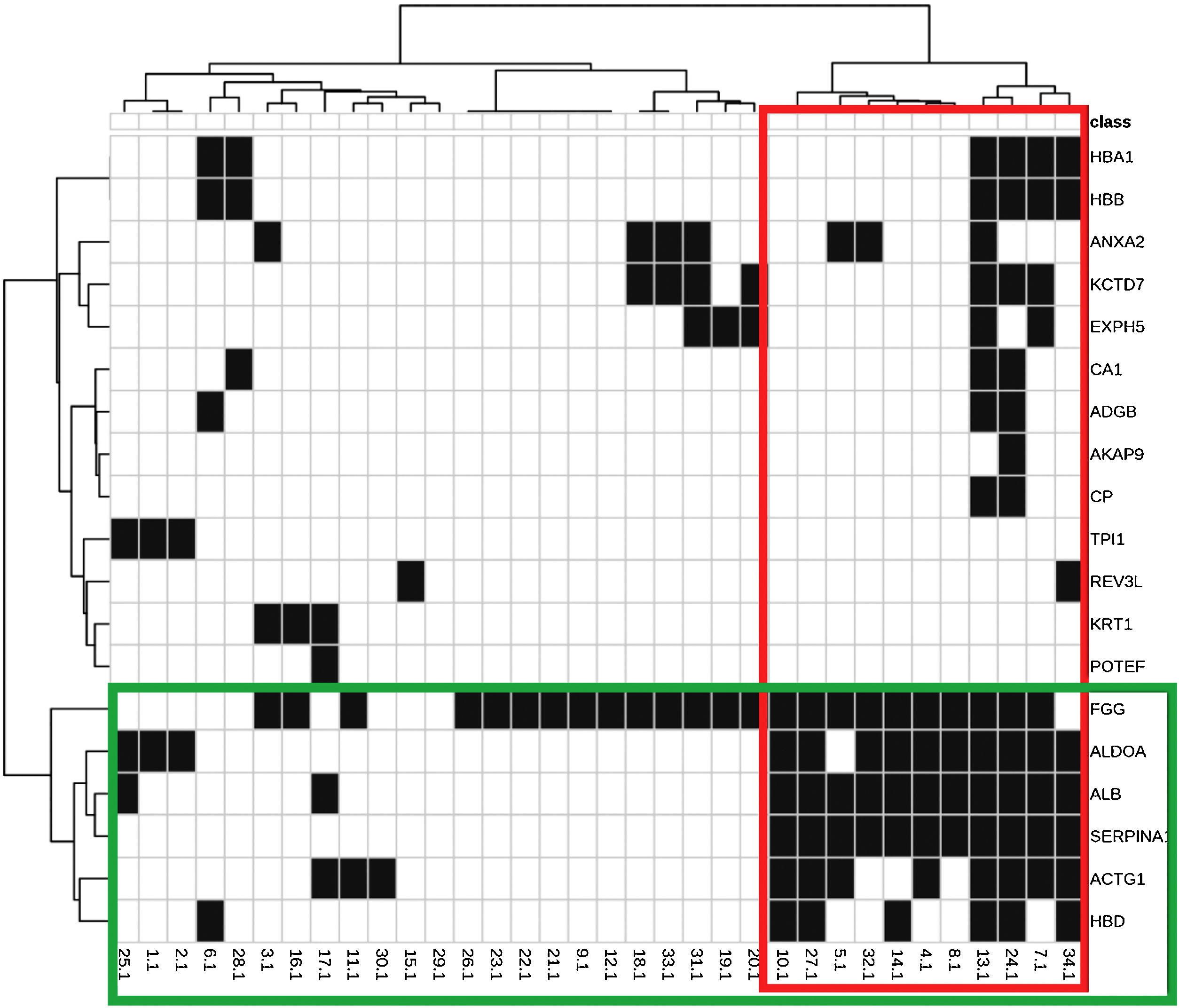
As principais funções biológicas dessas proteínas estão dispostas na tabela 2. A figura 1 representa a interação entre as 29 proteínas e suas funções biológicas. As proteínas ACTG1, ALB, SERPINA1, HBD, ALDOA e FGG mostraram‐se coparticipantes em diferentes processos biológicos, como consumo de oxigênio, glicólise, gliconeogênese e transporte celular, sugerindo aumento da atividade metabólica da pele com melasma.
Principais vias funcionais envolvidas com as 29 proteínas identificadas como diferenciais entre melasma e pele perilesional
| Funções | Proteínas envolvidas | n (%) | FDRa |
|---|---|---|---|
| 1. Glicólise canônica | p16, p29 | 2 (7) | < 0,0001 |
| 2. Gluconeogênese | p16, p29 | 2 (7) | < 0,0001 |
| 3. Fibrinólise | p8, p15, p23 | 2 (10) | < 0,0001 |
| 4. Degranulação plaquetária | p2, p5, p6, p15, p16 | 5 (17) | < 0,0001 |
| 5. Regulação de fluídos corporais | p2, p5, p6, p8, p15, p16, p21, p22, p23 | 8 (28) | < 0,0001 |
| 6. Transporte de oxigênio | p7, p19, p20, p21 | 4 (14) | < 0,0001 |
| 7. Transporte mediado por vesículas | p2, p5, p6, p10, p14, p15, p16, p19, p20 | 9 (31) | < 0,0001 |
| 8. Ativação plaquetária | p5, p6, p15, p16 | 4 (14) | < 0,0001 |
| 9. Regulação positiva da adesão celular | p15 | 1 (3) | 0,0001 |
| 10. Hemostasia | p2, p5, p6, p15, p16, p21 | 6 (20) | 0,0001 |
| 11. Agregação plaquetária | p2, p15 | 2 (7) | 0,0002 |
| 12. Ativação do plasminogênio | p15 | 1 (3) | 0,0003 |
| 13. Transporte de organismo único | p2, p5, p6, p7, p8, p10, p11, p12, p14, p15, p16, p19, p20, p21 | 14 (48) | 0,0004 |
| 14. Coagulação sanguínea | p2, p5, p6, p15, p16, p21 | 6 (21) | 0,0008 |
| 15. Síntese de translesão propensa a erros | p13 | 1 (3) | 0,0010 |
| 16. Cascata de ativação de proteínas | p15, p23 | 2 (7) | 0,0014 |
| 17. Homeostase da retina | p2, p5, p23, p24 | 4 (14) | 0,0015 |
| 18. Regulação negativa de resposta a trauma | p8, p10, p15 | 3 (10) | 0,0015 |
| 19. Regulação positiva da exocitose | p14, p15 | 2 (7) | 0,0016 |
| 20. Regulação da exocitose | p10, p14, p15 | 3 (10) | 0,0017 |
| 21. Regulação negativa do processo endotelial de apoptose celular | p15 | 1 (3) | 0,0022 |
| 22. Coagulação sanguínea, formação de coágulo de fibrina | p15 | 1 (3) | 0,0024 |
| 23. Regulação negativa da via de sinalização da apoptose extrínseca por meio da morte de receptores de domínio | p15 | 1 (3) | 0,0024 |
| 24. Transporte | p2, p4, p5, p6, p7, p10, p11, p12, p15, p16, p19, p20, p21 | 13 (45) | 0,0029 |
| 25. Processo metabólico do ácido monocarboxílico | p5, p16, p29 | 3 (10) | 0,0030 |
| 26. Regulação da disseminação de células dependentes da adesão | p15 | 1 (3) | 0,0033 |
| 27. Cicatrização de feridas | p2, p5, p6, p15, p16, p21 | 6 (20) | 0,0035 |
| 28. Transporte de bicarbonato | p11, p19, p20 | 3 (10) | 0,0044 |
| 29. Regulação positiva da vasoconstrição | p15 | 1 (3) | 0,0044 |
| 30. Resposta ao íon cálcio | p2 | 1 (3) | 0,0075 |
| 31. Regulação do transporte por vesículas | p8, p10, p14, p15 | 4 (14) | 0,0083 |
| 32. Secreção | p5, p6, p8, p15, p16 | 5 (17) | 0,0092 |
| 33. Regulação negativa a estímulos externos | p8, p10, p15 | 3 (10) | 0,0098 |
| 34. Resposta ao estresse | p2, p5, p6, p13, p16, p19, p20, p21, p23 | 9 (31) | 0,0100 |
Mapa térmico e dendrogramas entre proteínas identificadas (linhas) e funções biológicas (colunas). Destaques em verde: agrupamento de proteínas com padrão de ocorrência semelhante de acordo com as funções que exercem; e em vermelho: as funções com padrão de expressão semelhante, de acordo com as proteínas indicadas.
Exofilina‐5 (EXPH5) é ligada ao transporte de vesículas intracelulares. Foi super‐regulada (M/S = 8,94) no melasma, o que pode decorrer da intensa transferência epidérmica de melanossomas.1 Treze das proteínas diferencialmente identificadas no melasma foram ligadas a fenômenos de transporte intracelular, que compreendem uma série de processos que vão da endocitose à autofagia e diversas formas de exocitose. Como autofagia e senescência são fenômenos relacionados à melanogênese, a caracterização das vesículas de transporte no epitélio com melasma pode se revelar importante na fisiopatologia do melasma.5,6
Citoqueratinas (como KRT1) são constituintes estruturais dos queratinócitos induzidas em resposta ao estresse oxidativo. Foram identificadas em maior razão no melasma (M/S > 4,10). A hemoglobina‐δ (mas não as demais subunidades) apresentou elevada razão (M/S = 33,12) no melasma, e, além do transporte de oxigênio, sua expressão não eritrocítica ocorre em situações de estresse celular.7 Da mesma maneira, o aumento da regulação de alfa 1 antitripsina (SERPINA1) e actina gama‐1 (ACTG1) também é verificado em condições de estresse tecidual.8,9 As maiores expressões de HBD, ACTG1, SERPINA1 e KRT1 no melasma podem decorrer do estresse oxidativo sustentado pela atividade das triptases mastocitárias e do fenótipo secretório de fibroblastos da derme superior.3,6
Anidrase carbônica (CA1) acidifica o meio extracelular da derme, favorecendo o processo de reparo, sendo sub‐regulada (M/S < 0,33) no melasma.10 Senescência dos fibroblastos dérmicos, associada à atividade de MMP1 e MMP9, promovem um microambiente proinflamatório com degradação da matriz extracelular e da zona de membrana basal, cujo déficit de reparo pode ser fator de manutenção da melanogênese.1,6
Androglobina (ADGB) tem função regulatória da cisteína‐endopeptidase, e é identificada em razão menor (M/S < 0,33) no melasma. As endopeptidases participam da degradação dos melanossomas na epiderme, notavelmente reduzidas no melasma.
As proteínas de ancoragem alfa‐quinase (ANCHOR9, ANCHOR13) e a subunidade z‐catalítica da DNA polimerase (REV3L) apresentaram um desbalanço na pele com melasma. Elas são importantes na regulação da proteína quinase‐A e na via p38‐MAP‐quinase, envolvidas na ativação da proteína CREB, que levam à expressão de MTIF, promotora da melanogênese.3
Aldolase‐A (ALDOA) tem função glicolítica e está associada à atividade dos mastócitos, que, na derme superficial promovem alterações na membrana basal, elastose solar e dilatação endotelial, reforçando a ideia de que estímulos originários na derme desempenhem um papel central na melanogênese do melasma.2,3
O fibrinogênio‐γ (FFG) é uma proteína que compõe a matriz extracelular e interage em várias funções biológicas, incluindo fibrinólise, ativação do fibrinogênio e ativação da via ERK, promotora da melanogênese.
As principais limitações do estudo se referem às proteínas transmembranas, séricas e conjugadas com lipídeos que não são identificadas pelo método. Contudo, aponta de maneira consistente uma série de proteínas cujo papel fisiopatológico e potencial manipulação terapêutica devem ser explorados em em ensaios específicos.
Concluindo, identificamos 29 proteínas diferencialmente reguladas no melasma, envolvidas com metabolismo energético, fenômenos de transporte celular, regulação de vias da melanogênese, hemostasia/coagulação, reparo/cicatrização e resposta ao estresse oxidativo. Isso subsidia a pesquisa de estratégias terapêuticas voltadas às proteínas identificadas e suas funções, e evidencia que o melasma não depende exclusivamente da hiperfunção dos melanócitos, mas de alterações funcionais envolvendo a unidade epidermo‐melânica, zona da membrana basal e derme superior.
Suporte financeiroFUNADERSP (048/2016).
Contribuição dos autoresLuiza Vasconcelos Schaefer: Concepção e planejamento do estudo; Elaboração e redação do manuscrito; Obtenção, análise e interpretação dos dados; Participação intelectual em conduta propedêutica e/ou terapêutica de casos estudados; Revisão crítica da literatura.
Leticia Gomes de Pontes: Obtenção, análise e interpretação dos dados.
Nayara Rodrigues Vieira Cavassan: Obtenção, análise e interpretação dos dados.
Lucilene Delazari dos Santos: Revisão crítica da literatura; Revisão crítica do manuscrito; Obtenção, análise e interpretação dos dados.
Hélio Amante Miot: Revisão crítica da literatura; Revisão crítica do manuscrito; Análise estatística; Aprovação da versão final do manuscrito; Concepção e planejamento do estudo.
Conflito de interessesNenhum.
Como citar este artigo: Schaefer LV, Pontes LG, Cavassan NRV, Santos LD, Miot HÁ. Proteomic study of facial melasma. An Bras Dermatol. 2022;97:808–14.
Trabalho realizado no Departamento de Dermatologia e Radioterapia, FMB‐UNESP, Botucatu, SP, Brasil e no Centro de Estudos de Venenos e Animais Peçonhentos (CEVAP), UNESP, Botucatu, SP, Brasil.