Sífilis é infecção sexualmente transmissível de evolução polimórfica causada por Treponema Pallidum subespécie pallidum (TP). Os sistemas mucocutâneo, neurológico e cardiovascular são os mais afetados, e a transmissão de mãe para filho pode ocorrer em qualquer fase da gestação.1 A Organização Mundial da Saúde (OMS) estimou 6,3 milhões de casos anuais no mundo. No Brasil, a sífilis aumentou de 33,9 casos em 2015 para 74,2 casos por 100.000 habitantes em 2019.2
O TP não é cultivável com métodos de cultura padrão. Na prática clínica, o diagnóstico é presuntivo usando testes sorológicos. Diferentes técnicas de reação em cadeia da polimerase (PCR, do inglês polymerase chain reaction) têm sido usadas para o diagnóstico,3 e o sequenciamento de DNA é cada vez mais utilizado para estudar diversidade genética, dinâmica de transmissão, virulência e padrões de resistência.4 A genotipagem por tipagem de sequência multilocus (MLST, do inglês multilocus sequence typing) nos loci cromossômicos TP0136, TP0548 e TP0705 possibilita melhor discriminação de cepas de TP e tornou possível a criação de um banco de dados de análise epidemiológica (https://pubmlst.org/ organisms/treponema‐pallidum). Diferentes loci, alelos distintos e sua combinação definem o perfil alélico e o tipo de sequência (TS). A análise do rRNA do gene 23S pode complementar a identificação de mutações (A2058G ou A2059G) que estão relacionadas à resistência a macrolídeos.5
O objetivo do presente estudo foi detectar e genotipar TP por meio de MLST em úlceras genitais presuntivas de sífilis (GUPS, do inglês genital ulcers presumptive of syphilis).
Foram analisadas amostras de exsudato de GUPS obtidas de pacientes do gênero masculino com 18 anos ou mais atendidos em clínica pública de Infecções Sexualmente Transmissíveis (ISTs) em Porto Alegre, Brasil, de julho de 2019 a março de 2019. A coleta da amostra foi realizada com cotonete seco. A extração de DNA foi realizada utilizando PureLink® Genomic Kit (Invitrogen®, Thermo Fisher Scientific). A detecção foi feita por meio de amplificação por PCR de 260 pb do gene tpp47 com DNA usando os primers KO3 (5’‐GAAGTTTGTCCCAGTTGCTGCTTT‐3’) e KO4 (5’‐CAGAGCCATCAGCCCTTTTCA‐3’). A amplificação foi realizada com Taq platinum DNA polymerase (Invitrogem, Fisher Scientific, EUA) e Thermocycler PTC96 (Bioer Technology, China). A análise dos produtos da PCR foi realizada por eletroforese em gel de agarose (0,05% de brometo de etídio). A análise de MLST foi realizada por meio de sequenciamento de loci cromossômicos (TP0136, TP0548 e TP0705), como descrito anteriormente por Grillová.4 A avaliação das mutações A2058G e A2059G no gene 23S rRNA foi realizada pela técnica de nested PCR. A análise de sequenciamento foi realizada com o software Bioedit (Tom Hall, EUA), e a análise de genotipagem foi realizada pelo esquema incluindo os loci TP0136, TP0548 e TP0705,4 usando a plataforma de banco de dados público para tipagem molecular e diversidade do genoma microbiano (PubMLST, disponível em https://pubmlst.org/organisms/treponema‐pallidum). A análise de sequenciamento relacionada à resistência foi realizada por meio da anotação da sequência correspondente às posições 2058 e 2059 no rRNA do gene da Escherichia coli (número de acesso V0033, GenBank). Controles positivos e negativos foram usados em cada rodada. A aprovação ética foi obtida da Faculdade de Saúde Pública, do estado do Rio Grande do Sul (n° 3.232.889).
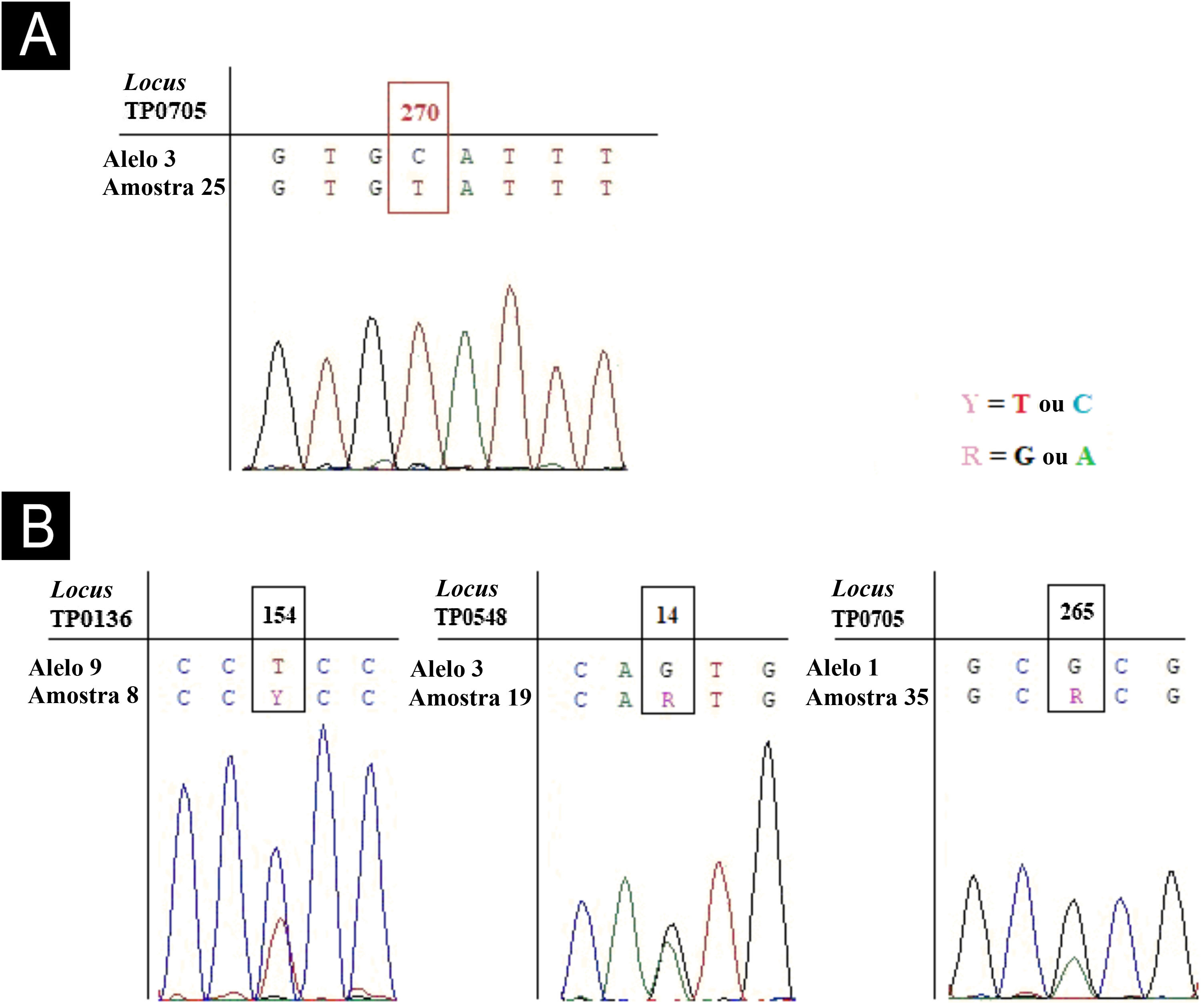
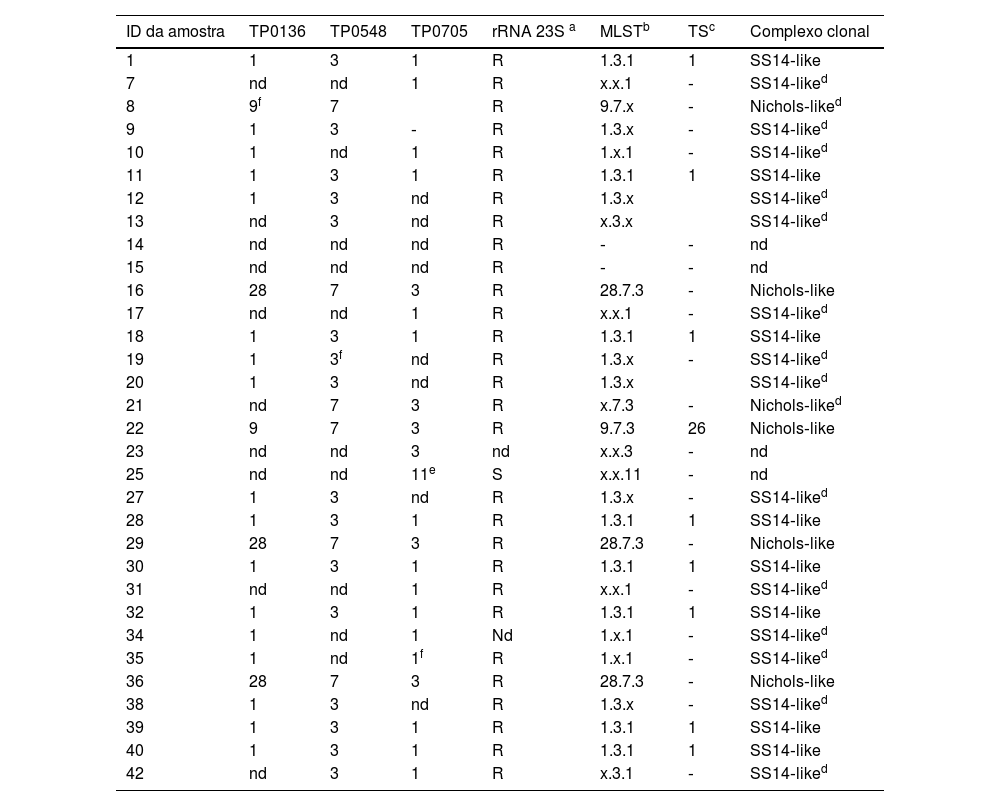
Foram recrutados 43 participantes. As idades variaram de 19 a 66 anos, e todos os participantes eram moradores da área metropolitana de Porto Alegre. Foi detectada sequência específica de TP em 32 (74%) das 43 amostras de DNA analisadas. Em 30 (94%) das 32, pelo menos um locus do esquema MLST foi sequenciado com sucesso. A mesma proporção do rRNA do gene 23S foi sequenciada com sucesso. Foi obtido sequenciamento de qualidade de TP0705 em 22 amostras, bem como para TP0136 e TP0548. A combinação de locus sequenciados com sucesso entre as amostras variou. Foram identificadas três variantes alélicas para TP0136, duas para TP0548 e três para TP0705. Foi identificado um novo alelo para TP0705 na única amostra caracterizada como genotipicamente suscetível a macrolídeos (designada alelo 11), que diferiu do alelo 3 na posição 270 do loci (C270T). Três outras amostras apresentaram picos heterozigotos (dois picos) nas posições 154 para TP0136, 14 para TP0548 e 265 para TP0705 (fig. 1). Os resultados da tipagem de TP, os alelos identificados, o perfil de resistência genotípica e o complexo clonal são apresentados na tabela 1.
Novo alelo para TP0705 e três picos heterozigotos para os loci TP0136, TP0548 e TP0705. (A) Cromatograma da amostra 25 mostrou a nova variante alélica chamada aqui de 11, caracterizada por um SNV na posição 270 (C→T) utilizando o alelo 3 (sequência mais similar) como referência. (B) Cromatogramas de três amostras mostraram pico heterozigoto para o locus TP0136 na posição 154 (C e T), TP0548 na posição 14 (G e A) e TP0705 na posição 265 (G e A). Os alelos utilizados como referência são a sequência mais similar observada no banco de dados PubMLST.
Genotipagem de T. pallidum por tipagem de sequência multilocus (MLST) nas amostras estudadas
| ID da amostra | TP0136 | TP0548 | TP0705 | rRNA 23S a | MLSTb | TSc | Complexo clonal |
|---|---|---|---|---|---|---|---|
| 1 | 1 | 3 | 1 | R | 1.3.1 | 1 | SS14‐like |
| 7 | nd | nd | 1 | R | x.x.1 | ‐ | SS14‐liked |
| 8 | 9f | 7 | R | 9.7.x | ‐ | Nichols‐liked | |
| 9 | 1 | 3 | ‐ | R | 1.3.x | ‐ | SS14‐liked |
| 10 | 1 | nd | 1 | R | 1.x.1 | ‐ | SS14‐liked |
| 11 | 1 | 3 | 1 | R | 1.3.1 | 1 | SS14‐like |
| 12 | 1 | 3 | nd | R | 1.3.x | SS14‐liked | |
| 13 | nd | 3 | nd | R | x.3.x | SS14‐liked | |
| 14 | nd | nd | nd | R | ‐ | ‐ | nd |
| 15 | nd | nd | nd | R | ‐ | ‐ | nd |
| 16 | 28 | 7 | 3 | R | 28.7.3 | ‐ | Nichols‐like |
| 17 | nd | nd | 1 | R | x.x.1 | ‐ | SS14‐liked |
| 18 | 1 | 3 | 1 | R | 1.3.1 | 1 | SS14‐like |
| 19 | 1 | 3f | nd | R | 1.3.x | ‐ | SS14‐liked |
| 20 | 1 | 3 | nd | R | 1.3.x | SS14‐liked | |
| 21 | nd | 7 | 3 | R | x.7.3 | ‐ | Nichols‐liked |
| 22 | 9 | 7 | 3 | R | 9.7.3 | 26 | Nichols‐like |
| 23 | nd | nd | 3 | nd | x.x.3 | ‐ | nd |
| 25 | nd | nd | 11e | S | x.x.11 | ‐ | nd |
| 27 | 1 | 3 | nd | R | 1.3.x | ‐ | SS14‐liked |
| 28 | 1 | 3 | 1 | R | 1.3.1 | 1 | SS14‐like |
| 29 | 28 | 7 | 3 | R | 28.7.3 | ‐ | Nichols‐like |
| 30 | 1 | 3 | 1 | R | 1.3.1 | 1 | SS14‐like |
| 31 | nd | nd | 1 | R | x.x.1 | ‐ | SS14‐liked |
| 32 | 1 | 3 | 1 | R | 1.3.1 | 1 | SS14‐like |
| 34 | 1 | nd | 1 | Nd | 1.x.1 | ‐ | SS14‐liked |
| 35 | 1 | nd | 1f | R | 1.x.1 | ‐ | SS14‐liked |
| 36 | 28 | 7 | 3 | R | 28.7.3 | ‐ | Nichols‐like |
| 38 | 1 | 3 | nd | R | 1.3.x | ‐ | SS14‐liked |
| 39 | 1 | 3 | 1 | R | 1.3.1 | 1 | SS14‐like |
| 40 | 1 | 3 | 1 | R | 1.3.1 | 1 | SS14‐like |
| 42 | nd | 3 | 1 | R | x.3.1 | ‐ | SS14‐liked |
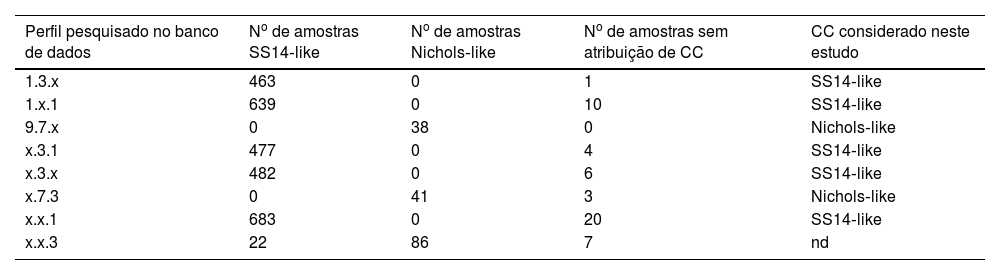
Foram obtidos tipos de sequência (TS), que são atribuídos a haplótipos completamente caracterizados (três loci MLST) para 11 amostras, e o mais frequente (8/11;72,7%) foi o perfil 1.3.1 (TS 1). O perfil 28.7.3 ainda não recebeu TS na plataforma PubMLST. Foram classificadas amostras que tiveram pelo menos um locus sequenciado com sucesso em um complexo clonal por aproximação. Essa classificação foi baseada no número de isolados sequenciados contendo o(s) alelo(s) armazenado(s) no banco de dados PubMLST (tabela 2). Foram identificados dois complexos clonais de cepas: seis (20%) de 30 isolados foram classificados como Nichols‐like e 22 (73%) como SS14‐like. Não foi viável atribuir um complexo clonal às duas amostras que tiveram apenas o locus TP0705 sequenciado com sucesso. Uma amostra foi caracterizada como alelo 3, enquanto a outra exibiu um alelo recém‐identificado designado como alelo TP0705‐22 (fig. 1).5 Quanto às mutações relacionadas à resistência, das 30 amostras que foram adequadamente caracterizadas, apenas uma não tinha a mutação A2058G no rRNA do gene 23S e, portanto, foi classificada como suscetível.
Complexo clonal de perfis genotípicos incompletos com base no número de amostras presentes no banco de dados PubMLST
| Perfil pesquisado no banco de dados | No de amostras SS14‐like | No de amostras Nichols‐like | No de amostras sem atribuição de CC | CC considerado neste estudo |
|---|---|---|---|---|
| 1.3.x | 463 | 0 | 1 | SS14‐like |
| 1.x.1 | 639 | 0 | 10 | SS14‐like |
| 9.7.x | 0 | 38 | 0 | Nichols‐like |
| x.3.1 | 477 | 0 | 4 | SS14‐like |
| x.3.x | 482 | 0 | 6 | SS14‐like |
| x.7.3 | 0 | 41 | 3 | Nichols‐like |
| x.x.1 | 683 | 0 | 20 | SS14‐like |
| x.x.3 | 22 | 86 | 7 | nd |
CC, complexo clonal.
O DNA do TP foi detectado em aproximadamente 2/3 das amostras. Elas pertenciam aos complexos clonais SS14‐like ou Nichols‐like. Um novo alelo foi identificado nas duas amostras não classificadas em um complexo clonal (x.x.3 e x.x.11).5 O haplótipo contendo esse alelo tinha apenas o locus TP0705 caracterizado. Esse locus pode compartilhar alelos idênticos entre cepas dos clados SS14 e Nichols, o que dificulta a atribuição de um complexo clonal.
Com exceção de uma, as amostras positivas para DNA, apresentaram a mutação A2058G no rRNA do gene 23S, que fornece resistência à classe de antibióticos macrolídeos.6 Os achados do presente estudo corroboram os de Grillová et al.7 e Giacani,8 que demonstraram alta e crescente proporção dessa mutação. Foram identificados os mesmos complexos clonais em 20% das amostras deste estudo (complexo clonal SS14 ou Nichols‐like). Foram encontrados três perfis MLST totalmente caracterizados de TP (1.3.1; 9.7.3; 28.7.3).
As amostras negativas para DNA de TP podem ter como explicação: 1) alguns pacientes não tinham sífilis; 2) a quantidade de microrganismos nas lesões diminuiu ao longo do tempo; e 3) o uso anterior de antibióticos tópicos e sistêmicos. O pequeno tamanho amostral impediu as associações do estudo de diferentes genótipos com dados demográficos, práticas sexuais ou origem geográfica dos participantes, uma vez que a epidemia de SARS‐CoV2 dificultou o recrutamento dos mesmos. O desenvolvimento de estudos com tamanhos amostrais maiores poderá fornecer informações adicionais que são cruciais para o controle da sífilis.
Suporte financeiroA Coordenação de Aperfeiçoamento de Pessoal de Nível Superior do Ministério da Educação (CAPES) concedeu bolsa de estudos de doutorado para V.M.T. Grassi por meio do CÓDIGO 001 de financiamento. O Ministério da Saúde do Brasil financiou o estudo por meio de uma Carta Acordo com a Organização Pan‐Americana da Saúde (OPAS), n° SCON00400/2019.
Contribuição dos autoresLeonardo Souza Esteves: Responsável pela concepção e planejamento do estudo, realizou análise de sequenciamento bioinformática e genotipagem, forneceu comentários e edições e aprovou o rascunho final antes da submissão.
Vera Mileide Trivellato Grassi: Responsável pela concepção e planejamento do estudo, forneceu comentários e edições e aprovou o rascunho final antes da submissão.
Liliane Trivellato Grassi: Realizou a organização das referências, forneceu comentários e edições e aprovou o rascunho final antes da submissão.
Maria Rita Castilhos Nicola: Participou da coleta e gerenciamento dos dados, forneceu comentários e edições e aprovou o rascunho final antes da submissão.
Marcia Susana Nunes Silva: Responsável pela concepção e planejamento do estudo, forneceu comentários e edições e aprovou o rascunho final antes da submissão.
Maria Lucia Rosa Rossetti: Responsável pela concepção e planejamento do estudo, forneceu comentários e edições e aprovou o rascunho final antes da submissão.
Mauro Cunha Ramos: Responsável pela concepção e planejamento do estudo, forneceu comentários e edições e aprovou o rascunho final antes da submissão.
Conflito de interessesNenhum.
Como citar este artigo: Esteves LS, Grassi VMT, Grassi LT, Nicola MRC, Silva MSN, Rossetti MLR, et al. Multilocus sequence typing of Treponema pallidum in male patients with genital ulcers in a public sexually transmitted infections clinic: a new allele and almost complete macrolide resistance. An Bras Dermatol. 2025;100:569–73.
Trabalho realizado no Ambulatório de Dermatologia Sanitária, Porto Alegre, RS, Brasil.